Diffusion biologique aux petits angles - Biological small-angle scattering
La diffusion biologique aux petits angles est une méthode de diffusion aux petits angles pour l'analyse de la structure des matériaux biologiques. La diffusion aux petits angles est utilisée pour étudier la structure d'une variété d'objets tels que des solutions de macromolécules biologiques, de nanocomposites, d'alliages et de polymères synthétiques. La diffusion des rayons X aux petits angles ( SAXS ) et la diffusion des neutrons aux petits angles ( SANS ) sont les deux techniques complémentaires connues conjointement sous le nom de diffusion aux petits angles (SAS). SAS est une méthode analogue à la diffraction des rayons X et des neutrons , à la diffusion des rayons X aux grands angles , ainsi qu'à la diffusion statique de la lumière . Contrairement aux autres méthodes de diffusion des rayons X et des neutrons, SAS fournit des informations sur la taille et la forme des particules cristallines et non cristallines. Lorsqu'il est utilisé pour étudier des matériaux biologiques, qui sont très souvent en solution aqueuse, le motif de diffusion est l'orientation moyenne.
Les motifs SAS sont collectés à de petits angles de quelques degrés. SAS est capable de fournir des informations structurelles dans une plage de résolution comprise entre 1 et 25 nm et des distances de répétition dans des systèmes partiellement ordonnés d'une taille allant jusqu'à 150 nm. La diffusion aux ultra petits angles (USAS) peut résoudre des dimensions encore plus grandes. La diffusion rasante aux petits angles (GISAS) est une technique puissante pour l'étude des couches de molécules biologiques sur les surfaces.
Dans les applications biologiques, SAS est utilisé pour déterminer la structure d'une particule en termes de taille et de forme moyennes des particules. On peut également obtenir des informations sur le rapport surface / volume . Typiquement, les macromolécules biologiques sont dispersées dans un liquide. La méthode est précise, généralement non destructive et ne nécessite généralement qu'un minimum de préparation d'échantillons. Cependant, les molécules biologiques sont toujours sensibles aux dommages causés par les radiations .
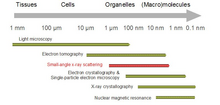
Par rapport à d'autres méthodes de détermination de structure, telles que la RMN en solution ou la cristallographie aux rayons X , SAS permet de surmonter certaines contraintes. Par exemple, la RMN en solution est limitée à la taille des protéines, tandis que le SAS peut être utilisé pour les petites molécules ainsi que pour les grands assemblages multimoléculaires. La RMN à l'état solide reste un outil indispensable pour déterminer les informations au niveau atomique des macromolécules supérieures à 40 kDa ou des échantillons non cristallins tels que les fibrilles amyloïdes. La détermination de la structure par cristallographie aux rayons X peut prendre plusieurs semaines, voire plusieurs années, alors que les mesures SAS prennent des jours. SAS peut également être couplé à d'autres techniques analytiques comme la chromatographie d'exclusion stérique pour étudier des échantillons hétérogènes. Cependant, avec SAS, il n'est pas possible de mesurer les positions des atomes au sein de la molécule.
Méthode
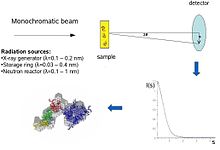
Conceptuellement, les expériences de diffusion aux petits angles sont simples : l'échantillon est exposé aux rayons X ou aux neutrons et le rayonnement diffusé est enregistré par un détecteur. Comme les mesures SAS sont effectuées très près du faisceau primaire ("petits angles"), la technique nécessite un faisceau de rayons X ou de neutrons hautement collimaté ou focalisé . La diffusion biologique des rayons X aux petits angles est souvent réalisée sur des sources de rayonnement synchrotron , car les molécules biologiques diffusent normalement faiblement et les solutions mesurées sont diluées . La méthode biologique SAXS bénéficie de la haute intensité des faisceaux de photons X fournis par les anneaux de stockage synchrotron . La courbe de diffusion des rayons X ou des neutrons ( intensité en fonction de l' angle de diffusion ) est utilisée pour créer un modèle basse résolution d'une protéine, montré ici sur l'image de droite. On peut en outre utiliser les données de diffusion des rayons X ou des neutrons et insérer des domaines séparés ( structures aux rayons X ou RMN ) dans "l'enveloppe SAXS".
Dans une expérience de diffusion, une solution de macromolécules est exposée à des rayons X (avec une longueur d' onde λ typiquement de l' ordre de 0,15 nm) ou thermiques des neutrons ( λ ≈0.5 nm). L'intensité diffusée I(s) est enregistrée en fonction du transfert de quantité de mouvement s ( s=4πsinθ/λ , où 2θ est l'angle entre le rayonnement incident et diffusé). De l'intensité de la solution, la diffusion du seul solvant est soustraite. Les positions et orientations aléatoires des particules donnent une distribution d'intensité isotrope qui, pour les particules monodisperses sans interaction, est proportionnelle à la diffusion d'une seule particule moyennée sur toutes les orientations. La diffusion nette des particules est proportionnelle à la différence au carré de la densité de longueur de diffusion ( densité électronique pour les rayons X et densité nucléaire/spin pour les neutrons) entre la particule et le solvant – ce qu'on appelle le contraste. Le contraste peut être modifié dans la diffusion des neutrons en utilisant des mélanges H 2 O/ D 2 O ou une deutération sélective pour fournir des informations supplémentaires. Le contenu informatif des données SAS est illustré ici dans la figure de droite, qui montre les schémas de diffusion des rayons X à partir de protéines avec différents plis et masses moléculaires . Aux faibles angles (résolution de 2 à 3 nm), les courbes sont des fonctions de s à déclin rapide essentiellement déterminées par la forme des particules, qui diffèrent clairement. A moyenne résolution (2 à 0,5 nm) les différences sont déjà moins prononcées et au-dessus de 0,5 nm de résolution toutes les courbes sont très similaires. SAS contient donc des informations sur les caractéristiques structurelles brutes - forme, structure quaternaire et tertiaire - mais n'est pas adapté à l'analyse de la structure atomique.
Histoire
Les premières applications remontent à la fin des années 1930 lorsque les grands principes du SAXS ont été développés dans les travaux fondamentaux de Guinier suite à ses études sur les alliages métalliques. Dans la première monographie sur SAXS par Guinier et Fournet, il a déjà été démontré que la méthode fournit non seulement des informations sur les tailles et les formes des particules, mais aussi sur la structure interne des systèmes désordonnés et partiellement ordonnés.
Dans les années 1960, la méthode est devenue de plus en plus importante dans l'étude des macromolécules biologiques en solution car elle a permis d'obtenir des informations structurelles à basse résolution sur la forme globale et la structure interne en l'absence de cristaux. Une percée dans les expériences SAXS et SANS a eu lieu dans les années 1970, grâce à la disponibilité de sources de rayonnement synchrotron et de neutrons, ces dernières ouvrant la voie à la variation de contraste par échange de solvants H 2 O contre D 2 O et méthodes de deutération spécifiques. On s'est rendu compte que les études de diffusion sur solution fournissent, pour un investissement minimal de temps et d'efforts, des informations utiles sur la structure des systèmes biochimiques non cristallins. De plus, SAXS/SANS a également rendu possible des études en temps réel des interactions intermoléculaires, y compris l'assemblage et les changements de conformation à grande échelle dans les assemblages macromoléculaires .
Le principal défi de SAS en tant que méthode structurelle est d'extraire des informations sur la structure tridimensionnelle de l'objet à partir des données expérimentales unidimensionnelles. Dans le passé, seuls les paramètres globaux des particules (par exemple le volume, le rayon de giration) des macromolécules étaient directement déterminés à partir des données expérimentales, alors que l'analyse en termes de modèles tridimensionnels était limitée à des corps géométriques simples (par exemple des ellipsoïdes, des cylindres, etc. .) ou a été effectué sur la base d'essais et d'erreurs ad hoc. La microscopie électronique a souvent été utilisée comme une contrainte dans la construction de modèles de consensus. Dans les années 1980, les progrès d'autres méthodes structurelles ont entraîné un déclin de l'intérêt des biochimistes pour les études SAS, qui tiraient des conclusions structurelles à partir de quelques paramètres généraux ou étaient basées sur des modèles d'essais et d'erreurs.
Les années 1990 ont apporté une percée dans les méthodes d'analyse de données SAXS/SANS, qui ont ouvert la voie à une modélisation ab initio fiable des complexes macromoléculaires, y compris la détermination détaillée de la forme et de la structure du domaine et l'application de techniques de raffinement des corps rigides. Ce progrès s'est accompagné de nouvelles avancées dans l'instrumentation, permettant d'atteindre des résolutions temporelles inférieures à la ms sur des sources SR de troisième génération dans les études de repliement des protéines et des acides nucléiques.
En 2005, un projet de quatre ans a été lancé. S mall- A ngle X - Ray diffraction I nitiative pour E u R ope (SAXIER) dans le but de combiner des méthodes SAXS avec d' autres techniques d' analyse et de créer un logiciel automatisé pour analyser rapidement de grandes quantités de données. Le projet a créé une infrastructure SAXS européenne unifiée, en utilisant les méthodes les plus avancées disponibles.
L'analyse des données
Dans une expérience SAS de bonne qualité, plusieurs solutions avec des concentrations variables de la macromolécule à l'étude sont mesurées. En extrapolant les courbes de diffusion mesurées à différentes concentrations à une concentration nulle, on peut obtenir une courbe de diffusion qui représente une dilution infinie. Ensuite, les effets de concentration ne devraient pas affecter la courbe de diffusion. L'analyse des données de la courbe de diffusion extrapolée commence par l'inspection du début de la courbe de diffusion dans la région autour de s = 0 . Si la région suit l' approximation de Guinier (également appelée loi de Guinier ), l'échantillon n'est pas agrégé . Ensuite, la forme de la particule en question peut être déterminée par différentes méthodes, dont certaines sont décrites dans la référence suivante.
Transformée de Fourier indirecte
La première étape consiste généralement à calculer une transformée de Fourier de la courbe de diffusion. La courbe transformée peut être interprétée comme une fonction de distribution de distance à l' intérieur d'une particule. Cette transformation donne également un avantage de régularisation des données d'entrée.
Modèles basse résolution
Un problème dans l'analyse des données SAS est d'obtenir une structure tridimensionnelle à partir d'un modèle de diffusion unidimensionnel. Les données SAS n'impliquent pas une solution unique. De nombreuses protéines différentes, par exemple, peuvent avoir la même courbe de diffusion. La reconstruction de la structure 3D peut entraîner un grand nombre de modèles différents. Pour éviter ce problème, un certain nombre de simplifications doivent être envisagées.
Une approche supplémentaire consiste à combiner des données et un modèle de diffusion des rayons X et des neutrons aux petits angles avec le programme MONSA.
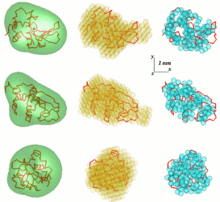
Des programmes informatiques d'analyse SAS disponibles gratuitement ont été intensivement développés à l' EMBL . Dans la première approche générale ab initio , une fonction d'enveloppe angulaire de la particule r=F(ω) , où ( r,ω ) sont des coordonnées sphériques, est décrite par une série d' harmoniques sphériques . La forme basse résolution est ainsi définie par quelques paramètres – les coefficients de cette série – qui s'ajustent aux données de diffusion. L'approche a été développée et mise en œuvre dans le programme informatique SASHA ( Détermination de la forme de diffusion aux petits angles ). Il a été démontré que dans certaines circonstances, une enveloppe unique peut être extraite des données de diffusion. Cette méthode n'est applicable qu'aux particules globulaires de formes relativement simples et sans cavités internes importantes. Pour surmonter ces limitations, une autre approche a été développée, qui utilise différents types de recherches Monte-Carlo. DALAI_GA est un programme élégant, qui prend une sphère de diamètre égal à la taille de particule maximale Dmax, qui est déterminée à partir des données de diffusion, et la remplit de billes. Chaque bille appartient soit à la particule (indice=1) soit au solvant (indice=0). La forme est ainsi décrite par la chaîne binaire de longueur M. À partir d'une chaîne aléatoire, un algorithme génétique recherche un modèle qui correspond aux données. Des contraintes de compacité et de connectivité sont imposées dans la recherche, implémentée dans le programme DAMMIN . Si la symétrie des particules est connue, SASHA et DAMMIN peuvent l'utiliser comme contraintes utiles. La procédure ' donner et prendre' SAXS3D et le programme SASMODEL , basé sur des ellipsoïdes interconnectés sont des approches de Monte Carlo ab initio sans limitation dans l'espace de recherche.
Une approche qui utilise un ensemble de résidus factices (DR) et un recuit simulé pour construire un modèle DR localement "compatible avec la chaîne" à l'intérieur d'une sphère de diamètre Dmax permet d'extraire plus de détails des données SAXS. Cette méthode est implémentée dans le programme GASBOR .
Les modèles de diffusion de solution de protéines multidomaines et de complexes macromoléculaires peuvent également être ajustés à l'aide de modèles construits à partir de structures à haute résolution ( RMN ou rayons X ) de domaines ou de sous-unités individuels en supposant que leur structure tertiaire est préservée. Selon la complexité de l'objet, différentes approches sont utilisées pour la recherche globale de la configuration optimale des sous-unités correspondant aux données expérimentales.
Modèle consensuel
Les modèles basés sur Monte-Carlo contiennent des centaines ou des milliers de paramètres, et la prudence est de mise pour éviter toute surinterprétation. Une approche courante consiste à aligner un ensemble de modèles résultant d'exécutions de reconstruction de forme indépendantes pour obtenir un modèle moyen conservant les caractéristiques les plus persistantes et peut-être aussi les plus fiables (par exemple en utilisant le programme SUPCOMB ).
Ajout de boucles manquantes
Les acides aminés de surface désordonnés (" boucles ") sont fréquemment non observés dans les études RMN et cristallographiques , et peuvent être laissés manquants dans les modèles rapportés. Un tel élément désordonné contribue à l'intensité de diffusion et leurs emplacements probables peuvent être trouvés en fixant la partie connue de la structure et en ajoutant les parties manquantes pour s'adapter au motif SAS de la particule entière. L'approche Dummy Residue a été étendue et les algorithmes d'ajout de boucles ou de domaines manquants ont été implémentés dans la suite de programmes CREDO .
Méthodes hybrides
Récemment, quelques méthodes ont proposé qui utilisent les données SAXS comme contraintes. Les auteurs visaient à améliorer les résultats des méthodes de reconnaissance de pli et de prédiction de la structure des protéines de novo . Les données SAXS fournissent la transformée de Fourier de l'histogramme des distances des paires atomiques (fonction de distribution des paires) pour une protéine donnée. Cela peut servir de contrainte structurelle sur les méthodes utilisées pour déterminer le pli conformationnel natif de la protéine. La reconnaissance de filetage ou de pli suppose que la structure 3D est plus conservée que la séquence. Ainsi, des séquences très divergentes peuvent avoir une structure similaire. Les méthodes ab initio, d'autre part, remettent en question l'un des plus gros problèmes de la biologie moléculaire, à savoir, prédire le repliement d'une protéine "à partir de zéro", en n'utilisant aucune séquence ou structure homologue. En utilisant le "filtre SAXS", les auteurs ont pu purifier de manière significative l'ensemble des modèles de protéines de novo. Ceci a en outre été prouvé par des recherches d' homologie de structure . Il a également été montré que la combinaison des scores SAXS avec les scores, utilisés dans les méthodes de threading, améliore considérablement les performances de reconnaissance des plis. Sur un exemple, il a été démontré comment une structure tertiaire approximative de protéines modulaires peut être assemblée à partir de structures de domaines RMN à haute résolution, en utilisant des données SAXS, en confinant les degrés de liberté traductionnels. Un autre exemple montre comment les données SAXS peuvent être combinées avec la RMN, la cristallographie aux rayons X et la microscopie électronique pour reconstruire la structure quaternaire d'une protéine multidomaine.
Systèmes flexibles
Une méthode élégante pour aborder le problème des protéines intrinsèquement désordonnées ou multi-domaines avec des lieurs flexibles a été proposée récemment. Il permet la coexistence de différentes conformations d'une protéine, qui contribuent ensemble au schéma de diffusion expérimental moyen. Initialement, EOM (méthode d'optimisation d'ensemble) génère un pool de modèles couvrant l'espace de configuration des protéines. La courbe de diffusion est ensuite calculée pour chaque modèle. Dans la deuxième étape, le programme sélectionne des sous-ensembles de modèles de protéines. La diffusion expérimentale moyenne est calculée pour chaque sous-ensemble et ajustée aux données expérimentales SAXS. Si le meilleur ajustement n'est pas trouvé, les modèles sont remaniés entre différents sous-ensembles et un nouveau calcul de diffusion moyenne et un ajustement aux données expérimentales sont effectués. Cette méthode a été testée sur deux protéines : le lysozyme dénaturé et la protéine kinase de Bruton . Cela a donné des résultats intéressants et prometteurs.
Couches de molécules biologiques et GISAS
Les revêtements de biomolécules peuvent être étudiés par diffusion de rayons X et de neutrons à incidence rasante. IsGISAXS (grazing incidence small angle X-ray scattering ) est un logiciel dédié à la simulation et à l'analyse de GISAXS à partir de nanostructures. IsGISAXS n'englobe que la diffusion par des particules de taille nanométrique, qui sont enterrées dans une sous-surface matricielle ou supportées sur un substrat ou enterrées dans une couche mince sur un substrat. Le cas des trous est également traité. La géométrie est restreinte à un plan de particules. La section efficace de diffusion est décomposée en termes de fonction d'interférence et de facteur de forme des particules . L'accent est mis sur la géométrie d'incidence rasante qui induit un "effet de réfraction du faisceau". Le facteur de forme des particules est calculé dans l' approximation de Born d'onde déformée (DWBA), en commençant par un état non perturbé avec des interfaces nettes ou avec le profil perpendiculaire réel de l' indice de réfraction . Divers types de formes géométriques simples sont disponibles avec un compte rendu complet des distributions de taille et de forme dans l'approximation de découplage (DA), dans l'approximation monodisperse locale (LMA) et également dans l'approximation de corrélation d'espacement de taille (SSCA). On considère à la fois des systèmes désordonnés de particules définis par leur fonction de corrélation particule-particule et un cristal ou paracristal bidimensionnel.
Voir également
- Anton Paar
- Bruker
- Microscopie électronique
- Diffusion des rayons X par fluctuation (FXS)
- Diffusion des rayons X aux petits angles à incidence rasante ( GISAXS )
- Modélisation d'homologie
- Echo de spin neutronique
- Banque de données sur les protéines
- Dynamique des protéines
- Repliement des protéines
- Enfilage de protéines
- Rigaku
- Rosetta@home
- Cristallographie aux rayons X
Les références
Lectures complémentaires
- Koch MH, Vachette P, Svergun DI (mai 2003). « Diffusion aux petits angles : une vue sur les propriétés, les structures et les changements structurels des macromolécules biologiques en solution ». Revues trimestrielles de biophysique . 36 (2) : 147-227. doi : 10.1017/S0033583503003871 . PMID 14686102 . S2CID 5432509 .
- Petoukhov MV, Svergun DI (août 2005). "Modélisation globale de corps rigides de complexes macromoléculaires contre des données de diffusion aux petits angles" . Journal biophysique . 89 (2) : 1237-1250. Bibcode : 2005BpJ .... 89.1237P . doi : 10.1529/biophysj.105.064154 . PMC 1366608 . PMID 15923225 .
- Bernadó P, Blackledge M (décembre 2010). "Biologie structurale : Protéines en équilibre dynamique" . Nature . 468 (7327) : 1046–8. Bibcode : 2010Natur.468.1046B . doi : 10.1038/4681046a . PMID 21179158 .
Liens externes
- Synchrotron australien de ligne de faisceau SAXS/WAXS , Melbourne, Australie
- SIBYLS – ligne de lumière à Advanced Light Source , Berkeley, USA
- SAXS – ligne de lumière au Laboratoire de lumière synchrotron ELETTRA , Trieste, Italie
- X33 – ligne de lumière à DESY , Hambourg, Allemagne
- D11A - ligne de lumière au laboratoire brésilien de lumière synchrotron , Campinas, Brésil
- X21 et X9 – lignes de lumière à la source de lumière synchrotron nationale du Brookhaven National Laboratory , Upton, États-Unis
- F2 et G1 – lignes de lumière au Cornell Laboratory for Accelerator-based Sciences and Education , Ithaca, USA
- Bio-SANS - à ligne de lumière réacteur à haut flux Isotope à Oak Ridge National Laboratory , Oak Ridge, TN, États - Unis