Histone - Histone
En biologie , les histones sont des protéines hautement basiques abondantes dans les résidus de lysine et d' arginine que l'on trouve dans les noyaux des cellules eucaryotes . Ils agissent comme des bobines autour desquelles l' ADN s'enroule pour créer des unités structurelles appelées nucléosomes . Les nucléosomes sont à leur tour enveloppés dans des fibres de 30 nanomètres qui forment de la chromatine étroitement tassée . Les histones empêchent l'ADN de s'emmêler et le protègent des dommages à l'ADN . De plus, les histones jouent un rôle important dans la régulation des gènes et la réplication de l'ADN . Sans histones, l'ADN déroulé dans les chromosomes serait très long. Par exemple, chaque cellule humaine a environ 1,8 mètre d'ADN si elle est complètement étirée, cependant lorsqu'elle est enroulée autour d'histones, cette longueur est réduite à environ 90 micromètres (0,09 mm) de fibres de chromatine de 30 nm de diamètre.
Il existe cinq familles d'histones qui sont désignées H1/H5 (histones de liaison), H2, H3 et H4 (histones de noyau). Le noyau du nucléosome est formé de deux dimères H2A-H2B et d'un tétramère H3-H4 . L'enveloppement serré de l'ADN autour des histones est dans une large mesure le résultat de l' attraction électrostatique entre les histones chargées positivement et le squelette phosphate de l'ADN chargé négativement.
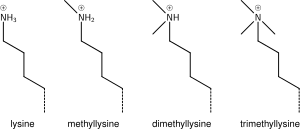
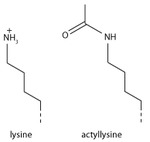
Les histones peuvent être modifiées chimiquement par l'action d'enzymes pour réguler la transcription des gènes. La modification la plus courante est la méthylation des résidus arginine ou lysine ou l' acétylation de la lysine. La méthylation peut affecter la façon dont d'autres protéines telles que les facteurs de transcription interagissent avec les nucléosomes. L'acétylation de la lysine élimine une charge positive sur la lysine, affaiblissant ainsi l'attraction électrostatique entre l'histone et l'ADN, entraînant un déroulement partiel de l'ADN le rendant plus accessible à l'expression génique.
Classes et variantes
Il existe cinq grandes familles d'histones : H1/H5 , H2A , H2B , H3 et H4 . Les histones H2A, H2B, H3 et H4 sont appelées histones centrales, tandis que les histones H1/H5 sont appelées histones de liaison.
Les histones centrales existent toutes sous forme de dimères , qui sont similaires en ce sens qu'elles possèdent toutes le domaine de repliement des histones : trois hélices alpha reliées par deux boucles. C'est cette structure hélicoïdale qui permet l'interaction entre des dimères distincts, en particulier de manière tête-queue (également appelée motif de poignée de main). Les quatre dimères distincts résultants se réunissent ensuite pour former un noyau de nucléosome octamère , d'environ 63 angströms de diamètre (une particule de type solénoïde (ADN) ). Environ 146 paires de bases (pb) d'ADN s'enroulent autour de cette particule centrale 1,65 fois dans un virage super-hélicoïdal à gauche pour donner une particule d'environ 100 angströms de diamètre. L'histone de liaison H1 lie le nucléosome aux sites d'entrée et de sortie de l'ADN, verrouillant ainsi l'ADN en place et permettant la formation d'une structure d'ordre supérieur. La formation la plus basique de ce type est la fibre ou les billes de 10 nm sur une conformation de chaîne. Cela implique l'enroulement de l'ADN autour des nucléosomes avec environ 50 paires de bases d' ADN séparant chaque paire de nucléosomes (également appelé ADN de liaison ). Les structures d'ordre supérieur comprennent la fibre de 30 nm (formant un zigzag irrégulier) et la fibre de 100 nm, ce sont les structures trouvées dans les cellules normales. Au cours de la mitose et de la méiose, les chromosomes condensés sont assemblés par des interactions entre les nucléosomes et d'autres protéines régulatrices.
Les histones sont subdivisées en histones canoniques dépendantes de la réplication qui sont exprimées pendant la phase S du cycle cellulaire et en variantes d'histones indépendantes de la réplication , exprimées pendant tout le cycle cellulaire. Chez les animaux, les gènes codant pour les histones canoniques sont généralement regroupés le long du chromosome, manquent d' introns et utilisent une structure tige-boucle à l' extrémité 3' au lieu d'une queue polyA . Les gènes codant pour les variantes d'histone ne sont généralement pas regroupés, ont des introns et leurs ARNm sont régulés par des queues polyA. Les organismes multicellulaires complexes ont généralement un nombre plus élevé de variantes d'histones offrant une variété de fonctions différentes. Des données récentes s'accumulent sur les rôles de diverses variantes d'histones mettant en évidence les liens fonctionnels entre les variantes et la régulation délicate du développement de l'organisme. Les variantes d'histone de différents organismes, leur classification et leurs caractéristiques spécifiques peuvent être trouvées dans la base de données "HistoneDB 2.0 - Variants" .
Voici une liste de protéines histones humaines :
| Super famille | Famille | Sous-famille | Membres |
|---|---|---|---|
| Éditeur de liens | H1 | H1F | H1F0 , H1FNT , H1FOO , H1FX |
| H1H1 | HIST1H1A , HIST1H1B , HIST1H1C , HIST1H1D , HIST1H1E , HIST1H1T | ||
| Coeur | H2A | H2AF | H2AFB1 , H2AFB2 , H2AFB3 , H2AFJ , H2AFV , H2AFX , H2AFY , H2AFY2 , H2AFZ |
| H2A1 | HIST1H2AA , HIST1H2AB , HIST1H2AC , HIST1H2AD , HIST1H2AE , HIST1H2AG , HIST1H2AI , HIST1H2AJ , HIST1H2AK , HIST1H2AL , HIST1H2AM | ||
| H2A2 | HIST2H2AA3 , HIST2H2AC | ||
| H2B | H2BF | H2BFM , H2BFS , H2BFWT | |
| H2B1 | HIST1H2BA , HIST1H2BB , HIST1H2BC , HIST1H2BD , HIST1H2B , HIST1H2BF , HIST1H2BG , HIST1H2BH , HIST1H2BI , HIST1H2BJ , HIST1H2BK , HIST1H2BK , HIST1H2H2BH , HIST1H2BI , HIST1H2BJ , HIST1H2BKBL1 , HIST2HBN1H2H2H | ||
| H2B2 | HIST2H2BE | ||
| H3 | H3A1 | HIST1H3A , HIST1H3B , HIST1H3C , HIST1H3D , HIST1H3E , HIST1H3F , HIST1H3G , HIST1H3H , HIST1H3I , HIST1H3J | |
| H3A2 | HIST2H3C | ||
| H3A3 | HIST3H3 | ||
| H4 | H41 | HIST1H4A , HIST1H4B , HIST1H4C , HIST1H4D , HIST1H4E , HIST1H4F , HIST1H4G , HIST1H4H , HIST1H4I , HIST1H4J , HIST1H4K , HIST1H4L | |
| H44 | HIST4H4 |
Structure
Le noyau du nucléosome est formé de deux dimères H2A-H2B et d'un tétramère H3-H4, formant deux moitiés presque symétriques par structure tertiaire ( symétrie C2 ; une macromolécule est l'image miroir de l'autre). Les dimères H2A-H2B et le tétramère H3-H4 présentent également une symétrie pseudodyadique. Les 4 histones « noyaux » (H2A, H2B, H3 et H4) ont une structure relativement similaire et sont hautement conservées au cours de l' évolution , toutes présentant un motif « hélice tour hélice tour hélice » (motif de protéine de liaison à l'ADN qui reconnaît une séquence d'ADN spécifique) . Ils partagent également la caractéristique de longues « queues » à une extrémité de la structure des acides aminés - c'est l'emplacement de la modification post-traductionnelle (voir ci-dessous).
L'histone archéenne ne contient qu'une structure dimère de type H3-H4, constituée de la même protéine. De telles structures dimères peuvent s'empiler en une grande superhélice ("hypernucléosome") sur laquelle l'ADN s'enroule d'une manière similaire aux bobines de nucléosomes. Seules certaines histones archées ont des queues.
La distance entre les bobines autour desquelles les cellules eucaryotes enroulent leur ADN a été déterminée entre 59 et 70 .
En tout, les histones font cinq types d'interactions avec l'ADN :
- Ponts salins et liaisons hydrogène entre les chaînes latérales d'acides aminés basiques (en particulier la lysine et l' arginine ) et les oxygènes phosphate sur l'ADN
- Les hélices -dipôles forment des hélices alpha dans H2B, H3 et H4 provoquent l'accumulation d'une charge positive nette au point d'interaction avec des groupes phosphates chargés négativement sur l'ADN
- Liaisons hydrogène entre le squelette de l'ADN et le groupe amide sur la chaîne principale des protéines histones
- Interactions non polaires entre les sucres histone et désoxyribose sur l'ADN
- Insertions de petits sillons non spécifiques des queues N-terminales H3 et H2B dans deux petits sillons chacun sur la molécule d'ADN
La nature très basique des histones, en plus de faciliter les interactions ADN-histones, contribue à leur solubilité dans l'eau.
Les histones sont sujettes à des modifications post-traductionnelles par des enzymes principalement sur leurs queues N-terminales, mais aussi dans leurs domaines globulaires. De telles modifications comprennent la méthylation , la citrullination , l' acétylation , la phosphorylation , la SUMOylation , l' ubiquitination et l' ADP-ribosylation . Cela affecte leur fonction de régulation des gènes.
En général, les gènes actifs ont moins d'histones liées, tandis que les gènes inactifs sont fortement associés aux histones pendant l' interphase . Il semble également que la structure des histones ait été conservée au cours de l' évolution , car toute mutation délétère serait gravement inadaptée. Toutes les histones ont une terminaison N fortement chargée positivement avec de nombreux résidus de lysine et d' arginine .
Évolution et distribution des espèces
Les histones centrales se trouvent dans les noyaux des cellules eucaryotes et dans la plupart des phylums d' archées , mais pas dans les bactéries . Cependant, les histones de liaison ont des homologues dans les bactéries. Les algues unicellulaires connues sous le nom de dinoflagellés étaient auparavant considérées comme les seuls eucaryotes à manquer complètement d'histones. Cependant, des études ultérieures ont montré que leur ADN code toujours pour les gènes des histones. Contrairement aux histones de base, les protéines d'histone de liaison riches en lysine (H1) se trouvent dans les bactéries, également connues sous le nom de nucléoprotéine HC1/HC2.
Il a été proposé que les protéines histones soient liées de manière évolutive à la partie hélicoïdale du domaine étendu AAA+ ATPase, au domaine C et au domaine de reconnaissance de substrat N-terminal des protéines Clp/Hsp100. Malgré les différences dans leur topologie, ces trois plis partagent un motif homologue hélice-brin-hélice (HSH).
Les histones archées pourraient bien ressembler aux précurseurs évolutifs des histones eucaryotes. De plus, les histones du nucléosome (noyau) peuvent avoir évolué à partir de protéines ribosomiques ( RPS6 / RPS15 ) avec lesquelles elles partagent de nombreux points communs, étant à la fois des protéines courtes et basiques. Les protéines histones sont parmi les protéines les plus conservées chez les eucaryotes, soulignant leur rôle important dans la biologie du noyau. En revanche, les spermatozoïdes matures utilisent largement des protamines pour emballer leur ADN génomique, très probablement parce que cela leur permet d'obtenir un rapport d'emballage encore plus élevé.
Il existe des variantes dans certaines des grandes classes. Ils partagent une homologie de séquence d'acides aminés et une similarité structurelle de base avec une classe spécifique d'histones majeures, mais ont également leur propre caractéristique qui est distincte des histones majeures. Ces histones mineures remplissent généralement des fonctions spécifiques du métabolisme de la chromatine. Par exemple, le CENPA de type histone H3 est associé uniquement à la région centromère du chromosome. Le variant d'histone H2A H2A.Z est associé aux promoteurs de gènes activement transcrits et également impliqué dans la prévention de la propagation de l' hétérochromatine silencieuse . De plus, H2A.Z joue un rôle dans la chromatine pour la stabilité du génome. Un autre variant H2A H2A.X est phosphorylé en S139 dans les régions autour des cassures double brin et marque la région en cours de réparation de l'ADN . L'histone H3.3 est associée au corps des gènes activement transcrits.
Fonction
Compacter des brins d'ADN
Les histones agissent comme des bobines autour desquelles s'enroule l'ADN. Cela permet le compactage nécessaire pour adapter les grands génomes des eucaryotes à l'intérieur des noyaux cellulaires : la molécule compactée est 40 000 fois plus courte qu'une molécule non compactée.
Régulation de la chromatine
Les histones subissent des modifications post-traductionnelles qui altèrent leur interaction avec l' ADN et les protéines nucléaires. Les histones H3 et H4 ont de longues queues dépassant du nucléosome , qui peuvent être modifiées de manière covalente à plusieurs endroits. Les modifications de la queue comprennent la méthylation , l' acétylation , la phosphorylation , l' ubiquitination , la SUMOylation , la citrullination et l'ADP-ribosylation. Le noyau des histones H2A et H2B peut également être modifié. On pense que des combinaisons de modifications constituent un code, le soi-disant « code histone ». Les modifications des histones agissent dans divers processus biologiques tels que la régulation des gènes , la réparation de l'ADN , la condensation des chromosomes ( mitose ) et la spermatogenèse ( méiose ).
La nomenclature commune des modifications des histones est :
- Le nom de l'histone (par exemple, H3)
- L' abréviation d' acide aminé à une lettre (par exemple, K pour Lysine ) et la position de l'acide aminé dans la protéine
- Le type de modification (Me : méthyle , P : phosphate , Ac : acétyle , Ub : ubiquitine )
- Le nombre de modifications (seul Me est connu pour se produire dans plus d'une copie par résidu. 1, 2 ou 3 est la mono-, di- ou tri-méthylation)
Ainsi, H3K4me1 désigne la monométhylation du 4ème résidu (une lysine) depuis le début (c'est-à-dire le N-terminal ) de la protéine H3.
| Type de modification |
Histone | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H3K36 | H4K20 | H2BK5 | H2BK20 | |
| mono- méthylation | Activation | Activation | Activation | Activation | Activation | Activation | |||
| diméthylation | répression | répression | Activation | ||||||
| triméthylation | Activation | répression | répression | activation, répression |
Activation | répression | |||
| acétylation | Activation | Activation | Activation | Activation | Activation | ||||
Modification
Un énorme catalogue de modifications d'histones a été décrit, mais une compréhension fonctionnelle de la plupart fait encore défaut. Collectivement, on pense que les modifications d'histones peuvent sous-tendre un code d'histones , les combinaisons de modifications d'histones ayant des significations spécifiques. Cependant, la plupart des données fonctionnelles concernent des modifications importantes des histones individuelles qui se prêtent biochimiquement à une étude détaillée.
Chimie
Méthylation de la lysine
L'ajout d'un, deux ou plusieurs groupes méthyle à la lysine a peu d'effet sur la chimie de l'histone ; la méthylation laisse la charge de la lysine intacte et ajoute un nombre minimal d'atomes, de sorte que les interactions stériques ne sont généralement pas affectées. Cependant, les protéines contenant des domaines Tudor, chromo ou PHD, entre autres, peuvent reconnaître la méthylation de la lysine avec une sensibilité exquise et différencier la mono, di et tri-méthyl lysine, dans la mesure où, pour certaines lysines (ex : H4K20) mono, di et tri -méthylation semblent avoir des significations différentes. Pour cette raison, la méthylation de la lysine a tendance à être une marque très informative et domine les fonctions de modification des histones connues.
Sérotonylation de la glutamine
Récemment, il a été démontré que l'ajout d'un groupe sérotonine à la glutamine en position 5 de H3, se produit dans les cellules sérotoninergiques telles que les neurones. Cela fait partie de la différenciation des cellules sérotoninergiques. Cette modification post-traductionnelle se produit en conjonction avec la modification H3K4me3. La sérotonylation potentialise la liaison du facteur de transcription général TFIID à la boîte TATA .
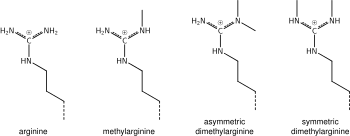
Méthylation de l'arginine
Ce qui a été dit ci-dessus de la chimie de la méthylation de la lysine s'applique également à la méthylation de l'arginine, et certains domaines protéiques, par exemple les domaines Tudor, peuvent être spécifiques de la méthyl arginine au lieu de la méthyl lysine. L'arginine est connue pour être mono- ou di-méthylée, et la méthylation peut être symétrique ou asymétrique, potentiellement avec des significations différentes.
Citrullination à l'arginine
Des enzymes appelées peptidylarginine déiminases (PAD) hydrolysent le groupe imine des arginines et attachent un groupe céto, de sorte qu'il y a une charge positive de moins sur le résidu d'acide aminé. Ce processus a été impliqué dans l'activation de l'expression des gènes en rendant les histones modifiées moins étroitement liées à l'ADN et en rendant ainsi la chromatine plus accessible. Les PAD peuvent également produire l'effet inverse en éliminant ou en inhibant la monométhylation des résidus d'arginine sur les histones et en s'opposant ainsi à l'effet positif de la méthylation de l'arginine sur l'activité transcriptionnelle.
Acétylation de la lysine
L'ajout d'un groupe acétyle a un effet chimique majeur sur la lysine car il neutralise la charge positive. Cela réduit l'attraction électrostatique entre l'histone et le squelette de l'ADN chargé négativement, relâchant la structure de la chromatine ; les histones hautement acétylées forment une chromatine plus accessible et ont tendance à être associées à une transcription active. L'acétylation de la lysine semble avoir une signification moins précise que la méthylation, en ce que les histone acétyltransférases ont tendance à agir sur plus d'une lysine; cela reflète probablement la nécessité de modifier plusieurs lysines pour avoir un effet significatif sur la structure de la chromatine. La modification comprend H3K27ac .
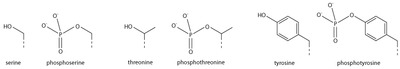
Phosphorylation sérine/thréonine/tyrosine
L'ajout d'un groupe phosphate chargé négativement peut entraîner des changements majeurs dans la structure des protéines, conduisant au rôle bien caractérisé de la phosphorylation dans le contrôle de la fonction des protéines. Les implications structurelles de la phosphorylation des histones ne sont pas claires, mais la phosphorylation des histones a des fonctions claires en tant que modification post-traductionnelle, et des domaines de liaison tels que BRCT ont été caractérisés.
Effets sur la transcription
La plupart des modifications d'histones bien étudiées sont impliquées dans le contrôle de la transcription.
Gènes transcrits activement
Deux modifications d'histones sont particulièrement associées à la transcription active :
- Triméthylation de H3 lysine 4 (H3K4me3)
- Cette triméthylation se produit au niveau du promoteur des gènes actifs et est réalisée par le complexe COMPASS . Malgré la conservation de ce complexe et la modification des histones de la levure aux mammifères, le rôle que joue cette modification n'est pas tout à fait clair. Cependant, c'est une excellente marque de promoteurs actifs et le niveau de cette modification d'histone au niveau du promoteur d'un gène est largement corrélé à l'activité transcriptionnelle du gène. La formation de cette marque est liée à la transcription de manière assez alambiquée : au début de la transcription d'un gène, l' ARN polymérase II subit un passage de l' initiation' à l' 'allongement' , marqué par un changement des états de phosphorylation de l' ARN polymérase II C domaine terminal (CTD) . La même enzyme qui phosphoryle le CTD phosphoryle également le complexe Rad6, qui à son tour ajoute une marque d'ubiquitine à H2B K123 (K120 chez les mammifères). H2BK123Ub se produit dans toutes les régions transcrites, mais cette marque est requise pour que COMPASS triméthyle H3K4 au niveau des promoteurs.
- Triméthylation de H3 lysine 36 ( H3K36me3 )
- Cette triméthylation se produit dans le corps des gènes actifs et est déposée par la méthyltransférase Set2. Cette protéine s'associe à l'allongement de l' ARN polymérase II et H3K36Me3 indique des gènes activement transcrits. H3K36Me3 est reconnu par le complexe d'histone désacétylase Rpd3, qui supprime les modifications acétyle des histones environnantes, augmentant la compaction de la chromatine et réprimant la transcription parasite. Une compaction accrue de la chromatine empêche les facteurs de transcription d'accéder à l'ADN et réduit la probabilité que de nouveaux événements de transcription soient initiés dans le corps du gène. Ce processus permet donc de s'assurer que la transcription n'est pas interrompue.
Gènes réprimés
Trois modifications d'histones sont particulièrement associées aux gènes réprimés :
- Triméthylation de la lysine H3 27 (H3K27me3)
- Cette modification des histones est déposée par le complexe polycomb PRC2. Il s'agit d'un marqueur clair de la répression génique, et est probablement lié par d'autres protéines pour exercer une fonction répressive. Un autre complexe polycomb , PRC1, peut se lier à H3K27me3 et ajoute la modification d'histone H2AK119Ub qui facilite le compactage de la chromatine. Sur la base de ces données, il apparaît que PRC1 est recruté par l'action de PRC2, cependant, des études récentes montrent que PRC1 est recruté sur les mêmes sites en l'absence de PRC2.
- Di et triméthylation de H3 lysine 9 (H3K9me2/3)
- H3K9me2/3 est un marqueur bien caractérisé de l' hétérochromatine et est donc fortement associé à la répression génique. La formation d'hétérochromatine a été mieux étudiée chez la levure Schizosaccharomyces pombe , où elle est initiée par le recrutement du complexe de silençage transcriptionnel induit par l' ARN (RITS) en ARN double brin produits à partir de répétitions centromériques . RITS recrute l' histone méthyltransférase Clr4 qui dépose H3K9me2/3. Ce processus est appelé méthylation des histones . H3K9Me2/3 sert de site de liaison pour le recrutement de Swi6 ( protéine d'hétérochromatine 1 ou HP1, un autre marqueur d'hétérochromatine classique) qui à son tour recrute d'autres activités répressives, notamment des modificateurs d'histones tels que les histones désacétylases et les histones méthyltransférases .
- Triméthylation de H4 lysine 20 ( H4K20me 3)
- Cette modification est étroitement associée à l'hétérochromatine, bien que son importance fonctionnelle reste incertaine. Cette marque est placée par la méthyltransférase Suv4-20h, qui est au moins en partie recrutée par la protéine hétérochromatine 1 .
Promoteurs bivalents
L'analyse des modifications des histones dans les cellules souches embryonnaires (et autres cellules souches) a révélé de nombreux promoteurs de gènes portant à la fois H3K4Me3 et H3K27Me3 , en d'autres termes, ces promoteurs présentent à la fois des marques d'activation et de répression. Cette combinaison particulière de modifications marque les gènes prêts pour la transcription ; ils ne sont pas requis dans les cellules souches, mais sont rapidement requis après différenciation en certaines lignées. Une fois que la cellule commence à se différencier, ces promoteurs bivalents sont résolus en états actifs ou répressifs en fonction de la lignée choisie.
Autres fonctions
Dommages à l'ADN
Le marquage des sites de dommages à l'ADN est une fonction importante pour les modifications des histones. Il protège également l'ADN de la destruction par les rayons ultraviolets du soleil.
- Phosphorylation de H2AX à la sérine 139 (γH2AX)
- Le H2AX phosphorylé (également connu sous le nom de gamma H2AX) est un marqueur des cassures double brin de l' ADN et fait partie de la réponse aux dommages de l'ADN . H2AX est phosphorylé tôt après la détection d'une rupture double brin de l'ADN et forme un domaine s'étendant sur plusieurs kilobases de chaque côté des dommages. Le gamma H2AX agit comme un site de liaison pour la protéine MDC1, qui à son tour recrute des protéines clés de réparation de l'ADN (ce sujet complexe est bien examiné dans) et en tant que tel, le gamma H2AX constitue une partie vitale de la machinerie qui assure la stabilité du génome.
- Acétylation de H3 lysine 56 (H3K56Ac)
- H3K56Acx est requis pour la stabilité du génome. H3K56 est acétylé par le complexe p300/Rtt109, mais est rapidement désacétylé autour des sites de dommages à l'ADN. L'acétylation H3K56 est également nécessaire pour stabiliser les fourches de réplication bloquées, empêchant ainsi les effondrements dangereux des fourches de réplication. Bien qu'en général les mammifères utilisent beaucoup plus les modifications des histones que les micro-organismes, un rôle majeur de H3K56Ac dans la réplication de l'ADN n'existe que chez les champignons, et cela est devenu une cible pour le développement d'antibiotiques.
réparation de l'ADN
- Triméthylation de H3 lysine 36 (H3K36me3)
H3K36me3 a la capacité de recruter le complexe MSH2-MSH6 (hMutSα) de la voie de réparation des mésappariements de l' ADN . Constamment, les régions du génome humain avec des niveaux élevés de H3K36me3 accumulent moins de mutations somatiques en raison de l' activité de réparation des mésappariements .
Condensation chromosomique
- Phosphorylation de H3 à la sérine 10 (phospho-H3S10)
- La kinase mitotique Aurora B phosphoryle l'histone H3 au niveau de la sérine 10, déclenchant une cascade de changements qui interviennent dans la condensation mitotique des chromosomes. Les chromosomes condensés se colorent donc très fortement pour cette marque, mais la phosphorylation de H3S10 est également présente sur certains sites chromosomiques en dehors de la mitose, par exemple dans l'hétérochromatine péricentrique des cellules au cours de G2. La phosphorylation de H3S10 a également été liée à des dommages à l'ADN causés par la formation de boucles R sur des sites hautement transcrits.
- Phosphorylation H2B à sérine 10/14 (phospho-H2BS10/14)
- La phosphorylation de H2B au niveau de la sérine 10 (levure) ou de la sérine 14 (mammifères) est également liée à la condensation de la chromatine, mais dans le but très différent de médier la condensation chromosomique lors de l'apoptose. Cette marque n'est pas simplement un témoin agissant tardivement dans l'apoptose, car les levures porteuses de mutations de ce résidu sont résistantes à la mort cellulaire apoptotique induite par le peroxyde d'hydrogène.
Dépendance
Les modifications épigénétiques des queues d'histone dans des régions spécifiques du cerveau sont d'une importance centrale dans les addictions. Une fois que des altérations épigénétiques particulières se produisent, elles semblent être des "cicatrices moléculaires" durables qui peuvent expliquer la persistance des dépendances.
Les fumeurs de cigarettes (environ 15 % de la population américaine) sont généralement dépendants de la nicotine . Après 7 jours de traitement à la nicotine des souris, l'acétylation de l'histone H3 et de l'histone H4 a augmenté au niveau du promoteur FosB dans le noyau accumbens du cerveau, provoquant une augmentation de 61 % de l'expression de FosB. Cela augmenterait également l'expression du variant d'épissage Delta FosB . Dans le noyau accumbens du cerveau, Delta FosB fonctionne comme un « commutateur moléculaire soutenu » et une « protéine de contrôle principale » dans le développement d'une dépendance .
Environ 7% de la population américaine est accro à l' alcool . Chez les rats exposés à l'alcool jusqu'à 5 jours, il y a eu une augmentation de l'acétylation de l'histone 3 lysine 9 dans le promoteur de la pronociceptine dans le complexe de l' amygdale cérébrale . Cette acétylation est un marqueur d'activation de la pronociceptine. Le système de récepteurs opioïdes nociceptine/nociceptine est impliqué dans les effets de renforcement ou de conditionnement de l'alcool.
La dépendance à la méthamphétamine survient chez environ 0,2% de la population américaine. La consommation chronique de méthamphétamine provoque la méthylation de la lysine en position 4 de l'histone 3 située au niveau des promoteurs des gènes c-fos et CC chemokine receptor 2 (ccr2) , activant ces gènes dans le noyau accumbens (NAc). c-fos est bien connu pour être important dans la toxicomanie . Le gène ccr2 est également important dans la toxicomanie, car l'inactivation mutationnelle de ce gène altère la dépendance.
Synthèse
La première étape de la duplication de la structure de la chromatine est la synthèse des protéines histones : H1, H2A, H2B, H3, H4. Ces protéines sont synthétisées au cours de la phase S du cycle cellulaire. Il existe différents mécanismes qui contribuent à l'augmentation de la synthèse des histones.
Levure
Les levures portent une ou deux copies de chaque gène d'histone, qui ne sont pas regroupées mais plutôt dispersées dans les chromosomes. La transcription du gène des histones est contrôlée par de multiples protéines régulatrices des gènes telles que les facteurs de transcription qui se lient aux régions promotrices des histones. Chez la levure bourgeonnante, le gène candidat pour l'activation de l'expression du gène des histones est le SBF. Le SBF est un facteur de transcription qui est activé en phase G1 tardive, lorsqu'il se dissocie de son répresseur Whi5 . Cela se produit lorsque Whi5 est phosphorylé par Cdc8 qui est un G1/S Cdk. La suppression de l'expression du gène des histones en dehors des phases S dépend des protéines Hir qui forment une structure de chromatine inactive au locus des gènes des histones, provoquant le blocage des activateurs de la transcription.
Métazoaire
Chez les métazoaires, l'augmentation du taux de synthèse des histones est due à l'augmentation du traitement du pré-ARNm en sa forme mature ainsi qu'à la diminution de la dégradation de l'ARNm ; cela se traduit par une augmentation de l'ARNm actif pour la traduction des protéines histones. Le mécanisme d'activation de l'ARNm s'est avéré être l'élimination d'un segment de l'extrémité 3' du brin d'ARNm et dépend de l'association avec la protéine de liaison tige-boucle ( SLBP ). SLBP stabilise également les ARNm des histones pendant la phase S en bloquant la dégradation par la nucléase 3'hExo. Les niveaux de SLBP sont contrôlés par les protéines du cycle cellulaire, provoquant l'accumulation de SLBP lorsque les cellules entrent en phase S et se dégradent lorsque les cellules quittent la phase S. Les SLBP sont marquées pour la dégradation par phosphorylation au niveau de deux résidus thréonine par des kinases dépendantes de la cycline, éventuellement la cycline A/cdk2, à la fin de la phase S. Les métazoaires ont également de multiples copies de gènes d'histones regroupés sur des chromosomes qui sont localisés dans des structures appelées corps de Cajal, telles que déterminées par l'analyse de capture de conformation chromosomique à l'échelle du génome (4C-Seq).
Lien entre le contrôle du cycle cellulaire et la synthèse
L'ataxie-télangiectasie nucléaire (NPAT), également connue sous le nom de coactivateur protéique nucléaire de la transcription des histones, est un facteur de transcription qui active la transcription du gène des histones sur les chromosomes 1 et 6 des cellules humaines. NPAT est également un substrat de la cycline E-Cdk2, qui est nécessaire pour la transition entre la phase G1 et la phase S. Le NPAT n'active l'expression du gène des histones qu'après avoir été phosphorylé par la cycline G1/S-Cdk E-Cdk2 au début de la phase S. Cela montre un lien régulateur important entre le contrôle du cycle cellulaire et la synthèse des histones.
Histoire
Les histones ont été découvertes en 1884 par Albrecht Kossel . Le mot "histone" date de la fin du 19ème siècle et est dérivé du mot allemand "Histon" , un mot lui-même d'origine incertaine, peut-être du grec ancien ἵστημι (hístēmi, "se tenir debout") ou ἱστός (histós, "tisser" ).
Au début des années 1960, avant que les types d'histones ne soient connus et avant que les histones soient connues pour être hautement conservées dans des organismes taxonomiquement divers, James F. Bonner et ses collaborateurs ont commencé une étude de ces protéines qui étaient connues pour être étroitement associées à l'ADN dans le noyau des organismes supérieurs. Bonner et son boursier postdoctoral Ru Chih C. Huang ont montré que la chromatine isolée ne prendrait pas en charge la transcription de l'ARN dans le tube à essai, mais si les histones étaient extraites de la chromatine, l'ARN pourrait être transcrit à partir de l'ADN restant. Leur article est devenu un classique de la citation. Paul T'so et James Bonner avaient convoqué un Congrès mondial sur la chimie et la biologie des histones en 1964, au cours duquel il est devenu clair qu'il n'y avait pas de consensus sur le nombre de types d'histones et que personne ne savait comment ils se compareraient lorsqu'ils seraient isolés de différents organismes. Bonner et ses collaborateurs ont ensuite développé des méthodes pour séparer chaque type d'histone, purifié des histones individuelles, comparé les compositions d'acides aminés dans la même histone de différents organismes et comparé les séquences d'acides aminés de la même histone de différents organismes en collaboration avec Emil Smith de l'UCLA. Par exemple, ils ont découvert que la séquence d'Histone IV était hautement conservée entre les pois et le thymus de veau. Cependant, leurs travaux sur les caractéristiques biochimiques des histones individuelles n'ont pas révélé comment les histones interagissaient les unes avec les autres ou avec l'ADN auquel elles étaient étroitement liées.
Toujours dans les années 1960, Vincent Allfrey et Alfred Mirsky avaient suggéré, sur la base de leurs analyses des histones, que l'acétylation et la méthylation des histones pourraient fournir un mécanisme de contrôle transcriptionnel, mais ne disposaient pas du type d'analyse détaillée que les chercheurs ultérieurs ont pu mener. pour montrer comment une telle régulation pourrait être spécifique d'un gène. Jusqu'au début des années 1990, les histones étaient généralement rejetées comme matériau de remplissage inerte pour l'ADN nucléaire eucaryote, une opinion basée en partie sur les modèles de Mark Ptashne et d'autres, qui pensaient que la transcription était activée par des interactions protéine-ADN et protéine-protéine sur une grande partie modèles d'ADN nus, comme c'est le cas chez les bactéries.
Au cours des années 1980, Yahli Lorch et Roger Kornberg ont montré qu'un nucléosome sur un promoteur central empêche l'initiation de la transcription in vitro, et Michael Grunstein a démontré que les histones répriment la transcription in vivo, conduisant à l'idée du nucléosome comme répresseur général des gènes. On pense que le soulagement de la répression implique à la fois la modification des histones et l'action des complexes de remodelage de la chromatine. Vincent Allfrey et Alfred Mirsky avaient précédemment proposé un rôle de la modification des histones dans l'activation transcriptionnelle, considérée comme une manifestation moléculaire de l'épigénétique. Michael Grunstein et David Allis ont trouvé un soutien pour cette proposition, dans l'importance de l'acétylation des histones pour la transcription chez la levure et l'activité de l'activateur transcriptionnel Gcn5 en tant qu'histone acétyltransférase.
La découverte de l'histone H5 semble remonter aux années 1970, et elle est maintenant considérée comme une isoforme de l' Histone H1 .