Microbiote - Microbiota
Les microbiotes sont des "communautés écologiques de micro-organismes commensaux , symbiotiques et pathogènes " trouvées dans et sur tous les organismes multicellulaires étudiés à ce jour, des plantes aux animaux. Le microbiote comprend les bactéries, les archées , les protistes, les champignons et les virus. Le microbiote s'est avéré crucial pour l' homéostasie immunologique, hormonale et métabolique de leur hôte. Le terme microbiome décrit soit les génomes collectifs des micro-organismes qui résident dans une niche environnementale, soit les micro-organismes eux-mêmes.
Le microbiome et l'hôte ont émergé au cours de l' évolution en tant qu'unité synergique à partir de l' épigénétique et des caractéristiques génétiques, parfois appelées collectivement holobionte . La présence de microbiote humain et d'autres intestins de métazoaires a été essentielle pour comprendre la co-évolution entre les métazoaires et les bactéries. Le microbiote joue un rôle clé dans les réponses immunitaires et métaboliques intestinales via leur produit de fermentation ( acide gras à chaîne courte ), l' acétate .
introduction
Toutes les plantes et tous les animaux, des formes de vie simples aux humains, vivent en étroite association avec des organismes microbiens. Plusieurs avancées ont stimulé la perception des microbiomes, notamment :
- la capacité d'effectuer des analyses d'expression génomique et génique de cellules individuelles et de communautés microbiennes entières dans les disciplines de la métagénomique et de la métatranscriptomique
- bases de données accessibles aux chercheurs de plusieurs disciplines
- méthodes d'analyse mathématique adaptées à des ensembles de données complexes
Les biologistes en sont venus à comprendre que les microbes constituent une partie importante du phénotype d'un organisme , bien au-delà de l'étude de cas symbiotique occasionnelle.
Types de relations microbe-hôte
Le commensalisme , concept développé par Pierre-Joseph van Beneden (1809-1894), professeur belge à l' Université de Louvain au XIXe siècle est au cœur du microbiome, où le microbiote colonise un hôte dans une coexistence non nocive. La relation avec leur hôte est dite mutualiste lorsque les organismes effectuent des tâches qui sont connues pour être utiles pour l'hôte, parasitaires , lorsqu'elles sont désavantageuses pour l'hôte. D'autres auteurs définissent une situation comme mutualiste où les deux bénéficient, et commensal, où l'hôte non affecté profite au symbiote. Un échange de nutriments peut être bidirectionnel ou unidirectionnel, peut dépendre du contexte et peut se produire de diverses manières. Le microbiote dont on s'attend à ce qu'il soit présent et qui, dans des circonstances normales, ne provoque pas de maladie, est considéré comme une flore normale ou un microbiote normal ; la flore normale peut non seulement être inoffensive, mais peut également protéger l'hôte.
Acquisition et changement
L' acquisition initiale du microbiote chez les animaux, des mammifères aux éponges marines, se fait à la naissance et peut même se produire à travers la lignée cellulaire germinale. Chez les plantes, le processus de colonisation peut être initié sous terre dans la zone racinaire , autour de la graine en germination, la spermosphère , ou provenir des parties aériennes, la phyllosphère et la zone florale ou anthosphère. La stabilité du microbiote de la rhizosphère au fil des générations dépend du type de plante mais plus encore de la composition du sol, c'est-à-dire du milieu vivant et non vivant. Cliniquement, un nouveau microbiote peut être acquis par greffe de microbiote fécal pour traiter des infections telles que l' infection chronique à C. difficile .
Microbiote par hôte
Humains
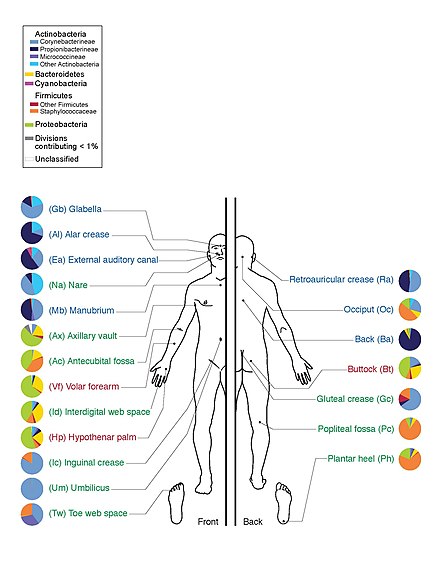
Le microbiote humain comprend des bactéries , des champignons , des archées et des virus. Les micro-animaux qui vivent sur le corps humain sont exclus. Le microbiome humain fait référence à leurs génomes .
Les humains sont colonisés par de nombreux micro-organismes ; l'estimation traditionnelle était que les humains vivent avec dix fois plus de cellules non humaines que de cellules humaines ; des estimations plus récentes ont abaissé ce chiffre à 3:1 et même à environ 1:1.
En fait, ceux-ci sont si petits qu'il y a environ 100 000 milliards de microbiotes sur le corps humain.
Le Human Microbiome Project a séquencé le génome du microbiote humain, en se concentrant particulièrement sur le microbiote qui habite normalement la peau, la bouche, le nez, le tube digestif et le vagin. Elle a franchi une étape importante en 2012 avec la publication des premiers résultats.
Animaux non humains
- Les amphibiens ont un microbiote sur leur peau. Certaines espèces sont capables de porter un champignon nommé Batrachochytrium dendrobatidis , qui chez d'autres peut provoquer une infection mortelle Chytridiomycose en fonction de leur microbiome, résistant à la colonisation par des agents pathogènes ou inhibant leur croissance avec des peptides cutanés antimicrobiens.
- Chez les mammifères, les herbivores tels que les bovins dépendent de leur microbiome ruminal pour convertir la cellulose en protéines, en acides gras à chaîne courte et en gaz. Les méthodes de culture ne peuvent pas fournir d'informations sur tous les micro-organismes présents. Des études métagénomiques comparatives ont donné le résultat surprenant que les bovins individuels possèdent des structures communautaires, des phénotypes prédits et des potentiels métaboliques nettement différents, même s'ils étaient nourris avec des régimes identiques, étaient logés ensemble et étaient apparemment fonctionnellement identiques dans leur utilisation des ressources de la paroi cellulaire végétale.
- Les souris sont devenues les mammifères les plus étudiés en ce qui concerne leurs microbiomes. Le microbiote intestinal a été étudié en relation avec les maladies allergiques des voies respiratoires, l'obésité, les maladies gastro-intestinales et le diabète. Le déplacement périnatal du microbiote par des antibiotiques à faible dose peut avoir des effets durables sur la susceptibilité future aux maladies allergiques des voies respiratoires. La fréquence de certains sous-ensembles de microbes a été liée à la gravité de la maladie. La présence de microbes spécifiques au début de la vie postnatale instruit les futures réponses immunitaires. Chez les souris gnotobiotiques, certaines bactéries intestinales se sont avérées transmettre un phénotype particulier aux souris receveuses exemptes de germes, ce qui a favorisé l'accumulation de cellules T régulatrices du côlon, et des souches qui ont modulé l'adiposité de la souris et les concentrations de métabolites caecaux. Cette approche combinatoire permet une compréhension au niveau des systèmes des contributions microbiennes à la biologie humaine. Mais aussi d'autres tissus mucoïdes comme les poumons et le vagin ont été étudiés en relation avec des maladies telles que l'asthme, l'allergie et la vaginose.
- Les insectes ont leurs propres microbiomes. Par exemple, les fourmis coupeuses de feuilles forment d'énormes colonies souterraines qui récoltent des centaines de kilogrammes de feuilles chaque année et sont incapables de digérer directement la cellulose des feuilles. Ils entretiennent des jardins de champignons comme principale source de nourriture de la colonie. Alors que le champignon lui-même ne digère pas la cellulose, une communauté microbienne contenant une diversité de bactéries le fait. L'analyse du génome de la population microbienne a révélé de nombreux gènes jouant un rôle dans la digestion de la cellulose. Le profil enzymatique de dégradation des glucides prédit de ce microbiome est similaire à celui du rumen bovin, mais la composition des espèces est presque entièrement différente. Le microbiote intestinal de la mouche des fruits peut affecter l'apparence de son intestin, en affectant le taux de renouvellement épithélial, l'espacement cellulaire et la composition des différents types de cellules dans l'épithélium. Lorsque le papillon Spodoptera exigua est infecté par le baculovirus, les gènes liés au système immunitaire sont régulés à la baisse et la quantité de son microbiote intestinal augmente. Dans l'intestin des diptères, les cellules entéroendocrines détectent les métabolites dérivés du microbiote intestinal et coordonnent les branches antibactériennes, mécaniques et métaboliques de la réponse immunitaire innée intestinale de l'hôte au microbiote commensal.
- Les poissons ont leurs propres microbiomes, y compris l'espèce à courte durée de vie Nothobranchius furzeri (killifish turquoise). Le transfert du microbiote intestinal de jeunes killfish à des killifish d'âge moyen prolonge considérablement la durée de vie des killifish d'âge moyen.
Les plantes
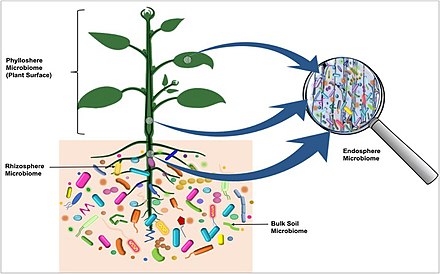
On a récemment découvert que le microbiome végétal provenait de la graine. Les micro-organismes transmis par les graines migrent dans la plantule en développement par une voie spécifique dans laquelle certaines communautés se déplacent vers les feuilles et d'autres vers les racines. Dans le schéma de droite, le microbiote colonisant la rhizosphère , entrant dans les racines et colonisant la prochaine génération de tubercules via les stolons , est visualisé avec une couleur rouge. Les bactéries présentes dans le tubercule mère , traversant les stolons et migrant dans la plante ainsi que dans la prochaine génération de tubercules sont indiquées en bleu.
- Le sol est le principal réservoir des bactéries qui colonisent les tubercules de pomme de terre
- Les bactéries sont recrutées dans le sol plus ou moins indépendamment de la variété de pomme de terre
- Les bactéries pourraient coloniser les tubercules principalement de l'intérieur des plantes via le stolon
- Le microbiote bactérien des tubercules de pomme de terre est constitué de bactéries transmises d'une génération de tubercules à l'autre et de bactéries recrutées dans le sol colonisent les plants de pomme de terre via la racine.
Les plantes sont des hôtes attrayants pour les micro-organismes car elles fournissent une variété de nutriments. Les micro-organismes sur les plantes peuvent être des épiphytes (trouvés sur les plantes) ou des endophytes (trouvés à l'intérieur des tissus végétaux). Les oomycètes et les champignons ont, grâce à une évolution convergente, développé une morphologie similaire et occupent des niches écologiques similaires. Ils développent des hyphes , des structures filiformes qui pénètrent dans la cellule hôte. Dans des situations mutualistes , la plante échange souvent des sucres hexose contre du phosphate inorganique du symbiote fongique. On suppose que de telles associations très anciennes ont aidé les plantes lorsqu'elles ont colonisé la terre pour la première fois. Les bactéries favorisant la croissance des plantes (PGPB) fournissent à la plante des services essentiels tels que la fixation de l'azote , la solubilisation des minéraux tels que le phosphore, la synthèse des hormones végétales , l'amélioration directe de l'absorption des minéraux et la protection contre les agents pathogènes. Les PGPB peuvent protéger les plantes des agents pathogènes en entrant en compétition avec l'agent pathogène pour une niche écologique ou un substrat, en produisant des produits allélochimiques inhibiteurs ou en induisant une résistance systémique des plantes hôtes à l'agent pathogène.
Recherche
La relation symbiotique entre un hôte et son microbiote fait l'objet de recherches en laboratoire sur la façon dont elle peut façonner le système immunitaire des mammifères. Chez de nombreux animaux, le système immunitaire et le microbiote peuvent s'engager dans une "diaphonie" en échangeant des signaux chimiques, ce qui peut permettre au microbiote d'influencer la réactivité immunitaire et le ciblage. Les bactéries peuvent être transmises de la mère à l'enfant par contact direct et après la naissance . Au fur et à mesure que le microbiome du nourrisson est établi, les bactéries commensales peuplent rapidement l'intestin, provoquant une gamme de réponses immunitaires et "programmant" le système immunitaire avec des effets durables. Les bactéries sont capables de stimuler le tissu lymphoïde associé à la muqueuse intestinale, ce qui permet au tissu de produire des anticorps contre les agents pathogènes susceptibles de pénétrer dans l'intestin.
Le microbiome humain peut jouer un rôle dans l'activation de récepteurs de type péage dans les intestins, un type de récepteur de reconnaissance de formes que les cellules hôtes utilisent pour reconnaître les dangers et réparer les dommages. Les agents pathogènes peuvent influencer cette coexistence conduisant à une dérégulation immunitaire, y compris une susceptibilité aux maladies, aux mécanismes d' inflammation , à la tolérance immunitaire et aux maladies auto-immunes .
Co-évolution du microbiote

Les organismes évoluent au sein des écosystèmes de sorte que le changement d'un organisme affecte le changement des autres. La théorie de l'évolution de l'hologénome propose qu'un objet de la sélection naturelle n'est pas l'organisme individuel, mais l'organisme avec ses organismes associés, y compris ses communautés microbiennes.
Récifs coralliens . La théorie de l'hologénome trouve son origine dans les études sur les récifs coralliens. Les récifs coralliens sont les plus grandes structures créées par des organismes vivants et contiennent des communautés microbiennes abondantes et très complexes. Au cours des dernières décennies, des déclins majeurs des populations de coraux se sont produits. Le changement climatique , la pollution de l'eau et la surpêche sont trois facteurs de stress qui ont été décrits comme menant à la susceptibilité aux maladies. Plus de vingt maladies différentes des coraux ont été décrites, mais parmi celles-ci, seule une poignée a vu ses agents responsables isolés et caractérisés. Le blanchissement des coraux est la plus grave de ces maladies. En mer Méditerranée, le blanchissement d' Oculina patagonica a été décrit pour la première fois en 1994 et a été rapidement déterminé comme étant dû à une infection par Vibrio shiloi . De 1994 à 2002, le blanchissement bactérien d' O. patagonica s'est produit chaque été en Méditerranée orientale. Étonnamment, cependant, après 2003, O. patagonica en Méditerranée orientale a été résistant à l' infection à V. shiloi , bien que d'autres maladies causent encore le blanchissement. La surprise vient du fait que les coraux vivent longtemps, avec une durée de vie de l'ordre de plusieurs décennies, et n'ont pas de système immunitaire adaptatif . Leurs systèmes immunitaires innés ne produisent pas d'anticorps et ils ne devraient apparemment pas être en mesure de répondre à de nouveaux défis, sauf sur des échelles de temps évolutives.
L'énigme de la façon dont les coraux ont réussi à acquérir une résistance à un agent pathogène spécifique a conduit à une proposition en 2007, selon laquelle une relation dynamique existe entre les coraux et leurs communautés microbiennes symbiotiques. On pense qu'en modifiant sa composition, l' holobionte peut s'adapter aux conditions environnementales changeantes beaucoup plus rapidement que par mutation génétique et sélection seules. L'extrapolation de cette hypothèse à d'autres organismes, y compris les plantes et les animaux supérieurs, a conduit à la proposition de la théorie de l'évolution de l'hologénome.
En 2007, la théorie de l'hologénome était encore débattue. Une critique majeure a été l'affirmation selon laquelle V. shiloi a été identifié à tort comme l'agent causal du blanchissement des coraux, et que sa présence dans O. patagonica blanchi était simplement celle d'une colonisation opportuniste. Si cela est vrai, l'observation de base menant à la théorie serait invalide. La théorie a gagné en popularité en tant que moyen d'expliquer les changements rapides d'adaptation qui ne peuvent pas autrement être expliqués par les mécanismes traditionnels de sélection naturelle. Au sein de la théorie de l'hologénome, l'holobionte est non seulement devenu l'unité principale de la sélection naturelle mais aussi le résultat d'autres étapes d'intégration qu'il est également observé au niveau cellulaire ( symbiogenèse , endosymbiose ) et génomique.
Méthodes de recherche
Séquençage ciblé d'amplicons
| Fait partie d'une série sur |
| Microbiomes |
|---|
 |
Le séquençage ciblé des amplicons repose sur le fait d'avoir certaines attentes quant à la composition de la communauté étudiée. Dans le séquençage d'amplicons cibles, un marqueur informatif phylogénétiquement est ciblé pour le séquençage. Un tel marqueur devrait être présent dans idéalement tous les organismes attendus. Il devrait également évoluer de manière à être suffisamment conservé pour que les amorces puissent cibler les gènes d'un large éventail d'organismes tout en évoluant suffisamment rapidement pour permettre une résolution plus fine au niveau taxonomique. Un marqueur commun pour les études sur le microbiome humain est le gène de l'ARNr 16S bactérien ( c'est-à-dire « l'ADNr 16S », la séquence d'ADN qui code pour la molécule d'ARN ribosomique). Étant donné que les ribosomes sont présents dans tous les organismes vivants, l'utilisation de l'ADNr 16S permet d'amplifier l'ADN à partir de beaucoup plus d'organismes que si un autre marqueur était utilisé. Le gène de l'ADNr 16S contient à la fois des régions à évolution lente et des régions à évolution rapide ; le premier peut être utilisé pour concevoir des amorces larges tandis que le second permet une distinction taxonomique plus fine. Cependant, la résolution au niveau de l'espèce n'est généralement pas possible en utilisant l'ADNr 16S. La sélection de l'amorce est une étape importante, car tout ce qui ne peut pas être ciblé par l'amorce ne sera pas amplifié et ne sera donc pas détecté. Il a été démontré que différents ensembles d'amorces amplifient différents groupes taxonomiques en raison de la variation de séquence.
Les études ciblées des communautés eucaryotes et virales sont limitées et soumises au défi d'exclure l'ADN de l'hôte de l'amplification et de la biomasse eucaryote et virale réduite dans le microbiome humain.
Une fois les amplicons séquencés, des méthodes de phylogénétique moléculaire sont utilisées pour déduire la composition de la communauté microbienne. Cela se fait en regroupant les amplicons en unités taxonomiques opérationnelles (OTU) et en inférant les relations phylogénétiques entre les séquences. En raison de la complexité des données, les mesures de distance telles que les distances UniFrac sont généralement définies entre les échantillons de microbiome, et des méthodes multivariées en aval sont effectuées sur les matrices de distance. Un point important est que l'échelle des données est vaste et que d'autres approches doivent être adoptées pour identifier des modèles à partir des informations disponibles. Les outils utilisés pour analyser les données incluent VAMPS, QIIME et mothur .
Séquençage métagénomique
La métagénomique est également largement utilisée pour étudier les communautés microbiennes. Dans le séquençage métagénomique, l'ADN est récupéré directement à partir d'échantillons environnementaux de manière non ciblée dans le but d'obtenir un échantillon impartial de tous les gènes de tous les membres de la communauté. Des études récentes utilisent le séquençage ou le pyroséquençage shotgun de Sanger pour récupérer les séquences des lectures. Les lectures peuvent ensuite être assemblées en contigs . Pour déterminer l'identité phylogénétique d'une séquence, celle-ci est comparée aux séquences complètes du génome disponibles à l'aide de méthodes telles que BLAST . Un inconvénient de cette approche est que de nombreux membres des communautés microbiennes n'ont pas de génome séquencé représentatif, mais cela s'applique également au séquençage d'amplicons d'ARNr 16S et constitue un problème fondamental. Avec le séquençage shotgun, il peut être résolu en ayant une couverture élevée (50-100x) du génome inconnu, réalisant efficacement un assemblage du génome de novo . Dès qu'il existe un génome complet d'un organisme inconnu, il peut être comparé phylogénétiquement et l'organisme mis à sa place dans l' arbre de vie , en créant de nouveaux taxons . Une approche émergente consiste à combiner le séquençage au fusil de chasse avec des données de ligature de proximité ( Hi-C ) pour assembler des génomes microbiens complets sans culture.
Malgré le fait que la métagénomique soit limitée par la disponibilité de séquences de référence, un avantage significatif de la métagénomique par rapport au séquençage ciblé d'amplicons est que les données de métagénomique peuvent élucider le potentiel fonctionnel de l'ADN communautaire. Les études de gènes ciblées ne peuvent pas le faire car elles ne révèlent que la relation phylogénétique entre le même gène provenant d'organismes différents. L'analyse fonctionnelle est effectuée en comparant les séquences récupérées à des bases de données d'annotations métagénomiques telles que KEGG . Les voies métaboliques dans lesquelles ces gènes sont impliqués peuvent ensuite être prédites avec des outils tels que MG-RAST, CAMERA et IMG/M .
Approches basées sur l'ARN et les protéines
Des études de métatranscriptomique ont été réalisées pour étudier l'expression génique des communautés microbiennes grâce à des méthodes telles que le pyroséquençage de l'ARN extrait. Des études basées sur la structure ont également identifié des ARN non codants (ARNnc) tels que les ribozymes du microbiote. La métaprotéomique est une approche qui étudie les protéines exprimées par le microbiote, donnant un aperçu de son potentiel fonctionnel.
Projets
Le projet sur le microbiome humain lancé en 2008 était une initiative des National Institutes of Health des États-Unis visant à identifier et à caractériser les micro-organismes trouvés chez les humains sains et malades. Le projet de cinq ans, mieux caractérisé comme une étude de faisabilité avec un budget de 115 millions de dollars, a testé comment les changements dans le microbiome humain sont associés à la santé ou à la maladie humaine.
Le Earth Microbiome Project (EMP) est une initiative visant à collecter des échantillons naturels et à analyser la communauté microbienne à travers le monde. Les microbes sont très abondants, diversifiés et jouent un rôle important dans le système écologique. Pourtant, en 2010, on estimait que l'effort total de séquençage de l'ADN environnemental mondial avait produit moins de 1% de l'ADN total trouvé dans un litre d'eau de mer ou un gramme de sol, et les interactions spécifiques entre les microbes sont largement inconnues. L'EMP vise à traiter jusqu'à 200 000 échantillons dans différents biomes, générant une base de données complète de microbes sur terre pour caractériser les environnements et les écosystèmes par la composition et l'interaction microbiennes. En utilisant ces données, de nouvelles théories écologiques et évolutives peuvent être proposées et testées.
Microbiote intestinal et diabète de type 2
Le microbiote intestinal est très important pour la santé de l'hôte car il joue un rôle dans la dégradation des polysaccharides non digestibles (fermentation d'amidon résistant, oligosaccharides, inuline) renforçant l'intégrité intestinale ou façonnant l'épithélium intestinal, récupérant l'énergie, protégeant contre les agents pathogènes et régulant l'hôte immunité.
Plusieurs études ont montré que la composition bactérienne intestinale chez les patients diabétiques était altérée avec des niveaux accrus de membres de Lactobacillus gasseri , Streptococcus mutans et Clostridiales, avec une diminution des bactéries productrices de butyrate telles que Roseburia intestinalis et Faecalibacterium prausnitzii . Cette altération est due à de nombreux facteurs tels que l'abus d'antibiotiques, l'alimentation et l'âge .
La diminution de la production de butyrate est associée à un défaut de perméabilité intestinale, ce défaut conduit au cas de l'endotoxémie, qui est l'augmentation du niveau de lipopolysaccharides circulants provenant de la paroi des cellules bactériennes à Gram négatif. On constate que l'endotoxémie est associée au développement de la résistance à l'insuline.
De plus, la production de butyrate affecte le niveau de sérotonine. Un niveau élevé de sérotonine contribue à l'obésité, qui est connue pour être un facteur de risque de développement du diabète.
Le microbiote peut être transplanté dans le corps humain à des fins médicales.
Développement du microbiote intestinal et antibiotiques
La colonisation du microbiote intestinal humain peut commencer déjà avant la naissance. Il existe de multiples facteurs environnementaux qui affectent le développement du microbiote, le mode de naissance étant l'un des plus impactants.
Un autre facteur qui a été observé comme provoquant d'énormes changements dans le microbiote intestinal, en particulier chez les enfants, est l'utilisation d'antibiotiques, associée à des problèmes de santé tels qu'un IMC plus élevé et un risque accru de maladies métaboliques telles que l'obésité. Chez les nourrissons, il a été observé que l'amoxicilline et les macrolides provoquent des changements importants dans le microbiote intestinal caractérisés par un changement dans les classes bactériennes Bifidobacteria, Enterobacteria et Clostridia. Une seule cure d'antibiotiques chez l'adulte provoque des changements dans le microbiote bactérien et fongique, avec des changements encore plus persistants dans les communautés fongiques. Les bactéries et les champignons vivent ensemble dans l'intestin et il y a très probablement une compétition pour les sources de nutriments présentes. Seelbinder et al . ont découvert que les bactéries commensales de l'intestin régulent la croissance et la pathogénicité de Candida albicans par leurs métabolites, en particulier par le propionate, l'acide acétique et le 5-dodécénoate. Le candida a déjà été associé aux MICI et en outre, il a été observé qu'il augmente chez les non-répondeurs à un médicament biologique, l'infliximab, administré aux patients atteints de MII souffrant de MII sévère. Le propionate et l'acide acétique sont tous deux des acides gras à chaîne courte (AGCC) qui se sont avérés bénéfiques pour la santé du microbiote intestinal. Lorsque les antibiotiques affectent la croissance des bactéries dans l'intestin, il peut y avoir une prolifération de certains champignons, qui peuvent être pathogènes lorsqu'ils ne sont pas réglementés.
Problèmes de confidentialité
L'ADN microbien qui habite le corps humain d'une personne peut identifier de manière unique la personne. La vie privée d'une personne peut être compromise si la personne a donné anonymement des données d'ADN microbien. Leur état de santé et leur identité pourraient être révélés.
Voir également
Les références
| Scholia a un profil thématique pour le microbiome . |