Régulation de l'expression des gènes - Regulation of gene expression
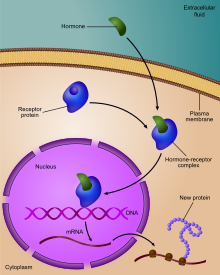
La régulation de l'expression génique , ou régulation génique , comprend un large éventail de mécanismes utilisés par les cellules pour augmenter ou diminuer la production de produits géniques spécifiques ( protéine ou ARN ). Des programmes sophistiqués d' expression génique sont largement observés en biologie, par exemple pour déclencher des voies de développement, répondre à des stimuli environnementaux ou s'adapter à de nouvelles sources de nourriture. Pratiquement n'importe quelle étape de l'expression génique peut être modulée, de l' initiation de la transcription au traitement de l'ARN et à la modification post-traductionnelle d'une protéine. Souvent, un régulateur génique en contrôle un autre, et ainsi de suite, dans un réseau de régulation génique .
La régulation des gènes est essentielle pour les virus , les procaryotes et les eucaryotes car elle augmente la polyvalence et l'adaptabilité d'un organisme en permettant à la cellule d'exprimer des protéines en cas de besoin. Bien que dès 1951, Barbara McClintock ait montré une interaction entre deux loci génétiques, l'activateur ( Ac ) et le dissociateur ( Ds ), dans la formation de la couleur des graines de maïs, la première découverte d'un système de régulation génique est largement considérée comme l'identification en 1961 de l' opéron lac , découvert par François Jacob et Jacques Monod , dans lequel certaines enzymes impliquées dans le métabolisme du lactose ne sont exprimées par E. coli qu'en présence de lactose et en absence de glucose.
Dans les organismes multicellulaires, la régulation génique entraîne la différenciation cellulaire et la morphogenèse dans l'embryon, conduisant à la création de différents types de cellules possédant des profils d'expression génique différents à partir de la même séquence génomique . Bien que cela n'explique pas l'origine de la régulation des gènes, les biologistes évolutionnistes l'incluent comme une explication partielle du fonctionnement de l' évolution au niveau moléculaire , et c'est au cœur de la science de la biologie du développement évolutif ("evo-devo").
Étapes régulées de l'expression des gènes
Toute étape d'expression génique peut être modulée, de l' étape de transcription ADN-ARN à la modification post-traductionnelle d'une protéine. Ce qui suit est une liste des étapes où l'expression des gènes est régulée, le point le plus largement utilisé est l'initiation de la transcription :
- Domaines de chromatine
- Transcription
- Modification post-transcriptionnelle
- Transport d'ARN
- Traduction
- dégradation de l'ARNm
Modification de l'ADN
Chez les eucaryotes, l'accessibilité de grandes régions d'ADN peut dépendre de la structure de sa chromatine , qui peut être altérée à la suite de modifications des histones dirigées par la méthylation de l'ADN , l' ARNnc ou la protéine de liaison à l'ADN . Par conséquent, ces modifications peuvent réguler à la hausse ou à la baisse l'expression d'un gène. Certaines de ces modifications qui régulent l'expression des gènes sont héréditaires et sont appelées régulation épigénétique .
De construction
La transcription de l'ADN est dictée par sa structure. En général, la densité de son tassement est révélatrice de la fréquence de transcription. Les complexes protéiques octamériques appelés histones ainsi qu'un segment d'ADN enroulé autour des huit protéines histones (appelés ensemble nucléosome) sont responsables de la quantité de superenroulement de l'ADN, et ces complexes peuvent être temporairement modifiés par des processus tels que la phosphorylation ou de manière plus permanente modifié par des processus tels que la méthylation . De telles modifications sont considérées comme responsables de changements plus ou moins permanents des niveaux d'expression des gènes.
Chimique
La méthylation de l'ADN est une méthode courante de silençage génique. L'ADN est typiquement méthylé par les enzymes méthyltransférase sur les nucléotides de la cytosine dans une séquence de dinucléotides CpG (également appelés « îlots CpG » lorsqu'ils sont densément regroupés). L'analyse du schéma de méthylation dans une région donnée de l'ADN (qui peut être un promoteur) peut être réalisée grâce à une méthode appelée cartographie au bisulfite. Les résidus cytosine méthylés ne sont pas modifiés par le traitement, tandis que les résidus non méthylés sont transformés en uracile. Les différences sont analysées par séquençage d'ADN ou par des méthodes développées pour quantifier les SNP, telles que le pyroséquençage ( Biotage ) ou le MassArray ( Sequenom ), mesurant les quantités relatives de C/T au niveau du dinucléotide CG. On pense que des modèles de méthylation anormaux sont impliqués dans l'oncogenèse.
L'acétylation des histones est également un processus important dans la transcription. Les enzymes histone acétyltransférase (HAT) telles que la protéine de liaison CREB dissocient également l'ADN du complexe histone, permettant ainsi à la transcription de se poursuivre. Souvent, la méthylation de l'ADN et la désacétylation des histones fonctionnent ensemble dans le silençage génique . La combinaison des deux semble être un signal pour que l'ADN soit plus dense, diminuant l'expression des gènes.
Règlement de la transcription

La régulation de la transcription contrôle donc le moment où la transcription se produit et la quantité d'ARN créée. La transcription d'un gène par l' ARN polymérase peut être régulée par plusieurs mécanismes. Les facteurs de spécificité modifient la spécificité de l'ARN polymérase pour un promoteur ou un ensemble de promoteurs donné, la rendant plus ou moins susceptible de s'y lier (c'est-à-dire les facteurs sigma utilisés dans la transcription procaryote ). Les répresseurs se lient à l' opérateur , codant des séquences sur le brin d'ADN qui sont proches ou chevauchant la région du promoteur, entravant la progression de l'ARN polymérase le long du brin, entravant ainsi l'expression du gène. L'image de droite montre la régulation par un répresseur dans l'opéron lac. Les facteurs de transcription généraux positionnent l'ARN polymérase au début d'une séquence codant pour une protéine, puis libèrent la polymérase pour transcrire l'ARNm. Les activateurs améliorent l'interaction entre l'ARN polymérase et un promoteur particulier , encourageant l'expression du gène. Les activateurs le font en augmentant l'attraction de l'ARN polymérase pour le promoteur, par le biais d'interactions avec des sous-unités de l'ARN polymérase ou indirectement en modifiant la structure de l'ADN. Les activateurs sont des sites sur l'hélice d'ADN qui sont liés par des activateurs afin de boucler l'ADN en apportant un promoteur spécifique au complexe d'initiation. Les activateurs sont beaucoup plus fréquents chez les eucaryotes que chez les procaryotes, où seuls quelques exemples existent (à ce jour). Les silencieux sont des régions de séquences d'ADN qui, lorsqu'elles sont liées par des facteurs de transcription particuliers, peuvent faire taire l'expression du gène.
Régulation de la transcription dans le cancer
Chez les vertébrés, la majorité des promoteurs de gènes contiennent un îlot CpG avec de nombreux sites CpG . Lorsque de nombreux sites CpG du promoteur d'un gène sont méthylés, le gène devient silencieux. Les cancers colorectaux ont généralement 3 à 6 mutations du conducteur et 33 à 66 mutations de l' auto - stoppeur ou du passager. Cependant, le silence transcriptionnel peut être plus important que la mutation dans la progression vers le cancer. Par exemple, dans les cancers colorectaux, environ 600 à 800 gènes sont réduits au silence transcriptionnellement par la méthylation des îlots CpG (voir régulation de la transcription dans le cancer ). La répression transcriptionnelle dans le cancer peut également se produire par d'autres mécanismes épigénétiques , tels que l'expression altérée des microARN . Dans le cancer du sein, la répression transcriptionnelle de BRCA1 peut survenir plus fréquemment par microARN-182 surexprimé que par hyperméthylation du promoteur BRCA1 (voir Faible expression de BRCA1 dans les cancers du sein et de l'ovaire ).
Régulation de la transcription dans l'addiction
L'une des caractéristiques cardinales de la toxicomanie est sa persistance. Les changements comportementaux persistants semblent être dus à des changements durables, résultant d' altérations épigénétiques affectant l'expression des gènes, dans des régions particulières du cerveau. Les drogues d'abus provoquent trois types d'altération épigénétique dans le cerveau. Ce sont (1) les acétylations et les méthylations des histones , (2) la méthylation de l'ADN au niveau des sites CpG et (3) la régulation négative ou positive épigénétique des microARN . (Voir Epigénétique de la dépendance à la cocaïne pour quelques détails.)
La consommation chronique de nicotine chez la souris altère le contrôle épigénétique des cellules cérébrales de l'expression des gènes par l' acétylation des histones . Cela augmente l'expression dans le cerveau de la protéine FosB, importante dans l'addiction. La dépendance à la cigarette a également été étudiée chez environ 16 000 humains, y compris des personnes n'ayant jamais fumé, des fumeurs actuels et des personnes ayant cessé de fumer depuis jusqu'à 30 ans. Dans les cellules sanguines, plus de 18 000 sites CpG (sur les quelque 450 000 sites CpG analysés dans le génome) avaient fréquemment modifié la méthylation chez les fumeurs actuels. Ces sites CpG sont présents dans plus de 7 000 gènes, soit environ un tiers des gènes humains connus. La majorité des sites CpG à méthylation différentielle sont revenus au niveau des non-fumeurs dans les cinq ans suivant l'arrêt du tabac. Cependant, 2 568 CpG parmi 942 gènes sont restés méthylés de manière différentielle chez les anciens par rapport aux non-fumeurs. Ces changements épigénétiques restants peuvent être considérés comme des « cicatrices moléculaires » qui peuvent affecter l'expression des gènes.
Dans les modèles de rongeurs, les drogues d'abus, y compris la cocaïne, la méthamphétamine, l'alcool et les produits de la fumée de tabac, causent toutes des dommages à l'ADN dans le cerveau. Au cours de la réparation des dommages à l'ADN, certains événements de réparation individuels peuvent altérer la méthylation de l'ADN et/ou les acétylations ou méthylations des histones sur les sites de dommages, et peuvent ainsi contribuer à laisser une cicatrice épigénétique sur la chromatine.
De telles cicatrices épigénétiques contribuent probablement aux changements épigénétiques persistants observés dans la toxicomanie.
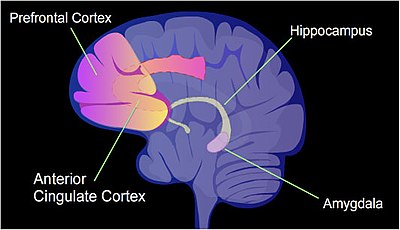
Régulation de la transcription dans l'apprentissage et la mémoire

Chez les mammifères, la méthylation de la cytosine (voir Figure) dans l'ADN est un médiateur régulateur majeur. Les cytosines méthylées se produisent principalement dans les séquences dinucléotidiques où la cytosine est suivie d'une guanine, un site CpG . Le nombre total de sites CpG dans le génome humain est d'environ 28 millions. et généralement environ 70 % de tous les sites CpG ont une cytosine méthylée.
Chez un rat, une expérience d'apprentissage douloureuse, le conditionnement de la peur contextuelle , peut entraîner un souvenir effrayant à vie après un seul événement d'entraînement. La méthylation de la cytosine est altérée dans les régions promotrices d'environ 9,17 % de tous les gènes de l'ADN des neurones de l'hippocampe d'un rat qui a été soumis à une brève expérience de conditionnement de la peur . L' hippocampe est l'endroit où les nouveaux souvenirs sont initialement stockés.
La méthylation des CpG dans une région promotrice d'un gène réprime la transcription tandis que la méthylation des CpG dans le corps d'un gène augmente l'expression. Les enzymes TET jouent un rôle central dans la déméthylation des cytosines méthylées. La déméthylation des CpG dans un promoteur de gène par l' activité enzymatique TET augmente la transcription du gène.
Lorsque le conditionnement contextuel de la peur est appliqué à un rat, plus de 5 000 régions à méthylation différentielle (DMR) (de 500 nucléotides chacune) se produisent dans le génome neuronal de l' hippocampe du rat une heure et 24 heures après le conditionnement dans l'hippocampe. Cela provoque une régulation positive d'environ 500 gènes (souvent en raison de la déméthylation des sites CpG dans une région promotrice) et une régulation négative d'environ 1 000 gènes (souvent en raison de la 5-méthylcytosine nouvellement formée sur les sites CpG dans une région promotrice). Le schéma des gènes induits et réprimés dans les neurones semble fournir une base moléculaire pour former le premier souvenir transitoire de cet événement d'entraînement dans l'hippocampe du cerveau de rat.
Régulation post-transcriptionnelle
Une fois l'ADN transcrit et l'ARNm formé, il doit y avoir une sorte de régulation sur la quantité d'ARNm traduit en protéines. Les cellules le font en modulant le coiffage, l'épissage, l'ajout d'une queue Poly (A), les taux d'exportation nucléaire spécifiques à la séquence et, dans plusieurs contextes, la séquestration du transcrit d'ARN. Ces processus se produisent chez les eucaryotes mais pas chez les procaryotes. Cette modulation est le résultat d'une protéine ou d'un transcrit qui, à son tour, est régulé et peut avoir une affinité pour certaines séquences.
Trois régions principales non traduites et microARN
Trois régions principales non traduites (3'-UTR) des ARN messagers (ARNm) contiennent souvent des séquences régulatrices qui influencent de manière post-transcriptionnelle l'expression des gènes. Ces 3'-UTR contiennent souvent à la fois des sites de liaison pour les microARN (miARN) ainsi que pour les protéines régulatrices. En se liant à des sites spécifiques dans la 3'-UTR, les miARN peuvent diminuer l'expression génique de divers ARNm en inhibant la traduction ou en provoquant directement la dégradation du transcrit. Le 3'-UTR peut également avoir des régions de silence qui se lient à des protéines répresseurs qui inhibent l'expression d'un ARNm.
Le 3'-UTR contient souvent des éléments de réponse miARN (MRE) . Les MRE sont des séquences auxquelles les miARN se lient. Ce sont des motifs répandus dans les 3'-UTR. Parmi tous les motifs régulateurs au sein des 3'-UTR (par exemple, y compris les régions de silencieux), les MRE représentent environ la moitié des motifs.
En 2014, le site Web miRBase , une archive de séquences et d'annotations de miARN , répertoriait 28 645 entrées dans 233 espèces biologiques. Parmi ceux-ci, 1 881 miARN se trouvaient dans des loci de miARN humains annotés. Les miARN devaient avoir en moyenne environ quatre cents ARNm cibles (affectant l'expression de plusieurs centaines de gènes). Freidman et al. estiment que > 45 000 sites cibles de miARN dans les 3'-UTR d'ARNm humains sont conservés au-dessus des niveaux de fond, et > 60 % des gènes codant pour des protéines humaines ont été soumis à une pression sélective pour maintenir l'appariement aux miARN.
Des expériences directes montrent qu'un seul miARN peut réduire la stabilité de centaines d'ARNm uniques. D'autres expériences montrent qu'un seul miARN peut réprimer la production de centaines de protéines, mais que cette répression est souvent relativement légère (moins de 2 fois).
Les effets de la dérégulation de l'expression des gènes par les miARN semblent être importants dans le cancer. Par exemple, dans les cancers gastro-intestinaux, un article de 2015 a identifié neuf miARN comme étant épigénétiquement modifiés et efficaces pour réguler à la baisse les enzymes de réparation de l'ADN.
Les effets du miARN dysrégulation de l' expression génétique semble également être important dans les troubles neuropsychiatriques, tels que la schizophrénie , le trouble bipolaire , le trouble dépressif majeur , la maladie de Parkinson , la maladie d'Alzheimer et de spectre de l' autisme troubles.
Règlement de la traduction
La traduction de l'ARNm peut également être contrôlée par un certain nombre de mécanismes, principalement au niveau de l'initiation. Le recrutement de la petite sous-unité ribosomique peut en effet être modulé par la structure secondaire de l'ARNm, la liaison à l'ARN antisens ou la liaison aux protéines. Chez les procaryotes comme chez les eucaryotes, il existe un grand nombre de protéines de liaison à l'ARN, qui sont souvent dirigées vers leur séquence cible par la structure secondaire du transcrit, qui peut changer en fonction de certaines conditions, telles que la température ou la présence d'un ligand (aptamère) . Certains transcrits agissent comme des ribozymes et autorégulent leur expression.
Exemples de régulation génique
- L'induction enzymatique est un processus dans lequel une molécule (par exemple, un médicament) induit (c'est-à-dire initie ou augmente) l'expression d'une enzyme.
- L'induction de protéines de choc thermique chez la mouche des fruits Drosophila melanogaster .
- L' opéron Lac est un exemple intéressant de la façon dont l'expression des gènes peut être régulée.
- Les virus, bien qu'ils n'aient que quelques gènes, possèdent des mécanismes pour réguler leur expression génique, généralement dans une phase précoce et tardive, en utilisant des systèmes colinéaires régulés par des anti-terminateurs ( phage lambda ) ou des modulateurs d'épissage ( VIH ).
- Gal4 est un activateur transcriptionnel qui contrôle l'expression de GAL1, GAL7 et GAL10 (qui codent tous pour le métabolisme du galactose chez la levure). Le système GAL4/UAS a été utilisé dans une variété d'organismes dans divers phylums pour étudier l'expression des gènes.
Biologie du développement
Un grand nombre de systèmes de régulation étudiés sont issus de la biologie du développement . Les exemples comprennent:
- La colinéarité du groupe de gènes Hox avec leur structuration antéro-postérieure imbriquée
- Génération de motifs de la main (chiffres - interchiffres) : le gradient de sonic hedgehog (facteur inducteur sécrété) à partir de la zone d'activité polarisante dans le membre, qui crée un gradient de Gli3 active, qui active Gremlin, qui inhibe les BMP également sécrétées dans le membre, entraîne la formation d'un schéma d'activité alterné à la suite de ce système de réaction-diffusion .
- Somitogenèse est la création de segments (somites) d'un tissu uniforme (Pre-somitique mésoderme ). Ils sont formés séquentiellement d'avant en arrière. Ceci est réalisé dans les amniotes éventuellement au moyen de deux gradients opposés, l'acide rétinoïque dans la partie antérieure (front d'onde) et Wnt et Fgf dans la partie postérieure, couplés à un motif oscillant (horloge de segmentation) composé de FGF + Notch et Wnt en antiphase.
- La détermination du sexe dans le soma d'une drosophile nécessite la détection du rapport des gènes autosomiques aux gènes codés par les chromosomes sexuels, ce qui entraîne la production d'un facteur d'épissage sans sexe chez les femelles, ce qui donne l'isoforme féminine du double sexe.
Circuits
Régulation à la hausse et régulation à la baisse
La régulation à la hausse est un processus qui se produit dans une cellule déclenché par un signal (provenant de l'intérieur ou de l'extérieur de la cellule), qui entraîne une expression accrue d'un ou plusieurs gènes et, par conséquent, des protéines codées par ces gènes. Inversement, la régulation négative est un processus entraînant une diminution de l'expression des gènes et des protéines correspondantes.
- La régulation à la hausse se produit, par exemple, lorsqu'une cellule est déficiente en un certain type de récepteur. Dans ce cas, davantage de protéines réceptrices sont synthétisées et transportées vers la membrane de la cellule et, ainsi, la sensibilité de la cellule est ramenée à la normale, rétablissant l' homéostasie .
- Une régulation à la baisse se produit, par exemple, lorsqu'une cellule est surstimulée par un neurotransmetteur , une hormone ou un médicament pendant une période prolongée et que l'expression de la protéine réceptrice est diminuée afin de protéger la cellule (voir aussi tachyphylaxie ).
Systèmes inductibles vs. répressibles
La régulation des gènes peut être résumée par la réponse du système respectif :
- Systèmes inductibles - Un système inductible est désactivé à moins qu'il n'y ait la présence d'une molécule (appelée inducteur) qui permet l'expression génique. La molécule est dite "induire l'expression". La manière dont cela se produit dépend des mécanismes de contrôle ainsi que des différences entre les cellules procaryotes et eucaryotes.
- Systèmes répressibles - Un système répressible est activé sauf en présence d'une molécule (appelée corépresseur) qui supprime l'expression des gènes. On dit que la molécule « réprime l'expression ». La manière dont cela se produit dépend des mécanismes de contrôle ainsi que des différences entre les cellules procaryotes et eucaryotes.
Le système GAL4/UAS est un exemple à la fois de système inductible et répressible. Gal4 se lie à une séquence d'activation en amont (UAS) pour activer la transcription de la cassette GAL1/GAL7/GAL10. En revanche, une réponse MIG1 à la présence de glucose peut inhiber GAL4 et donc stopper l'expression de la cassette GAL1/GAL7/GAL10.
Circuits théoriques
- Répresseur/Inducteur : une activation d'un capteur entraîne le changement d'expression d'un gène
- rétroaction négative : le produit du gène régule à la baisse sa propre production directement ou indirectement, ce qui peut entraîner
- maintenir les niveaux de transcription constants/proportionnels à un facteur
- inhibition des réactions d'emballement lorsqu'il est couplé à une boucle de rétroaction positive
- créer un oscillateur en profitant du délai de transcription et de traduction, étant donné que la demi-vie de l'ARNm et de la protéine est plus courte
- rétroaction positive : le produit du gène régule à la hausse sa propre production directement ou indirectement, ce qui peut entraîner
- amplification du signal
- commutateurs bistables lorsque deux gènes s'inhibent et que les deux ont une rétroaction positive
- génération de modèle
Méthodes d'étude
En général, la plupart des expériences portant sur l'expression différentielle ont utilisé des extraits de cellules entières d'ARN, appelés niveaux à l'état d'équilibre, pour déterminer quels gènes ont changé et dans quelle mesure. Celles-ci ne renseignent cependant pas sur l'endroit où la régulation s'est produite et peuvent masquer des processus de régulation conflictuels ( voir régulation post-transcriptionnelle ), mais c'est toujours la plus couramment analysée ( PCR quantitative et puce à ADN ).
Lorsqu'on étudie l'expression des gènes, il existe plusieurs méthodes pour examiner les différentes étapes. Chez les eucaryotes, il s'agit de :
- L'environnement local de la chromatine de la région peut être déterminé par l' analyse de la puce ChIP en tirant vers le bas l' ARN polymérase II , les modifications de l' Histone 3 , la protéine du groupe Trithorax , la protéine du groupe Polycomb ou tout autre élément de liaison à l'ADN pour lequel un bon anticorps est disponible .
- Les interactions épistatiques peuvent être étudiées par analyse génétique synthétique
- En raison de la régulation post-transcriptionnelle, les taux de transcription et les niveaux d'ARN total diffèrent considérablement. Pour mesurer les taux de transcription, des tests nucléaires peuvent être effectués et de nouvelles méthodes à haut débit sont en cours de développement, utilisant le marquage au thiol au lieu de la radioactivité .
- Seuls 5% de l'ARN polymérisé dans le noyau sort, et pas seulement les introns, les produits abortifs et les transcrits non-sens sont dégradés. Par conséquent, les différences de niveaux nucléaires et cytoplasmiques peuvent être observées en séparant les deux fractions par une lyse douce.
- L'épissage alternatif peut être analysé avec une matrice d'épissage ou avec une matrice de tuilage ( voir puce à ADN ).
- Tout l' ARN in vivo est complexé sous forme de RNP . La quantité de transcrits liés à une protéine spécifique peut également être analysée par RIP-Chip . Par exemple, DCP2 donnera une indication de protéine séquestrée ; Les données liées au ribosome et l'indication des transcrits actifs dans la transcription (bien qu'une méthode plus ancienne, appelée fractionnement des polysomes , soit toujours populaire dans certains laboratoires)
- Les niveaux de protéines peuvent être analysés par spectrométrie de masse , qui ne peut être comparée qu'à des données PCR quantitatives , car les données de microarray sont relatives et non absolues.
- Les taux de dégradation de l'ARN et des protéines sont mesurés au moyen d'inhibiteurs de transcription ( actinomycine D ou -amanitine ) ou d'inhibiteurs de traduction ( cycloheximide ), respectivement.
Voir également
- Facteurs de transcription artificiels (petites molécules qui imitent la protéine du facteur de transcription)
- Modèle cellulaire
- Séquence d'ADN non codante conservée
- Enhancer (génétique)
- Structure des gènes
- Expression génique spatiotemporelle
Notes et références
Bibliographie
- Latchman, David S. (2005). Régulation des gènes : une perspective eucaryote . Presse de psychologie. ISBN 978-0-415-36510-9.
Liens externes
- Base de données des facteurs de transcription des plantes et plateforme de données et d'analyse de la régulation de la transcription des plantes
- Régulation de l'expression génique (MeSH) à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis
- ChIPBase Une base de données ouverte pour décoder les réseaux de régulation transcriptionnelle des ARN non codants et des gènes codant pour les protéines à partir des données ChIP-seq.