ADN ribosomique - Ribosomal DNA

L'ADN ribosomique ( ADNr ) est une séquence d' ADN qui code pour l'ARN ribosomique . Ces séquences régulent la transcription initiation et d' amplification, et contiennent à la fois une transcription et non transcrite écartement des segments. L'ARNr transcrit à partir d'environ 600 répétitions d'ADN r forme la section d'ARN la plus abondante trouvée dans les cellules des eucaryotes . Les ribosomes sont des assemblages de protéines et de molécules d'ARNr qui traduisent les molécules d' ARNm pour produire des protéines. Comme le montre la figure, l'ADNr des eucaryotes se compose d'une répétition en tandem d'un segment unitaire, composé des faisceaux NTS, ETS, 18S , ITS1 , 5.8S , ITS2 et 28S . L'ADNr possède un autre gène, codant pour l' ARNr 5S , situé dans le génome de la plupart des eucaryotes. L'ADNr 5S est également présent dans des répétitions en tandem comme chez la drosophile . Les régions d'ADN qui sont répétitives subissent souvent des événements de recombinaison. Les répétitions d'ADNr ont de nombreux mécanismes de régulation qui empêchent l'ADN de subir des mutations, gardant ainsi l'ADNr conservé.
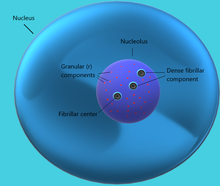
Dans le noyau, la région ADNr du chromosome est visualisée sous la forme d'un nucléole qui forme des boucles chromosomiques étendues avec l'ADNr. Les unités transcriptionnelles d'ARNr sont regroupées en répétitions en tandem. Ces régions d'ADNr sont également appelées régions organisatrices du nucléole , car elles donnent naissance au nucléole. Dans l'ADNr, les répétitions en tandem se trouvent principalement dans le nucléole ; mais l'ADNr hétérochromatique se trouve à l'extérieur du nucléole. Cependant, l'ADNr transcriptionnellement actif réside à l'intérieur du nucléole lui-même.
Dans le génome humain, il existe 5 chromosomes avec des régions organisatrices du nucléole : les chromosomes acrocentriques 13 ( RNR1 ), 14 ( RNR2 ), 15 ( RNR3 ), 21 ( RNR4 ) et 22 ( RNR5 ). Les gènes responsables du codage des différentes sous-unités de l'ARNr sont situés sur plusieurs chromosomes chez l'homme. Mais les gènes qui codent pour l'ARNr sont hautement conservés dans les domaines, seuls les nombres de copies impliqués pour les gènes ayant des nombres variables par espèce. Dans les bactéries , les archées et les chloroplastes, l'ARNr est composé de différentes unités (plus petites), le grand ARN ribosomique (23S) , l' ARN ribosomique 16S et l'ARNr 5S. L'ARNr 16S est largement utilisé pour les études phylogénétiques .
Homogénéité de la séquence
Dans le grand réseau d'ADNr, les polymorphismes entre les unités répétées d'ADNr sont très faibles, ce qui indique que les réseaux en tandem d'ADNr évoluent grâce à une évolution concertée . Cependant, le mécanisme de l'évolution concertée est imparfait, de sorte que les polymorphismes entre les répétitions au sein d'un individu peuvent se produire à des niveaux significatifs et peuvent confondre les analyses phylogénétiques pour les organismes étroitement liés.
Les séquences répétées en tandem 5S chez plusieurs drosophiles ont été comparées entre elles ; le résultat a révélé que les insertions et les suppressions se produisaient fréquemment entre les espèces et souvent flanquées de séquences conservées. Ils pourraient se produire par glissement du brin nouvellement synthétisé lors de la réplication de l'ADN ou par conversion génique.
Divergence de séquence
Les tracts de transcription de l'ADNr ont un faible taux de polymorphisme parmi les espèces, ce qui permet une comparaison interspécifique pour élucider la relation phylogénétique en utilisant seulement quelques spécimens. Les régions codantes de l'ADNr sont hautement conservées parmi les espèces, mais les régions ITS sont variables en raison des insertions, des suppressions et des mutations ponctuelles. Entre les espèces éloignées comme l'homme et la grenouille, la comparaison des séquences dans les secteurs ITS n'est pas appropriée. Les séquences conservées au niveau des régions codantes de l'ADNr permettent des comparaisons d'espèces éloignées, même entre la levure et l'homme. L'ARNr 5.8S humain a 75 % d'identité avec l'ARNr 5.8S de levure. Dans les cas d'espèces jumelles, la comparaison du segment d'ADNr, y compris les tracts ITS entre les espèces et l'analyse phylogénétique sont effectuées de manière satisfaisante. Les différentes régions codantes des répétitions d'ADNr présentent généralement des taux d'évolution distincts. En conséquence, cet ADN peut fournir des informations phylogénétiques d'espèces appartenant à de larges niveaux systématiques.
Activité stimulant la recombinaison
Un fragment d'ADNr de levure contenant le gène 5S, un ADN espaceur non transcrit et une partie du gène 35S a une activité de stimulation de la recombinaison mitotique localisée à action cis . Ce fragment d'ADN contient un point chaud de recombinaison mitotique , appelé HOT1. HOT1 exprime une activité stimulant la recombinaison lorsqu'il est inséré dans de nouveaux emplacements dans le génome de la levure . HOT1 comprend un promoteur de transcription de l' ARN polymérase I (PolI) qui catalyse la transcription du gène de l' ARNr ribosomique 35S . Chez un mutant défectif PolI, l'activité de stimulation de la recombinaison du hotspot HOT1 est abolie. Le niveau de transcription PolI dans HOT1 semble déterminer le niveau de recombinaison .
Signification clinique
Les maladies peuvent être associées à des mutations de l'ADN où l'ADN peut être développé, comme la maladie de Huntington , ou perdu en raison de mutations par délétion. La même chose est vraie pour les mutations qui se produisent dans les répétitions d'ADNr ; il a été découvert que si les gènes associés à la synthèse des ribosomes sont perturbés ou mutés, cela peut entraîner diverses maladies associées au squelette ou à la moelle osseuse. En outre, tout dommage ou perturbation des enzymes qui protègent les répétitions en tandem de l'ADNr peut entraîner une synthèse plus faible des ribosomes, ce qui entraîne également d'autres défauts dans la cellule. Les maladies neurologiques peuvent également résulter de mutations dans les répétitions en tandem de l'ADNr, telles que le syndrome de Bloom , qui survient lorsque le nombre de répétitions en tandem augmente de près de cent fois; par rapport au nombre normal de répétitions en tandem. Divers types de cancers peuvent également naître de mutations des répétitions en tandem dans l'ADN ribosomique. Les lignées cellulaires peuvent devenir malignes à la suite d'un réarrangement des répétitions en tandem ou d'une expansion des répétitions dans l'ADNr.