ADNG - DnaG
| ADN primase | |||||||
|---|---|---|---|---|---|---|---|
| Identifiants | |||||||
| Organisme | |||||||
| symbole | adn | ||||||
| Alt. symboles | adn | ||||||
| Entrez | 947570 | ||||||
| APD | 1D0Q, 1DD9, 1DDE, 1EQ9, 2R6A, 2R6C | ||||||
| RefSeq (Prot) | NP_417538 | ||||||
| UniProt | P0ABS5 | ||||||
| Autre informations | |||||||
| Numéro CE | 2.7.7.7 | ||||||
| Chromosome | chromosome : 3,21 - 3,21 Mo | ||||||
| |||||||
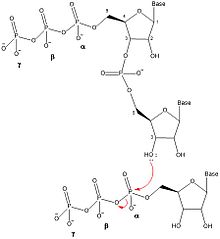
DnaG est un ADN primase bactérien et est codé par le gène dnaG . L'enzyme DnaG, et toute autre ADN primase, synthétise de courts brins d' ARN appelés oligonucléotides lors de la réplication de l'ADN . Ces oligonucléotides sont appelés amorces car ils servent de point de départ à la synthèse d'ADN. L'ADNG catalyse la synthèse d'oligonucléotides de 10 à 60 nucléotides (l'unité fondamentale de l'ADN et de l'ARN) de long, cependant la plupart des oligonucléotides synthétisés sont de 11 nucléotides. Ces oligonucléotides d'ARN servent d'amorces, ou de points de départ, pour la synthèse d'ADN par l' ADN polymérase III bactérienne (Pol III). L'ADNG est important dans la réplication de l'ADN bactérien car l'ADN polymérase ne peut pas initier la synthèse d'un brin d'ADN, mais ne peut qu'ajouter des nucléotides à un brin préexistant. DnaG synthétise une seule amorce d'ARN à l' origine de la réplication . Cette amorce sert à amorcer la synthèse d'ADN du brin principal . Pour l'autre brin parental, le brin retardé, DnaG synthétise une amorce d'ARN toutes les quelques kilobases (kb). Ces amorces servent de substrats pour la synthèse de fragments d'Okazaki .
Dans E. coli, DnaG s'associe par le biais d'interactions non covalentes avec l' hélicase réplicative bactérienne DnaB pour exercer son activité primase, avec trois protéines primases DnaG s'associant à chaque hélicase DnaB pour former le primosome . Les primases ont tendance à initier la synthèse au niveau de trois séquences nucléotidiques spécifiques sur des matrices d'ADN simple brin (ADNsb) et pour E. coli DnaG, la séquence est 5'-CTG-3'.
DnaG contient trois domaines protéiques distincts : un domaine de liaison au zinc, un domaine ARN polymérase et un domaine de liaison hélicase DnaB. Il existe plusieurs bactéries qui utilisent l'ADN primase DnaG. Quelques organismes qui ont DnaG comme ADN primase sont Escherichia coli ( E. coli ), Bacillus stearothermophilus et Mycobacterium tuberculosis (MTB). E. coli DnaG a un poids moléculaire de 60 kilodaltons (kDa) et contient 581 acides aminés .
Une fonction
L'ADNG catalyse la synthèse d'oligonucléotides en cinq étapes distinctes : liaison à la matrice, liaison au nucléoside triphosphate (NTP), initiation, extension pour former une amorce et transfert d'amorce à l'ADN polymérase III. DnaG effectue cette catalyse à proximité de la fourche de réplication formée par l'hélicase DnaB lors de la réplication de l'ADN. Le DnaG doit être complexé avec le DnaB pour qu'il catalyse la formation des amorces oligonucléotidiques.
Le mécanisme de synthèse des amorces par les primases implique deux sites de liaison NTP sur la protéine primase (DnaG). Avant la liaison de tout NTP pour former l'amorce d'ARN, la séquence matrice d'ADNsb se lie à l'ADNG. L'ADNsb contient une séquence de reconnaissance à trois nucléotides qui recrute les NTP sur la base de l'appariement de bases Watson-Crick . Après avoir lié l'ADN, DnaG doit lier deux NTP afin de générer un complexe quaternaire enzyme-ADN-NTP-NTP. La constante de Michaelis (km) pour les NTP varie en fonction de la primase et des modèles. Les deux sites de liaison NTP sur DnaG sont appelés site d'initiation et site d'élongation. Le site d'initiation est le site auquel la NTP à incorporer à l'extrémité 5' de l'amorce se lie. Le site d'élongation lie le NTP qui est ajouté à l'extrémité 3' de l'amorce.
Une fois que deux nucléotides sont liés à la primase, le DnaG catalyse la formation d'un dinucléotide en formant une liaison phosphodiester via la synthèse de déshydratation entre le 3' hydroxyle du nucléotide dans le site d'initiation et le -phosphate du nucléotide dans le site d'élongation. Cette réaction se traduit par un dinucléotide et une rupture de la liaison entre le phosphore et β, libérant du pyrophosphate. Cette réaction est irréversible car le pyrophosphate qui se forme est hydrolysé en deux molécules de phosphate inorganique par l'enzyme pyrophosphatase inorganique . Cette réaction de synthèse de dinucléotide est la même réaction que toute autre enzyme qui catalyse la formation d'ADN ou d'ARN ( ADN polymérase , ARN polymérase ), donc DnaG doit toujours synthétiser des oligonucléotides dans le sens 5' à 3'. Dans E. coli , les amorces commencent par un dinucléotide triphosphate adénine-guanine (pppAG) à l'extrémité 5'.
Pour qu'un allongement supplémentaire du dinucléotide se produise, l'oligonucléotide doit être déplacé de sorte que le 3' NTP soit transféré du site d'élongation au site d'initiation, permettant à un autre NTP de se lier au site d'élongation et de se fixer au 3' hydroxyle de l'oligonucléotide. Une fois qu'un oligonucléotide de longueur appropriée a été synthétisé à partir de l'étape d'élongation de la synthèse de l'amorce, DnaG transfère l'amorce nouvellement synthétisée à l'ADN polymérase III pour qu'elle synthétise le brin principal d'ADN ou les fragments d'Okazaki pour le brin retardé. L'étape limitant la vitesse de la synthèse de l'amorce se produit après la liaison NTP mais avant ou pendant la synthèse des dinucléotides.
Structure
La primase E. Coli DnaG est une protéine monomère de 581 résidus avec trois domaines fonctionnels, selon des études de protéolyse. Il existe un domaine de liaison au zinc N-terminal (résidus 1-110) où un ion zinc est coordonné de manière tétraédrique entre un résidu histidine et trois résidus cystéine, qui joue un rôle dans la reconnaissance des sites de liaison à l'ADN spécifiques de la séquence. Le domaine central (résidus 111-433) présente des activités d'ARN polymérase et est le site de synthèse d'amorces d'ARN. Le domaine C-terminal (résidus 434-581) est responsable de la liaison non covalente de DnaG à la protéine hélicase DnaB .
Domaine de liaison au zinc

Le domaine de liaison au zinc, le domaine responsable de la reconnaissance des sites de liaison à l'ADN spécifiques à une séquence, est conservé dans toutes les primases d'ADN virales, bactériophages, procaryotes et eucaryotes. Le domaine de liaison au zinc de la primase fait partie de la sous-famille des domaines de liaison au zinc connus sous le nom de ruban de zinc . Les domaines de ruban de zinc sont caractérisés par deux boucles en épingle à cheveux qui forment le domaine de liaison au zinc. En règle générale, on pense que les domaines de ruban de zinc manquent d' hélices , ce qui les distingue des autres domaines de liaison au zinc. Cependant, en 2000 , le domaine de liaison de zinc de DnaG a été cristallisé à partir de Bacillus stearothermophilus révélant que le nom de domaine est composée de cinq brins antiparallèles feuille de β adjacentes à quatre hélices a et un 3 10 hélice à l'extrémité C-terminale du domaine.
Le site de liaison au zinc de B. stearothermophilus se compose de trois résidus cystéine, Cys40, Cys61 et Cys64, et d'un résidu histidine, His43. Cys40 et His43 sont situés sur l'épingle entre la deuxième et la troisième feuille β. Cys61 est situé sur la cinquième feuille et Cys64 est sur l'épingle entre la quatrième et la cinquième feuille β. Ces quatre résidus coordonnent l'ion zinc de manière tétraédrique. On pense que l'ion zinc stabilise les boucles entre les deuxième et troisième feuillets ainsi que les quatrième et cinquième feuillets . Le domaine est en outre stabilisé par un certain nombre d'interactions hydrophobes entre la surface interne hydrophobe de la feuille qui est tassée contre les deuxième et troisième hélices . La surface externe de la feuille a également de nombreux résidus hydrophobes et basiques conservés. Ces résidus sont Lys30, Arg34, Lys46, Pro48, Lys56, Ile58, His60 et Phe62.
Liaison à l'ADN
On pense que la fonction du domaine de liaison au zinc est pour la reconnaissance d'ADN spécifique à une séquence. Les primases d'ADN fabriquent des amorces d'ARN qui sont ensuite utilisées pour la synthèse d'ADN. Le placement des amorces d'ARN n'est pas aléatoire, ce qui suggère qu'elles sont placées sur des séquences d'ADN spécifiques. En effet, il a été démontré que d'autres primases à ADN reconnaissent des séquences triplées ; la séquence spécifique reconnue par B. stearothermophilus n'a pas encore été identifiée. Il a été démontré que si les résidus cystine qui coordonnent l'ion zinc sont mutés, l'ADN primase cesse de fonctionner. Cela indique que le domaine de liaison au zinc joue un rôle dans la reconnaissance de séquence. De plus, la surface hydrophobe de la feuille β, ainsi que les résidus basiques qui sont regroupés principalement sur un bord de la feuille, servent à attirer l'ADN simple brin, facilitant encore la liaison à l'ADN.
Sur la base d'études antérieures sur la liaison à l'ADN par les DNA Primases, on pense que l'ADN se lie au domaine de liaison au zinc à travers la surface de la feuille , les trois nucléotides se liant à travers trois brins de la feuille . Les résidus chargés positivement dans la feuille pourraient former des contacts avec les phosphates et les résidus aromatiques formeraient des interactions d'empilement avec les bases. C'est le modèle de liaison à l'ADN par le domaine de liaison à l'ADNsb de la protéine de réplication A (RPA) . Il est logique de supposer que le domaine de liaison au zinc de B. stearothermophilus se lie à l'ADN d'une manière similaire, car les résidus importants pour la liaison à l'ADN dans la RPA se trouvent dans des positions structurellement équivalentes dans B. stearothermophilus .
Domaine de l'ARN polymérase

Comme son nom l'indique, le domaine ARN polymérase (RNAP) de DnaG est responsable de la synthèse des amorces d'ARN sur l'ADN simple brin. In-vivo, DnaG est capable de synthétiser des fragments d'amorces jusqu'à 60 nucléotides, mais les fragments d'amorces in-vivo sont limités à environ 11 nucléotides. Au cours de la synthèse du brin retardé, le DnaG synthétise entre 2000 et 3000 amorces à raison d'une amorce par seconde.
Le domaine RNAP de DnaG a trois sous-domaines, le domaine N-terminal, qui a un pli mixte α et , le domaine central constitué d'un feuillet β à 5 brins et 6 hélices α, et enfin le domaine C-terminal qui est composé de un faisceau hélicoïdal constitué de 3 hélices α antiparallèles. Le domaine central est constitué en partie du pli toprim , un pli qui a été observé dans de nombreuses protéines de phosphotransfert liant les métaux. Le domaine central et le domaine N-terminal forment une fente peu profonde, qui constitue le site actif de l'allongement de la chaîne d'ARN dans DnaG. L'ouverture de la fente est bordée par plusieurs résidus basiques hautement conservés : Arg146, Arg221 et Lys229. Ces résidus font partie de la crête électrostatiquement positive du sous-domaine N-terminal. C'est cette crête qui interagit avec l'ADNsb et aide à le guider dans la fente, qui se compose du centre de liaison métallique du motif toprim sur le sous-domaine central et des motifs primase conservés du domaine N-terminal. Le site de liaison métallique du domaine toprim est l'endroit où l'amorce est synthétisée. Le duplex ARN:ADN sort ensuite par une autre dépression basique.
Domaine C-Terminal

Contrairement aux domaines de liaison au zinc et aux domaines d'ARN polymérase, les domaines C-terminaux des ADN primases ne sont pas conservés. Chez les primases procaryotes, la seule fonction connue de ce domaine est d'interagir avec l'hélicase, DnaB. Ainsi, ce domaine est appelé domaine de liaison à l'hélicase (HBD). Le HBD de DnaG se compose de deux sous-domaines : un faisceau hélicoïdal , le sous-domaine C1, et une épingle à cheveux hélicoïdale, le sous-domaine C2. Pour chacune des deux à trois molécules DnaG qui se lient à l'hexamère DnaB, les sous-domaines C1 des HBD interagissent avec DnaB au niveau de ses domaines N-terminaux sur la surface interne de l'anneau hexamère, tandis que les sous-domaines C2 interagissent avec les domaines N-terminaux sur la surface externe de l'hexamère.

Trois résidus dans B. stearothermophilus DnaB ont été identifiés comme importants pour la formation de l'interface DnaB, DnaG. Ces résidus comprennent Tyr88, Ile119 et Ile125. Tyr88 est à proximité, mais n'entre pas en contact avec, le HBD de DnaG. La mutation de Tyr88 inhibe la formation du faisceau hélicoïdal du domaine N-terminal de DnaB, interrompant les contacts avec le HBD de DnaG. La structure hexamérique de l'ADNB est en réalité un trimère de dimères. Ile119 et Ile125 sont tous deux enfouis dans l'interface dimère du domaine N-terminal de DnaB et la mutation de ces résidus inhibe la formation de la structure hexamère et donc l'interaction avec le DnaG. Un autre résidu qui a été identifié comme jouant un rôle crucial dans l'interaction de DnaB et DnaG est Glu15. La mutation de Glu15 ne perturbe pas la formation du complexe DnaB, DnaG, mais joue plutôt un rôle dans la modulation de la longueur des amorces synthétisées par DnaG.
Inhibition de l'ADNG
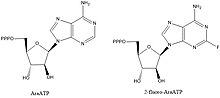
Les inhibiteurs d'ADN primases sont des composés précieux pour l'élucidation des voies biochimiques et des interactions clés, mais ils présentent également un intérêt en tant que composés phares pour concevoir des médicaments contre les maladies bactériennes. La plupart des composés connus pour inhiber les primases sont des analogues de nucléotides tels que l'AraATP (voir Vidarabine ) et le 2-fluoro-AraATP. Ces composés seront souvent utilisés comme substrats par la primase, mais une fois incorporés, la synthèse ou l'allongement ne peut plus se produire. Par exemple, E. coli DnaG utilisera les 2',3'-didésoxynucléoside 5'-triphosphates (ddNTP) comme substrats, qui agissent comme des terminateurs de chaîne en raison de l'absence d'un hydroxyle 3' pour former une liaison phosphodiester avec le nucléotide suivant.
Le nombre relativement faible d'inhibiteurs de primase reflète probablement la difficulté inhérente aux tests de primase plutôt qu'un manque de sites de liaison potentiels sur l'enzyme. La courte longueur des produits synthétisés et le taux généralement lent de l'enzyme par rapport à d'autres enzymes de réplication rendent plus difficile le développement d'approches de criblage à haut débit (HTS). Malgré les difficultés, il existe plusieurs inhibiteurs connus de DnaG qui ne sont pas des analogues de NTP. La doxorubicine et la suramine sont à la fois des inhibiteurs compétitifs de l'ADN et du NTP de Mycobacterium Tuberculosis DnaG. La suramine est également connue pour inhiber l'ADN primase eucaryote en entrant en compétition avec le GTP, de sorte que la suramine est susceptible d'inhiber le DnaG via un mécanisme similaire.
Liens externes
- dnaG+protein,+E+coli à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis
- DnaG+ (Primase) à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis
Les références