Affichage de l'ARNm - mRNA display
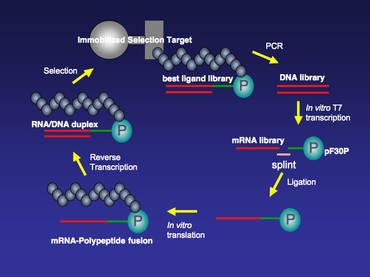
L'affichage de l'ARNm est une technique d'affichage utilisée pour l' évolution in vitro de protéines et/ou de peptides afin de créer des molécules pouvant se lier à une cible souhaitée. Le processus aboutit à des peptides ou des protéines traduits qui sont associés à leur progéniteur d' ARNm via une liaison puromycine . Le complexe se lie ensuite à une cible immobilisée dans une étape de sélection ( chromatographie d'affinité ). Les fusions ARNm-protéine qui se lient bien sont ensuite transcrites de manière inverse en ADNc et leur séquence est amplifiée via une réaction en chaîne par polymérase . Le résultat est une séquence nucléotidique qui code pour un peptide avec une affinité élevée pour la molécule d'intérêt.
La puromycine est un analogue de l'extrémité 3' d'un tyrosyl-ARNt dont une partie de sa structure imite une molécule d' adénosine et l'autre partie imite une molécule de tyrosine . Par rapport à la liaison ester clivable dans un tyrosyl-ARNt, la puromycine a une liaison amide non hydrolysable. En conséquence, la puromycine interfère avec la traduction et provoque une libération prématurée des produits de traduction.

Toutes les matrices d'ARNm utilisées pour la technologie d'affichage d'ARNm ont de la puromycine à leur extrémité 3'. Au fur et à mesure que la traduction progresse, le ribosome se déplace le long de la matrice d'ARNm, et une fois qu'il atteint l'extrémité 3' de la matrice, la puromycine fusionnée entrera dans le site A du ribosome et sera incorporée dans le peptide naissant. La fusion ARNm-polypeptide est ensuite libérée du ribosome (figure 1).
Pour synthétiser une fusion ARNm-polypeptide, la puromycine fusionnée n'est pas la seule modification de la matrice d'ARNm. Les oligonucléotides et autres espaceurs doivent être recrutés avec la puromycine pour fournir une flexibilité et une longueur appropriée à la puromycine pour entrer dans le site A. Idéalement, le lieur entre l'extrémité 3' d'un ARNm et la puromycine doit être suffisamment flexible et long pour permettre à la puromycine d'entrer dans le site A lors de la traduction du dernier codon. Cela permet la production efficace d'une fusion ARNm-polypeptide de haute qualité et de pleine longueur. Rihe Liu et al. optimisé l'espaceur oligonucléotidique 3'-puromycine. Ils ont rapporté que dA25 en combinaison avec un Spacer 9 (Glen Research) et dAdCdCP à l'extrémité 5' fonctionnaient le mieux pour la réaction de fusion. Ils ont découvert que les linkers plus longs que 40 nucléotides et plus courts que 16 nucléotides montraient une efficacité considérablement réduite de la formation de fusion. De plus, lorsque la séquence rUrUP était adjacente à la puromycine, la fusion ne s'est pas formée efficacement.
En plus de fournir une flexibilité et une longueur, la partie poly dA du lieur permet également une purification supplémentaire de la fusion ARNm-polypeptide en raison de sa haute affinité pour la résine de cellulose dT. Les fusions ARNm-polypeptide peuvent être sélectionnées sur des cibles de sélection immobilisées pendant plusieurs cycles avec une stringence croissante. Après chaque cycle de sélection, les membres de la bibliothèque qui restent liés à la cible immobilisée sont amplifiés par PCR et les non-liants sont lavés.
Méthode
La synthèse d'une bibliothèque d'affichage d'ARNm commence à partir de la synthèse d'une bibliothèque d'ADN. Une banque d'ADN pour toute protéine ou petit peptide d'intérêt peut être synthétisée par synthèse en phase solide suivie d'une amplification PCR. Habituellement, chaque membre de cette banque d'ADN possède un site de transcription de l'ARN polymérase T7 et un site de liaison ribosomique à l'extrémité 5'. La région du promoteur T7 permet à une transcription T7 in vitro à grande échelle de transcrire la bibliothèque d'ADN dans une bibliothèque d'ARNm, qui fournit ultérieurement des matrices pour la réaction de traduction in vitro . Le site de liaison ribosomique dans la région 5' non traduite (5' UTR) est conçu selon le système de traduction in vitro à utiliser. Il existe deux systèmes de traduction in vitro populaires disponibles dans le commerce . L'un est le système d'extrait E. Coli S30 (Promega) qui nécessite une séquence Shine-Dalgarno dans l'UTR 5' comme site de liaison ribosomique ; l'autre est le Red Nova Lysate (Novagen), qui nécessite un site de liaison ribosomique ΔTMV.
Une fois la bibliothèque d'ARNm générée, elle sera purifiée à l'urée-PAGE et ligaturée à l'aide d' ADN ligase T4 au lieur espaceur d'ADN contenant de la puromycine à l'extrémité 3'. Dans cette étape de ligature, un morceau d'ARNm est ligaturé avec un ADN simple brin à l'aide de l'ADN ligase T4. Il ne s'agit pas d'une réaction de ligature d'ADN ligase T4 standard, où deux morceaux d'ADN double brin sont ligaturés ensemble. Pour augmenter le rendement de cette ligature spéciale, une attelle d'ADN simple brin peut être utilisée pour faciliter la réaction de ligature. L'extrémité 5' de l'attelle est conçue pour être complémentaire à l'extrémité 3' de l'ARNm, et l'extrémité 3' de l'attelle est conçue pour être complémentaire à l'extrémité 5' de l'espaceur de l'ADN, qui se compose généralement de poly nucléotides dA (Figure 2).
La banque d'ARNm-ADN-puromycine ligaturée est traduite dans Red Nova Lysate (Novagen) ou E. Coli S30 Extract System (Promega), résultant en des polypeptides liés de manière covalente en cis à l'ARNm codant. La traduction in vitro peut également se faire dans un système PURE (synthèse de protéines utilisant des éléments recombinants). Le système PURE est un système de traduction sans cellules E. Coli dans lequel seuls les composants essentiels de la traduction sont présents. Certains composants, tels que les acides aminés et les aminoacyl-ARNt synthases (AARS) peuvent être omis du système. Au lieu de cela, de l'ARNt acylé chimiquement peut être ajouté dans le système PURE. Il a été démontré que certains acides aminés non naturels, tels que l'ARNt acylé d'acides aminés N-méthyle, peuvent être incorporés dans des peptides ou des fusions ARNm-polypeptide dans un système PURE.
Après traduction, les portions d'ARNm simple brin des fusions seront converties en hétéroduplex d'ARN/ADN par la transcriptase inverse pour éliminer toute structure secondaire d'ARN indésirable et rendre la portion d'acide nucléique de la fusion plus stable. Cette étape est une réaction de transcription inverse standard. Par exemple, cela peut être fait en utilisant Superscript II (GIBCO-BRL) en suivant le protocole du fabricant.
Les fusions ARNm/ADN-polypeptide peuvent être sélectionnées sur des cibles de sélection immobilisées pendant plusieurs tours (figure 3). Il peut y avoir un bruit de fond relativement élevé pour les premiers tours de sélection, et cela peut être minimisé en augmentant la rigueur de la sélection, par exemple en ajustant la concentration en sel, la quantité de détergent et/ou la température pendant la période de liaison cible/fusion. Après la sélection de liaison, les membres de la bibliothèque qui restent liés à la cible immobilisée sont amplifiés par PCR. L'étape d'amplification PCR enrichira la population à partir de la bibliothèque d'affichage d'ARNm qui a une affinité plus élevée pour la cible immobilisée. Une PCR sujette aux erreurs peut également être effectuée entre chaque cycle de sélection pour augmenter encore la diversité de la bibliothèque d'affichage d'ARNm et réduire le bruit de fond dans la sélection.
Un protocole moins long pour l'affichage de l'ARNm a été récemment publié.
Avantages
Bien qu'il existe de nombreuses autres technologies d'affichage moléculaire, telles que l'affichage sur phage , l'affichage bactérien , l'affichage sur levure et l' affichage sur ribosome , la technologie d'affichage de l'ARNm présente de nombreux avantages par rapport aux autres. Les trois premières bibliothèques de présentation biologique répertoriées contiennent des polypeptides ou des protéines exprimés à la surface du micro-organisme respectif et les informations de codage qui l'accompagnent pour chaque polypeptide ou protéine peuvent être récupérées à partir du génome du micro-organisme. Cependant, la taille de la bibliothèque pour ces trois systèmes d'affichage in vivo est limitée par l'efficacité de transformation de chaque organisme. Par exemple, la taille de la bibliothèque pour l'affichage des phages et des bactéries est limitée à 1-10 × 10^9 membres différents. La taille de la bibliothèque pour l'affichage de la levure est encore plus petite. De plus, ces systèmes d'affichage à base de cellules ne permettent que le criblage et l'enrichissement de peptides/protéines contenant des acides aminés naturels. En revanche, la présentation de l'ARNm et la présentation du ribosome sont des méthodes de sélection in vitro . Ils permettent une taille de bibliothèque aussi grande que 10^15 membres différents. La grande taille de la bibliothèque augmente la probabilité de sélectionner des séquences très rares, et améliore également la diversité des séquences sélectionnées. De plus, les méthodes de sélection in vitro éliminent la pression de sélection indésirable, telle qu'une mauvaise expression des protéines et une dégradation rapide des protéines, ce qui peut réduire la diversité des séquences sélectionnées. Enfin, les méthodes de sélection in vitro permettent l'application de techniques de mutagenèse et de recombinaison in vitro tout au long du processus de sélection.
Bien que l'affichage du ribosome et l'affichage de l'ARNm soient tous deux des méthodes de sélection in vitro , l'affichage de l'ARNm présente un certain avantage par rapport à la technologie d'affichage du ribosome. L'affichage de l'ARNm utilise des complexes covalents ARNm-polypeptide liés par la puromycine; alors que la présentation du ribosome utilise des complexes ribosome-ARNm-polypeptide bloqués et non covalents. Pour l'affichage des ribosomes, la stringence de sélection est limitée pour maintenir le ribosome-ARNm-polypeptide dans un complexe en raison des complexes ribosome-ARNm-polypeptide non covalents. Cela peut entraîner des difficultés pour réduire la liaison d'arrière-plan pendant le cycle de sélection. De plus, les polypeptides sélectionnés dans un système de présentation de ribosomes sont attachés à un énorme complexe ARNr-protéine, un ribosome, qui a un poids moléculaire de plus de 2 000 000 Da. Il peut y avoir une interaction imprévisible entre la cible de sélection et le ribosome, ce qui peut entraîner une perte de liants potentiels au cours du cycle de sélection. En revanche, le lieur espaceur d'ADN puromycine utilisé dans la technologie d'affichage d'ARNm est beaucoup plus petit par rapport à un ribosome. Ce lieur peut avoir moins de chance d'interagir avec une cible de sélection immobilisée. Ainsi, la technologie d'affichage de l'ARNm est plus susceptible de donner des résultats moins biaisés.
Application
En 1997, Roberts et Szostak ont montré que les fusions entre un ARNm synthétique et son épitope myc codé pouvaient être enrichies à partir d'un pool de fusions ARNm-polypeptide à séquence aléatoire par immunoprécipitation.
Neuf ans plus tard, Fukuda et ses collègues ont choisi la méthode d'affichage de l'ARNm pour l' évolution in vitro de fragments d'anticorps Fv (scFv) à chaîne unique. Ils ont sélectionné six mutants scFv différents avec cinq mutations consensus. Cependant, l'analyse cinétique de ces mutants a montré que leur spécificité antigénique restait similaire à celle du type sauvage. Cependant, ils ont démontré que deux des cinq mutations consensus se trouvaient dans les régions déterminant la complémentarité (CDR). Et ils ont conclu que l'affichage de l'ARNm a le potentiel pour une évolution artificielle rapide des anticorps diagnostiques et thérapeutiques de haute affinité en optimisant leurs CDR.
Roberts et ses collaborateurs ont démontré que des oligomères peptidiques non naturels constitués d'un acide aminé N-substitué peuvent être synthétisés sous forme de fusions ARNm-polypeptide. Les peptides contenant des acides aminés N-substitués ont été associés à une bonne stabilité protéolytique et à des propriétés pharmacocinétiques améliorées. Ce travail indique que la technologie d'affichage de l'ARNm a le potentiel de sélectionner des peptides de type médicament pour un usage thérapeutique résistant à la protéolyse.