Affichage des phages - Phage display
Le phage display est une technique de laboratoire pour l'étude des interactions protéine-protéine , protéine - peptide et protéine- ADN qui utilise des bactériophages ( virus qui infectent les bactéries ) pour relier les protéines à l' information génétique qui les code . Dans cette technique, un gène codant pour une protéine d'intérêt est inséré dans un gène de protéine d'enveloppe de phage , ce qui amène le phage à « afficher » la protéine à l'extérieur tout en contenant le gène de la protéine à l'intérieur, ce qui entraîne une connexion entre le génotype et phénotype . Ces phages d'affichage peuvent ensuite être criblés contre d'autres protéines, peptides ou séquences d'ADN, afin de détecter une interaction entre la protéine affichée et ces autres molécules. De cette façon, de grandes bibliothèques de protéines peuvent être criblées et amplifiées dans un processus appelé sélection in vitro , qui est analogue à la sélection naturelle .
Le plus bactériophages couramment utilisés dans l' affichage de phages sont M13 et fd phages filamenteux , bien que T4 , T7 et λ phage ont également été utilisés.
Histoire
La présentation des phages a été décrite pour la première fois par George P. Smith en 1985, lorsqu'il a démontré la présentation de peptides sur des phages filamenteux (virus longs et minces qui infectent les bactéries) en fusionnant la protéine de capside du virus à un peptide d'une collection de séquences peptidiques. Cela a montré les différents peptides sur les surfaces externes de la collection de clones viraux, où l'étape de criblage du processus a isolé les peptides avec la plus haute affinité de liaison. En 1988, Stephen Parmley et George Smith ont décrit le biopanning pour la sélection par affinité et ont démontré que des cycles de sélection récursifs pouvaient enrichir les clones présents à 1 sur un milliard ou moins. En 1990, Jamie Scott et George Smith ont décrit la création de grandes bibliothèques de peptides aléatoires affichées sur des phages filamenteux. La technologie d'affichage sur phage a été développée et améliorée par des groupes du Laboratoire de biologie moléculaire avec Greg Winter et John McCafferty , du Scripps Research Institute avec Richard Lerner et Carlos Barbas et du Centre allemand de recherche sur le cancer avec Frank Breitling et Stefan Dübel pour l'affichage de protéines telles que comme anticorps pour l' ingénierie des protéines thérapeutiques . Smith et Winter ont reçu la moitié du prix Nobel de chimie 2018 pour leur contribution au développement de la présentation sur phage. Un brevet de George Pieczenik revendiquant la priorité de 1985 décrit également la génération de bibliothèques de peptides.
Principe
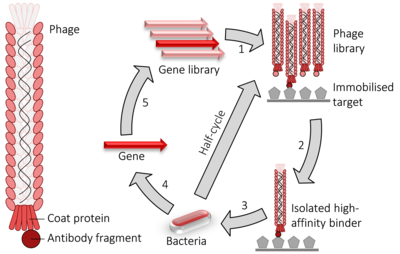
Comme le système à deux hybrides , la présentation sur phage est utilisée pour le criblage à haut débit des interactions protéiques. Dans le cas de la présentation sur phage filamenteux M13 , l'ADN codant pour la protéine ou le peptide d'intérêt est ligaturé dans le gène pIII ou pVIII, codant pour la protéine d'enveloppe mineure ou majeure , respectivement. Des sites de clonage multiples sont parfois utilisés pour garantir que les fragments sont insérés dans les trois cadres de lecture possibles afin que le fragment d' ADNc soit traduit dans le cadre approprié. Le gène de phage et l'hybride d'ADN d' insertion sont ensuite insérés (un processus connu sous le nom de « transduction ») dans des cellules bactériennes E. coli telles que TG1, SS320, ER2738 ou XL1-Blue E. coli . Si un « phagemide » vecteur est utilisé (un vecteur de construction d'affichage simplifié) des particules de phage ne seront pas libérés des E. coli cellules jusqu'à ce qu'elles sont infectées par le phage auxiliaire , qui permet le conditionnement de l'ADN de phage et l' assemblage des matures virions avec le fragment de protéine pertinent dans le cadre de leur enveloppe externe sur la protéine d'enveloppe mineure (pIII) ou majeure (pVIII). En immobilisant une ou des cibles d'ADN ou de protéines pertinentes à la surface d'un puits de plaque de microtitrage , un phage qui affiche une protéine qui se lie à l'une de ces cibles sur sa surface restera tandis que d'autres seront éliminés par lavage. Ceux qui restent peuvent être élués , utilisés pour produire plus de phages (par infection bactérienne avec des phages auxiliaires) et pour produire un mélange de phages enrichi en phages pertinents (c'est-à-dire de liaison). Le cycle répété de ces étapes est appelé « panning » , en référence à l'enrichissement d'un échantillon d'or en éliminant les matériaux indésirables. Le phage élué dans l'étape finale peut être utilisé pour infecter un hôte bactérien approprié, à partir duquel les phagémides peuvent être collectés et la séquence d'ADN pertinente excisée et séquencée pour identifier les protéines ou fragments de protéines interagissant pertinents.
L'utilisation d'un phage auxiliaire peut être éliminée en utilisant la technologie de « lignée cellulaire d'emballage bactérien ».
L'élution peut être réalisée en combinant un tampon d' élution à faible pH avec une sonification, qui, en plus de relâcher l'interaction peptide-cible, sert également à détacher la molécule cible de la surface d'immobilisation. Cette méthode basée sur les ultrasons permet la sélection en une seule étape d'un peptide de haute affinité.
Applications
Les applications de la technologie de présentation sur phage comprennent la détermination des partenaires d'interaction d'une protéine (qui serait utilisée comme « appât » de phage immobilisé avec une banque d'ADN constituée de toutes les séquences codantes d'une cellule, d'un tissu ou d'un organisme) de sorte que la fonction ou le mécanisme de la fonction de cette protéine peut être déterminée. La présentation sur phage est également une méthode largement utilisée pour l'évolution des protéines in vitro (également appelée ingénierie des protéines ). En tant que tel, la présentation sur phage est un outil utile dans la découverte de médicaments . Il est utilisé pour trouver de nouveaux ligands (inhibiteurs enzymatiques, agonistes et antagonistes des récepteurs) pour cibler les protéines. La technique est également utilisée pour déterminer les antigènes tumoraux (à utiliser dans le diagnostic et le ciblage thérapeutique) et dans la recherche d' interactions protéine-ADN à l' aide de bibliothèques d'ADN spécialement construites avec des segments randomisés. Récemment, la présentation sur phage a également été utilisée dans le cadre de traitements contre le cancer - tels que l'approche de transfert cellulaire adoptif. Dans ces cas, la présentation sur phage est utilisée pour créer et sélectionner des anticorps synthétiques qui ciblent les protéines de surface tumorale. Ceux-ci sont transformés en récepteurs synthétiques pour les lymphocytes T collectés auprès du patient et utilisés pour lutter contre la maladie.
Méthodes concurrentes pour in vitro l' évolution des protéines comprennent l' affichage de levure , affichage bactérien , affichage ribosome et affichage de l' ARNm .
Maturation des anticorps in vitro
L'invention de la présentation sur phage d'anticorps a révolutionné la découverte de médicaments à base d'anticorps. Les premiers travaux ont été effectués par des laboratoires du MRC Laboratory of Molecular Biology ( Greg Winter et John McCafferty ), du Scripps Research Institute (Richard Lerner et Carlos F. Barbas) et du German Cancer Research Center (Frank Breitling et Stefan Dübel). En 1991, le groupe Scripps a signalé la première exposition et sélection d'anticorps humains sur un phage. Cette étude initiale a décrit l'isolement rapide de l'anticorps humain Fab qui liait la toxine tétanique et la méthode a ensuite été étendue pour cloner rapidement des anticorps humains anti-VIH-1 pour la conception et la thérapie de vaccins.
La présentation sur phage de bibliothèques d'anticorps est devenue une méthode puissante pour étudier à la fois la réponse immunitaire et une méthode pour sélectionner et faire évoluer rapidement des anticorps humains pour la thérapie. La présentation sur phage d'anticorps a ensuite été utilisée par Carlos F. Barbas du Scripps Research Institute pour créer des bibliothèques d'anticorps humains synthétiques, un principe breveté pour la première fois en 1990 par Breitling et ses collègues (brevet CA 2035384), permettant ainsi de créer des anticorps humains in vitro à partir de produits synthétiques. éléments de diversité.
Les bibliothèques d'anticorps affichant des millions d'anticorps différents sur les phages sont souvent utilisées dans l'industrie pharmaceutique pour isoler des pistes d'anticorps thérapeutiques hautement spécifiques, pour le développement en médicaments à base d'anticorps principalement comme thérapies anticancéreuses ou anti-inflammatoires. L'un des plus réussis a été l' adalimumab , découvert par Cambridge Antibody Technology sous le nom de D2E7 et développé et commercialisé par Abbott Laboratories . L'adalimumab, un anticorps anti-TNF alpha , a été le premier anticorps entièrement humain au monde, qui a réalisé des ventes annuelles supérieures à 1 milliard de dollars.
Protocole général
Vous trouverez ci-dessous la séquence d'événements qui sont suivies dans le criblage de phage display pour identifier les polypeptides qui se lient avec une affinité élevée à la protéine cible ou à la séquence d'ADN souhaitée :
- Les protéines cibles ou les séquences d'ADN sont immobilisées dans les puits d'une plaque de microtitration .
- De nombreuses séquences génétiques sont exprimées dans une banque de bactériophages sous forme de fusions avec la protéine d'enveloppe du bactériophage, de sorte qu'elles sont affichées à la surface de la particule virale. La protéine affichée correspond à la séquence génétique au sein du phage.
- Cette bibliothèque d'affichage de phage est ajoutée à la boîte et après avoir laissé le temps au phage de se lier, la boîte est lavée.
- Les protéines présentant des phages qui interagissent avec les molécules cibles restent attachées à la boîte, tandis que toutes les autres sont emportées.
- Le phage attaché peut être élué et utilisé pour créer plus de phage par infection d'hôtes bactériens appropriés. Le nouveau phage constitue un mélange enrichi, contenant considérablement moins de phages non pertinents (c'est-à-dire non liants) que ceux présents dans le mélange initial.
- Les étapes 3 à 5 sont éventuellement répétées une ou plusieurs fois, enrichissant davantage la bibliothèque de phages en protéines de liaison.
- Après une amplification bactérienne supplémentaire, l'ADN contenu dans le phage en interaction est séquencé pour identifier les protéines ou fragments de protéines en interaction.
Sélection de la protéine d'enveloppe
Phages filamenteux
pIII
pIII est la protéine qui détermine l'infectivité du virion. pIII est composé de trois domaines (N1, N2 et CT) reliés par des lieurs riches en glycine. Le domaine N2 se lie au pilus F lors de l'infection par le virion libérant le domaine N1 qui interagit alors avec une protéine TolA à la surface de la bactérie. Les insertions au sein de cette protéine sont généralement ajoutées en position 249 (au sein d'une région de liaison entre CT et N2), en position 198 (au sein du domaine N2) et à l'extrémité N-terminale (insérée entre la séquence de sécrétion N-terminale et l'extrémité N-terminale de pIII). Cependant, lors de l'utilisation du site BamHI situé à la position 198, il faut faire attention au résidu cystéine non apparié (C201) qui pourrait causer des problèmes lors de la présentation du phage si l'on utilise une version non tronquée de pIII.
Un avantage d'utiliser pIII plutôt que pVIII est que pIII permet une présentation monovalente lors de l'utilisation d'un phagemide (plasmide dérivé des phages Ff ) combiné avec un phage auxiliaire. De plus, pIII permet l'insertion de séquences protéiques plus grandes (> 100 acides aminés) et y est plus tolérant que pVIII. Cependant, l'utilisation de pIII comme partenaire de fusion peut entraîner une diminution de l'infectivité des phages, entraînant des problèmes tels qu'un biais de sélection causé par une différence de taux de croissance des phages ou pire encore, l'incapacité du phage à infecter son hôte. La perte de l'infectivité du phage peut être évitée en utilisant un plasmide phagémide et un phage auxiliaire de sorte que le phage résultant contienne à la fois un pIII de type sauvage et de fusion.
L'ADNc a également été analysé en utilisant pIII via un système à deux fermetures à glissière leucine complémentaires, Direct Interaction Rescue ou en ajoutant un lieur de 8 à 10 acides aminés entre l'ADNc et pIII à l'extrémité C-terminale.
pVIII
pVIII est la principale protéine d'enveloppe des phages Ff. Les peptides sont généralement fusionnés à l'extrémité N-terminale de pVIII. Habituellement, les peptides qui peuvent être fusionnés à pVIII ont une longueur de 6 à 8 acides aminés. La restriction de taille semble avoir moins à voir avec l'obstacle structurel causé par la section ajoutée et plus à voir avec l'exclusion de taille causée par pIV pendant l'exportation de la protéine d'enveloppe. Puisqu'il existe environ 2700 copies de la protéine sur un phage typique, il est plus probable que la protéine d'intérêt sera exprimée de manière polyvalente même si un phagemide est utilisé. Cela rend l'utilisation de cette protéine défavorable pour la découverte de partenaires de liaison de haute affinité.
Pour surmonter le problème de taille de pVIII, des protéines d'enveloppe artificielles ont été conçues. Un exemple est la protéine d'enveloppe artificielle inversée (ACP) de Weiss et Sidhu qui permet l'affichage de grandes protéines à l'extrémité C-terminale. Les ACP pourraient afficher une protéine de 20 kDa, cependant, uniquement à de faibles niveaux (principalement uniquement de manière monovalente).
pVI
pVI a été largement utilisé pour l'affichage de bibliothèques d'ADNc. L'affichage de bibliothèques d'ADNc via phage display est une alternative intéressante à la méthode hybride levure-2 pour la découverte de protéines et de peptides en interaction en raison de sa capacité de débit élevé. pVI a été utilisé préférentiellement à pVIII et pIII pour l'expression de bibliothèques d'ADNc car on peut ajouter la protéine d'intérêt à l'extrémité C-terminale de pVI sans affecter grandement le rôle de pVI dans l'assemblage des phages. Cela signifie que le codon stop dans l'ADNc n'est plus un problème. Cependant, la présentation sur phage de l'ADNc est toujours limitée par l'incapacité de la plupart des procaryotes à produire des modifications post-traductionnelles présentes dans les cellules eucaryotes ou par le mauvais repliement de protéines multidomaines.
Alors que pVI a été utile pour l'analyse de bibliothèques d'ADNc, pIII et pVIII restent les protéines d'enveloppe les plus utilisées pour la présentation sur phage.
pVII et pIX
Dans une expérience en 1995, l'affichage de la glutathion S-transférase a été tenté à la fois sur pVII et pIX et a échoué. Cependant, la présentation sur phage de cette protéine a été réalisée avec succès après l'ajout d'une séquence signal périplasmique (pelB ou ompA) sur l'extrémité N-terminale. Dans une étude récente, il a été démontré qu'AviTag, FLAG et His pouvaient être affichés sur pVII sans avoir besoin d'une séquence signal. Ensuite, l'expression des Fv monocaténaires (scFv) et des récepteurs des cellules T monocaténaires (scTCR) a été exprimée à la fois avec et sans la séquence signal.
PelB (une séquence signal d'acides aminés qui cible la protéine vers le périplasme où une peptidase signal clive ensuite PelB) a amélioré le niveau d'affichage du phage par rapport aux fusions pVII et pIX sans la séquence signal. Cependant, cela a conduit à l'incorporation de plus de génomes de phages auxiliaires plutôt que de génomes de phagemides. Dans tous les cas, les niveaux de phage display étaient inférieurs à ceux de la fusion pIII. Cependant, un affichage inférieur pourrait être plus favorable pour la sélection de liants en raison d'un affichage inférieur étant plus proche d'un véritable affichage monovalent. Dans cinq fois sur six, les fusions pVII et pIX sans pelB étaient plus efficaces que les fusions pIII dans les tests de sélection par affinité. Le document poursuit même en affirmant que les plates-formes d'affichage pVII et pIX peuvent surpasser pIII à long terme.
L'utilisation de pVII et pIX au lieu de pIII pourrait également être un avantage car la récupération du virion peut être entreprise sans rompre la liaison virion-antigène si le pIII utilisé est de type sauvage. Au lieu de cela, on pourrait couper dans une section entre la bille et l'antigène pour éluer. Puisque le pIII est intact, peu importe que l'antigène reste lié au phage.
Phage T7
Le problème de l'utilisation des phages Ff pour la présentation sur phage est qu'ils nécessitent que la protéine d'intérêt soit transloquée à travers la membrane interne bactérienne avant d'être assemblées dans le phage. Certaines protéines ne peuvent pas subir ce processus et ne peuvent donc pas être affichées à la surface des phages Ff. Dans ces cas, l'affichage sur phage T7 est utilisé à la place. Dans la présentation sur phage T7, la protéine à afficher est attachée à l'extrémité C-terminale de la protéine de capside du gène 10 de T7.
L'inconvénient de l'utilisation de T7 est que la taille de la protéine qui peut être exprimée à la surface est limitée à des peptides plus courts, car de grands changements dans le génome de T7 ne peuvent pas être pris en compte comme c'est le cas dans M13 où le phage allonge simplement son manteau pour s'adapter au plus grand génome en son sein. Cependant, il peut être utile pour la production d'une grande banque de protéines pour la sélection scFV où le scFV est exprimé sur un phage M13 et les antigènes sont exprimés à la surface du phage T7.
Ressources et outils bioinformatiques
Les bases de données et les outils de calcul pour les mimotopes ont été une partie importante de l'étude de la présentation des phages. Les bases de données, les programmes et les serveurs Web ont été largement utilisés pour exclure les peptides non liés à la cible, caractériser les interactions petites molécules-protéines et cartographier les interactions protéine-protéine. Les utilisateurs peuvent utiliser la structure tridimensionnelle d'une protéine et les peptides sélectionnés à partir de l'expérience d'affichage sur phage pour cartographier les épitopes conformationnels. Certaines des méthodes de calcul rapides et efficaces sont disponibles en ligne.
Voir également
Techniques de compétition :
Les références
Lectures complémentaires
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). "Bases de la technologie d'affichage de phage d'anticorps" (PDF) . Toxines . 10 (6) : 236. doi : 10.3390/toxines10060236 . PMC 6024766 . PMID 29890762 .
- Sélection versus conception en génie chimique
- La bibliothèque de phages d'anticorps humains ETH-2
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). « Affichage de phage pour la sélection de nouveaux peptides de liaison ». méth. Enzyme . Méthodes en enzymologie. 328 : 333-63. doi : 10.1016/S0076-6879(00)28406-1 . ISBN 9780121822293. PMID 11075354 .
Liens externes
|
Ressources de la bibliothèque sur l' affichage des phages |
