Polyomaviridae -Polyomaviridae
| Polyomaviridae | |
|---|---|
|
|
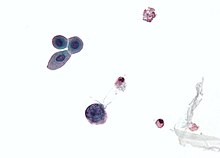
| Micrographie montrant une cellule infectée par le polyomavirus —grande cellule (bleue) en bas au centre à gauche. Spécimen de cytologie urinaire. | |
|
Classification des virus |
|
| (non classé): | Virus |
| Royaume : | Monodnavirie |
| Royaume: | Shotokuvirae |
| Phylum: | Cossaviricota |
| Classer: | Papovaviricetes |
| Commander: | Sépolyvirales |
| Famille: | Polyomaviridae |
| Genres | |
Les Polyomaviridae sont une famille de virus dont les hôtes naturelssont principalement les mammifères et les oiseaux. En 2020, il existe six genres et 117 espèces reconnus, dont cinq ne sont pas attribués à un genre. 14 espèces sont connues pour infecter l'homme, tandis que d'autres, comme le virus simien 40 , ont été identifiées chez l'homme dans une moindre mesure. La plupart de ces virus sont très courants et généralement asymptomatiques dans la plupart des populations humaines étudiées. Virus BK est associé à la néphropathie en transplantation rénale etpatients transplantés d'organes solides non rénales,virus JC avec leucoencéphalopathie multifocale progressive , et le virus de cellules de Merkel avec le cancer des cellules de Merkel.
Structure et génome

Les polyomavirus sont des virus à ADN double brin non enveloppés avec des génomes circulaires d'environ 5000 paires de bases . Le génome est emballé dans une capside virale d'environ 40 à 50 nanomètres de diamètre, de forme icosaédrique (symétrie T=7). La capside est composée de 72 capsomères pentamères d'une protéine appelée VP1 , qui est capable de s'auto-assembler en un icosaèdre fermé ; chaque pentamère de VP1 est associé à une molécule de l'une des deux autres protéines de capside, VP2 ou VP3 .

Le génome d'un polyomavirus typique code entre 5 et 9 protéines , divisées en deux régions transcriptionnelles appelées régions précoces et tardives en raison du temps pendant l'infection dans lequel elles sont transcrites. Chaque région est transcrite par l' ARN polymérase II de la cellule hôte en un seul ARN pré-messager contenant plusieurs gènes. La région précoce code généralement pour deux protéines, les petits et les grands antigènes tumoraux, produits par épissage alternatif . La région tardive contient les trois protéines structurelles de capside VP1, VP2 et VP3, produites par d'autres sites d' initiation de la traduction . Des gènes supplémentaires et d'autres variations sur ce thème sont présents dans certains virus : par exemple, les polyomavirus de rongeurs ont une troisième protéine appelée antigène tumoral moyen dans la région précoce, qui est extrêmement efficace pour induire la transformation cellulaire ; SV40 a une protéine de capside supplémentaire VP4; certains exemples ont une protéine régulatrice supplémentaire appelée agnoprotéine exprimée à partir de la région tardive. Le génome contient également une région de contrôle ou de régulation non codante contenant les promoteurs des régions précoces et tardives , les sites de démarrage de la transcription et l' origine de la réplication .
Réplication et cycle de vie
Le cycle de vie du polyomavirus commence par l'entrée dans une cellule hôte . Les récepteurs cellulaires des polyomavirus sont des résidus d' acide sialique de glycanes , généralement des gangliosides . La fixation des polyomavirus aux cellules hôtes est médiée par la liaison de VP1 aux glycanes sialylés sur la surface cellulaire. Dans certains virus particuliers, des interactions cellulaires supplémentaires se produisent; par exemple, on pense que le virus JC nécessite une interaction avec le récepteur 5HT2A et le virus des cellules de Merkel avec du sulfate d'héparane . Cependant, en général, les interactions virus-cellule sont médiées par des molécules couramment présentes à la surface cellulaire et ne contribuent donc probablement pas de manière majeure au tropisme de type cellulaire observé chez les virus individuels . Après s'être lié à des molécules à la surface cellulaire, le virion est endocytosé et pénètre dans le réticulum endoplasmique - un comportement unique parmi les virus non enveloppés connus - où la structure de la capside virale est susceptible d'être perturbée par l'action des enzymes disulfure isomérase de la cellule hôte .
Les détails du transit vers le noyau ne sont pas clairs et peuvent varier selon les polyomavirus individuels. Il a été fréquemment rapporté qu'une particule de virion intacte, bien que déformée, est libérée du réticulum endoplasmique dans le cytoplasme cellulaire, où le génome est libéré de la capside, probablement en raison de la faible concentration en calcium dans le cytoplasme. L'expression des gènes viraux et la réplication du génome viral se produisent dans le noyau en utilisant la machinerie de la cellule hôte. Les gènes précoces - comprenant au minimum le petit antigène tumoral (ST) et le grand antigène tumoral (LT) - sont exprimés en premier, à partir d'un seul brin d' ARN messager épissé alternativement . Ces protéines servent à manipuler le de l' hôte du cycle cellulaire - dérèglement de la transition de la phase G1 à la phase S , alors que le génome de la cellule hôte est répliqué - parce que la machinerie de replication de l' ADN de la cellule hôte est nécessaire pour la replication du génome viral. Le mécanisme précis de cette dérégulation dépend du virus ; par exemple, SV40 LT peut se lier directement à la cellule hôte p53 , mais pas le polyomavirus murin LT. La LT induit la réplication de l'ADN à partir de la région de contrôle non codante du génome viral (NCCR), après quoi l'expression de l'ARNm précoce est réduite et l'expression de l'ARNm tardif, qui code pour les protéines de la capside virale, commence. Au début de ces interactions, les LT appartenant à plusieurs polyomavirus, dont le polyomavirus des cellules de Merkel , présentent un potentiel oncogène. Plusieurs mécanismes ont été décrits pour réguler la transition de l'expression génique précoce à tardive, notamment l'implication de la protéine LT dans la répression du promoteur précoce, l'expression d'ARNm tardifs non terminés avec des extensions complémentaires à l'ARNm précoce et l'expression de microARN régulateurs. . L'expression des gènes tardifs entraîne l'accumulation des protéines de capside virale dans le cytoplasme de la cellule hôte. Les composants de la capside pénètrent dans le noyau afin d'encapsider un nouvel ADN génomique viral. De nouveaux virions peuvent être assemblés dans des usines virales . Le mécanisme de libération virale de la cellule hôte varie selon les polyomavirus ; certaines expriment des protéines qui facilitent la sortie cellulaire, comme l' agnoprotéine ou VP4 . Dans certains cas, des niveaux élevés de virus encapsidés entraînent une lyse cellulaire , libérant les virions.
Protéines virales
Antigènes tumoraux
Le grand antigène tumoral joue un rôle clé dans la régulation du cycle de vie viral en se liant à l'origine virale de la réplication de l'ADN où il favorise la synthèse de l'ADN. De plus, comme le polyomavirus s'appuie sur la machinerie de la cellule hôte pour se répliquer, la cellule hôte doit être en phase s pour que cela commence. Pour cette raison, le grand antigène T module également les voies de signalisation cellulaire pour stimuler la progression du cycle cellulaire en se liant à un certain nombre de protéines de contrôle cellulaire. Ceci est réalisé par une attaque à deux volets consistant à inhiber les gènes suppresseurs de tumeurs p53 et les membres de la famille des rétinoblastomes (pRB), et à stimuler les voies de croissance cellulaire en liant l'ADN cellulaire, l'association ATPase-hélicase, l'ADN polymérase et la liaison des facteurs complexes de pré-initiation de la transcription. . Cette stimulation anormale du cycle cellulaire est une force puissante pour la transformation oncogène.
La petite protéine antigénique tumorale est également capable d'activer plusieurs voies cellulaires qui stimulent la prolifération cellulaire. Les petits antigènes T du polyomavirus ciblent généralement la protéine phosphatase 2A ( PP2A ), un régulateur multi-sous-unité clé de plusieurs voies, notamment Akt , la voie de la protéine kinase activée par les mitogènes (MAPK) et la voie de la protéine kinase activée par le stress (SAPK). Le petit antigène T du polyomavirus des cellules de Merkel code pour un domaine unique, appelé domaine de stabilisation LT (LSD), qui se lie à et inhibe la ligase FBXW7 E3 régulant à la fois les oncoprotéines cellulaires et virales. Contrairement au SV40, le petit antigène T du MCV transforme directement les cellules de rongeur in vitro.
L' antigène tumoral moyen est utilisé dans des organismes modèles développés pour étudier le cancer, tels que le système MMTV-PyMT où le T moyen est couplé au promoteur MMTV . Là, il fonctionne comme un oncogène , tandis que le tissu où la tumeur se développe est déterminé par le promoteur MMTV.
Protéines de capside
La capside du polyomavirus se compose d'un composant majeur, la protéine de capside majeure VP1 et d'un ou deux composants mineurs, les protéines de capside mineures VP2 et VP3 . Les pentamères VP1 forment la capside virale icosaédrique fermée , et à l'intérieur de la capside, chaque pentamère est associé à une molécule de VP2 ou VP3. Certains polyomavirus, tels que le polyomavirus des cellules de Merkel , ne codent ni n'expriment VP3. Les protéines de capside sont exprimées à partir de la région tardive du génome.
Agnoprotéine
L' agnoprotéine est une petite phosphoprotéine multifonctionnelle trouvée dans la partie codante tardive du génome de certains polyomavirus, notamment les virus BK , JC et SV40 . Il est essentiel à la prolifération des virus qui l'expriment et serait impliqué dans la régulation du cycle de vie viral, en particulier la réplication et la sortie virale de la cellule hôte, mais les mécanismes exacts ne sont pas clairs.
Taxonomie
Les polyomavirus sont membres du groupe I (virus à ADNdb). La classification des polyomavirus a fait l'objet de plusieurs révisions proposées à mesure que de nouveaux membres du groupe sont découverts. Autrefois, les polyomavirus et les papillomavirus , qui partagent de nombreuses caractéristiques structurelles mais ont des organisations génomiques très différentes, étaient classés ensemble dans la famille désormais obsolète des Papovaviridae . (Le nom Papovaviridae dérive de trois abréviations : Pa pour Papillomavirus , Po pour Polyomavirus et Va pour « vacuolating. ») Les polyomavirus ont été divisés en trois grands clades (c'est-à-dire des groupes génétiquement apparentés) : le clade SV40, le clade aviaire , et le clade du polyomavirus murin. Une reclassification ultérieure proposée par le Comité international de taxonomie des virus (ICTV) a recommandé de diviser la famille des Polyomaviridae en trois genres :
- Genre Orthopolyomavirus (espèce type SV40 )
- Genre Wukipolyomavirus (type espèce KI polyomavirus )
- Genre Avipolyomavirus (espèce type Polyomavirus aviaire )
Le système actuel de classification ICTV reconnaît six genres et 117 espèces, dont cinq n'ont pas pu se voir attribuer un genre. Ce système conserve la distinction entre les virus aviaires et mammifères, regroupant le sous-ensemble aviaire dans le genre Gammapolyomavirus . Les six genres sont :
- Alphapolyomavirus
- Bêtapolyomavirus
- Deltapolyomavirus
- Epsilonpolyomavirus
- Gammapolyomavirus
- Zêtapolyomavirus
Les espèces suivantes ne sont pas attribuées à un genre :
- Centropristis striata polyomavirus 1
- Rhynchobatus djiddensis polyomavirus 1
- Sparus aurata polyomavirus 1
- Trematomus bernacchii polyomavirus 1
- Trematomus pennellii polyomavirus 1
La description de virus supplémentaires est en cours. Ceux-ci incluent le polyomavirus de la loutre de mer 1 et le polyomavirus de l'alpaga. Un autre virus est le polyomavirus du panda géant 1. Un autre virus a été décrit chez les rongeurs sigmodontines. Un autre - tree shrew polyomavirus 1 - a été décrit chez la musaraigne arboricole.
Polyomavirus humains
La plupart des polyomavirus n'infectent pas les humains. Parmi les polyomavirus catalogués en 2017, 14 au total étaient connus avec des hôtes humains. Cependant, certains polyomavirus sont associés à des maladies humaines, en particulier chez les individus immunodéprimés . Le MCV est très différent des autres polyomavirus humains et est le plus étroitement apparenté au polyomavirus murin. Le polyomavirus associé à la trichodysplasie spinulosa (TSV) est éloigné du MCV. Deux virus—HPyV6 et HPyV7—sont les plus étroitement apparentés aux virus KI et WU, tandis que HPyV9 est plus étroitement apparenté au polyomavirus lymphotrope (LPV) dérivé du singe vert africain.
Un quatorzième virus a été décrit. Le polyomavirus du CIRC de Lyon est apparenté au polyomavirus du raton laveur.
Liste des polyomavirus humains
Les 14 polyomavirus suivants avec des hôtes humains ont été identifiés et leurs génomes ont été séquencés en 2017 :
| Espèce | Genre proposé | Nom du virus | Abréviation | NCBI RefSeq | Année de découverte | Corrélat clinique (le cas échéant) | Les références |
|---|---|---|---|---|---|---|---|
| Polyomavirus humain 5 | Alpha | Polyomavirus à cellules de Merkel | MCPyV | NC_010277 | 2008 | Cancer à cellules de Merkel | |
| Polyomavirus humain 8 | Alpha | Trichodysplasia spinulosa polyomavirus | TSPyV | NC_014361 | 2010 | Trichodysplasie spinuleuse | |
| Polyomavirus humain 9 | Alpha | Polyomavirus humain 9 | HPyV9 | NC_015150 | 2011 | Aucun connu | |
| Polyomavirus humain 12 | Alpha | Polyomavirus humain 12 | HPyV12 | NC_020890 | 2013 | Aucun connu | |
| Polyomavirus humain 13 | Alpha | Polyomavirus du New Jersey | NJPyV | NC_024118 | 2014 | Aucun connu | |
| Polyomavirus humain 1 | Bêta | Polyomavirus BK | BKPyV | NC_001538 | 1971 | Néphropathie associée au polyomavirus ; cystite hémorragique | |
| Polyomavirus humain 2 | Bêta | Polyomavirus JC | JCPyV | NC_001699 | 1971 | Leucoencéphalopathie multifocale progressive | |
| Polyomavirus humain 3 | Bêta | Polyomavirus KI | KIPyV | NC_009238 | 2007 | Aucun connu | |
| Polyomavirus humain 4 | Bêta | Polyomavirus WU | WUPyV | NC_009539 | 2007 | Aucun connu | |
| Polyomavirus humain 6 | Delta | Polyomavirus humain 6 | HPyV6 | NC_014406 | 2010 | Dermatose prurigineuse et dyskératosique associée à HPyV6 (H6PD) | |
| Polyomavirus humain 7 | Delta | Polyomavirus humain 7 | HPyV7 | NC_014407 | 2010 | Hyperplasie épithéliale liée à HPyV7 | |
| Polyomavirus humain 10 | Delta | MW polyomavirus | MWPyV | NC_018102 | 2012 | Aucun connu | |
| Polyomavirus humain 11 | Delta | polyomavirus STL | STLPyV | NC_0220106 | 2013 | Aucun connu | |
| Polyomavirus humain 14 | Alpha | Polyomavirus du CIRC de Lyon | LIPyV | NC_034253.1 | 2017 | Aucun connu |
Le deltapolyomavirus ne contient que les quatre virus humains indiqués dans le tableau ci-dessus. Les groupes Alpha et Beta contiennent des virus qui infectent une variété de mammifères. Le groupe Gamma contient les virus aviaires. Les associations de maladies cliniquement significatives ne sont montrées que là où une causalité est attendue.
Des anticorps contre le polyomavirus lymphotrope du singe ont été détectés chez l'homme, ce qui suggère que ce virus - ou un virus étroitement apparenté - peut infecter l'homme.
Pertinence clinique
Tous les polyomavirus sont des infections très courantes chez les enfants et les jeunes adultes. La plupart de ces infections semblent causer peu ou pas de symptômes. Ces virus sont probablement persistants toute la vie chez presque tous les adultes. Les maladies causées par les infections à polyomavirus humains sont les plus courantes chez les personnes immunodéprimées ; les associations comprennent la maladie du virus BK avec Néphropathie en transplantation rénale et les patients transplantés d'organes solides non rénales, le virus JC avec leucoencéphalopathie multifocale progressive , et le virus de cellules de Merkel (MCV) avec le cancer à cellules de Merkel .
SV40
Le SV40 se réplique dans les reins des singes sans provoquer de maladie, mais peut provoquer le cancer chez les rongeurs dans des conditions de laboratoire. Dans les années 1950 et au début des années 1960, bien plus de 100 millions de personnes pourraient avoir été exposées au SV40 en raison d'une contamination par le SV40 du vaccin antipoliomyélitique non détectée auparavant , ce qui suscite des inquiétudes quant à la possibilité que le virus puisse provoquer une maladie chez l'homme. Bien qu'il ait été rapporté comme présent dans certains cancers humains, y compris les tumeurs cérébrales , les tumeurs osseuses , les mésothéliomes et les lymphomes non hodgkiniens , une détection précise est souvent confondu par des niveaux élevés de réactivité croisée pour SV40 avec polyomavirus humains généralisées. La plupart des virologues rejettent le SV40 comme cause de cancers humains.
Diagnostic
Le diagnostic de polyomavirus survient presque toujours après la primo-infection car il est soit asymptomatique, soit subclinique. Les dosages d'anticorps sont couramment utilisés pour détecter la présence d'anticorps contre des virus individuels. Des tests de compétition sont fréquemment nécessaires pour distinguer des polyomavirus très similaires.
Dans les cas de leucoencéphalopathie multifocale progressive (LMP), un anticorps à réaction croisée contre l'antigène SV40 T (généralement Pab419) est utilisé pour colorer les tissus directement pour détecter la présence de l'antigène T du virus JC. La PCR peut être utilisée sur une biopsie du tissu ou du liquide céphalo-rachidien pour amplifier l'ADN du polyomavirus. Cela permet non seulement de détecter le polyomavirus mais aussi de quel sous-type il s'agit.
Il existe trois principales techniques de diagnostic utilisées pour le diagnostic de la réactivation du polyomavirus dans la néphropathie à polyomavirus (PVN) : la cytologie urinaire, la quantification de la charge virale à la fois dans l'urine et le sang, et une biopsie rénale . La réactivation du polyomavirus dans les reins et les voies urinaires provoque l'excrétion des cellules infectées, des virions et/ou des protéines virales dans l'urine. Cela permet à la cytologie urinaire d'examiner ces cellules, ce qui, s'il y a inclusion de polyomavirus dans le noyau, est un diagnostic d'infection. De plus, comme l'urine d'un individu infecté contiendra des virions et/ou de l'ADN viral, la quantification de la charge virale peut être effectuée par PCR. Ceci est également vrai pour le sang.
La biopsie rénale peut également être utilisée si les deux méthodes qui viennent d'être décrites ne sont pas concluantes ou si la charge virale spécifique pour le tissu rénal est souhaitée. De manière similaire à la cytologie urinaire, les cellules rénales sont examinées au microscope optique pour l'inclusion de polyomavirus dans le noyau, ainsi que la lyse cellulaire et les partiels viraux dans le liquide extracellulaire. La charge virale comme précédemment est également mesurée par PCR.
La coloration des tissus à l'aide d'un anticorps monoclonal dirigé contre l'antigène MCV T montre son utilité pour différencier le carcinome à cellules de Merkel des autres petites tumeurs à cellules rondes. Des tests sanguins pour détecter les anticorps contre le MCV ont été développés et montrent que l'infection par le virus est répandue bien que les patients atteints de carcinome à cellules de Merkel aient des réponses anticorps exceptionnellement plus élevées que les personnes infectées de manière asymptomatique.
Utilisation dans le traçage de la migration humaine
Le virus JC offre un marqueur génétique prometteur pour l'évolution et la migration humaines. Il est porté par 70 à 90 pour cent des humains et est généralement transmis des parents à la progéniture. Cette méthode ne semble pas fiable pour retracer l' origine africaine récente de l'homme moderne .
Histoire
Le polyomavirus murin a été le premier polyomavirus découvert, ayant été rapporté par Ludwik Gross en 1953 comme un extrait de leucémies de souris capable d'induire des tumeurs de la glande parotide . L'agent causal a été identifié comme un virus par Sarah Stewart et Bernice Eddy , d'après qui il était autrefois appelé « SE polyoma ». Le terme "polyome" fait référence à la capacité des virus à produire de multiples (poly-) tumeurs (-oma) dans certaines conditions. Le nom a été critiqué comme un « sandwich linguistique sans viande » (« sans viande » parce que les deux morphèmes dans « polyome » sont des affixes) donnant peu de renseignements sur la biologie des virus ; en fait, des recherches ultérieures ont montré que la plupart des polyomavirus provoquent rarement des maladies cliniquement significatives chez leurs organismes hôtes dans des conditions naturelles.
Des dizaines de polyomavirus ont été identifiés et séquencés dès 2017, infectant principalement les oiseaux et les mammifères. Deux polyomavirus sont connus pour infecter les poissons, le bar noir et la dorade royale . Au total, quatorze polyomavirus sont connus pour infecter les humains.
