Modèles moléculaires d'ADN - Molecular models of DNA
Les modèles moléculaires de structures d'ADN sont des représentations de la géométrie moléculaire et de la topologie des molécules d'acide désoxyribonucléique ( ADN ) à l'aide de plusieurs moyens, dans le but de simplifier et de présenter les propriétés physiques et chimiques essentielles des structures moléculaires de l'ADN in vivo ou en vitro . Ces représentations comprennent des sphères serrées ( modèles CPK ) en plastique, des fils métalliques pour les modèles squelettiques , des calculs graphiques et des animations par ordinateurs, des rendus artistiques. Les modèles moléculaires informatiques permettent également des animations et des simulations de dynamique moléculaire qui sont très importantes pour comprendre le fonctionnement de l'ADN in vivo .
Les modèles moléculaires informatiques les plus avancés de l'ADN impliquent des simulations de dynamique moléculaire et des calculs de mécanique quantique des vibro-rotations, des orbitales moléculaires délocalisées (MO), des moments dipolaires électriques , des liaisons hydrogène , etc. La modélisation de la dynamique moléculaire de l'ADN consiste à simuler les changements de géométrie moléculaire et de topologie de l'acide désoxyribonucléique ( ADN ) dans le temps en raison des interactions intra et intermoléculaires de l'ADN. Alors que les modèles moléculaires de molécules d'ADN telles que les sphères étroitement emballées (modèles CPK) en plastique ou en fils métalliques pour les modèles squelettiques sont des représentations utiles des structures statiques de l'ADN, leur utilité est très limitée pour représenter la dynamique complexe de l'ADN. La modélisation moléculaire par ordinateur permet à la fois des animations et des simulations de dynamique moléculaire qui sont très importantes pour comprendre le fonctionnement de l'ADN in vivo .
Histoire
Dès les toutes premières étapes des études structurelles de l'ADN par diffraction des rayons X et par des moyens biochimiques, des modèles moléculaires tels que le modèle à double hélice d'acide nucléique Watson-Crick ont été utilisés avec succès pour résoudre le « casse-tête » de la structure de l'ADN et découvrir comment le ce dernier concerne ses fonctions clés dans les cellules vivantes. Les premiers modèles de diffraction des rayons X de haute qualité de l'ADN-A ont été rapportés par Rosalind Franklin et Raymond Gosling en 1953. Rosalind Franklin a fait l'observation critique que l'ADN existe sous deux formes distinctes, A et B, et a produit les images les plus nettes des deux à travers Technique de diffraction des rayons X. Les premiers calculs de la transformée de Fourier d'une hélice atomique ont été rapportés un an plus tôt par Cochran, Crick et Vand, et ont été suivis en 1953 par le calcul de la transformée de Fourier d'une bobine enroulée par Crick.
Les informations structurelles sont générées à partir d'études de diffraction des rayons X de fibres d'ADN orientées à l'aide de modèles moléculaires d'ADN qui sont combinés à une analyse cristallographique et mathématique des modèles de rayons X.
Les premiers rapports d'un modèle moléculaire à double hélice de la structure de l'ADN-B ont été faits par James Watson et Francis Crick en 1953. Cette même année, Maurice F. Wilkins, A. Stokes et HR Wilson, ont signalé les premiers modèles de rayons X de dans ADN B vivo dans des têtes de spermatozoïdes de saumon partiellement orientées.
Le développement du premier modèle moléculaire correct d'ADN à double hélice par Crick et Watson n'aurait peut-être pas été possible sans les preuves biochimiques de l'appariement des bases de nucléotides ([A---T]; [C---G]), ou Les règles de Chargaff . Bien que de telles études initiales des structures de l'ADN à l'aide de modèles moléculaires aient été essentiellement statiques, leurs conséquences pour expliquer les fonctions in vivo de l'ADN étaient importantes dans les domaines de la biosynthèse des protéines et de la quasi-universalité du code génétique. Les études de transformation épigénétique de l'ADN in vivo ont cependant été beaucoup plus lentes à se développer malgré leur importance pour l'embryologie, la morphogenèse et la recherche sur le cancer. De telles dynamiques chimiques et réactions biochimiques de l'ADN sont beaucoup plus complexes que la dynamique moléculaire des interactions physiques de l'ADN avec l'eau, les ions et les protéines/enzymes dans les cellules vivantes.
Importance
Un vieux problème dynamique permanent est de savoir comment « l'auto-réplication » de l'ADN a lieu dans les cellules vivantes, ce qui devrait impliquer un déroulement transitoire des fibres d'ADN superenroulées. Bien que l'ADN soit constitué de molécules de biopolymère allongées relativement rigides et de très grande taille appelées fibres ou chaînes (qui sont constituées d' unités nucléotidiques répétitives de quatre types de base, attachées à des groupes désoxyribose et phosphate), sa structure moléculaire in vivo subit des changements de configuration dynamiques impliquant molécules d'eau et ions. Le superenroulement, l'accumulation d'histones dans les structures chromosomiques et d'autres aspects supramoléculaires impliquent également une topologie de l'ADN in vivo qui est encore plus complexe que la géométrie moléculaire de l'ADN, faisant ainsi de la modélisation moléculaire de l'ADN un problème particulièrement difficile pour les biologistes moléculaires et les biotechnologues. Comme d'autres grandes molécules et biopolymères, l'ADN existe souvent dans de multiples géométries stables (c'est-à-dire qu'il présente une isomérie conformationnelle ) et des états quantiques configurationnels qui sont proches les uns des autres en énergie sur la surface d'énergie potentielle de la molécule d'ADN.
Ces géométries moléculaires variables peuvent également être calculées, au moins en principe, en utilisant des méthodes de chimie quantique ab initio qui peuvent atteindre une précision élevée pour les petites molécules, bien que des allégations selon lesquelles une précision acceptable puisse également être obtenue pour les polynuclélotides et les conformations de l'ADN aient été récemment formulées sur la base des données spectrales du dichroïsme circulaire vibrationnel (VCD). De telles géométries quantiques définissent une classe importante de modèles moléculaires ab initio d'ADN dont l'exploration a à peine commencé, notamment en rapport avec les résultats obtenus par VCD en solution. Des comparaisons plus détaillées avec de tels calculs quantiques ab initio peuvent en principe être obtenues par spectroscopie RMN 2D-FT et études de relaxation de solutions de polynucléotides ou d'ADN spécifiquement marqué, comme par exemple avec des marqueurs au deutérium.
Dans une tournure intéressante des rôles, la molécule d'ADN a été proposée pour être utilisée pour l'informatique quantique via l'ADN. Des nanostructures d'ADN et des biopuces de calcul d'ADN ont été construites.
Concepts fondamentaux
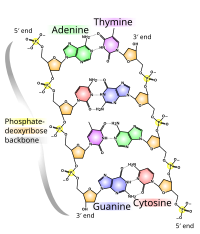
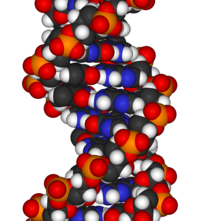
La structure chimique de l'ADN est insuffisante pour comprendre la complexité des structures 3D de l'ADN. En revanche, les modèles moléculaires animés permettent d'explorer visuellement la structure tridimensionnelle (3D) de l'ADN. Le modèle d'ADN illustré (à l'extrême droite) est un modèle de remplissage d'espace, ou CPK , de la double hélice d'ADN. Les modèles moléculaires animés, tels que le type de fil, ou squelettique, montré en haut de cet article, permettent d'explorer visuellement la structure tridimensionnelle (3D) de l'ADN. Un autre type de modèle d'ADN est le modèle de remplissage d'espace, ou CPK.
La dynamique des liaisons hydrogène et l'échange de protons sont très différents de plusieurs ordres de grandeur entre les deux systèmes d'ADN entièrement hydraté et de molécules d'eau dans la glace. Ainsi, la dynamique de l'ADN est complexe, impliquant des échelles de temps de la nanoseconde et de plusieurs dizaines de picosecondes, alors que celle de la glace liquide est à l'échelle de la picoseconde, et celle de l'échange de protons dans la glace est à l'échelle de la milliseconde. Les taux d'échange de protons dans l'ADN et les protéines attachées peuvent varier de la picoseconde à la nanoseconde, des minutes ou des années, selon les emplacements exacts des protons échangés dans les grands biopolymères.
Une simple « vibration » d' oscillateur harmonique n'est qu'une représentation dynamique simplifiée des vibrations longitudinales des hélices entrelacées d'ADN qui se sont avérées être anharmoniques plutôt qu'harmoniques, comme cela est souvent supposé dans les simulations dynamiques quantiques de l'ADN.
Structure de l'ADN
La structure de l' ADN présente une variété de formes, à la fois double brin et simple brin. Les propriétés mécaniques de l'ADN, qui sont directement liées à sa structure, sont un problème important pour les cellules . Chaque processus qui lie ou lit l' ADN est capable d'utiliser ou de modifier les propriétés mécaniques de l'ADN à des fins de reconnaissance, de conditionnement et de modification. La longueur extrême (un chromosome peut contenir un brin d'ADN de 10 cm de long), la rigidité relative et la structure hélicoïdale de l'ADN ont conduit à l' évolution d' histones et d' enzymes telles que les topoisomérases et les hélicases pour gérer l'ADN d'une cellule. Les propriétés de l'ADN sont étroitement liées à sa structure moléculaire et à sa séquence, en particulier la faiblesse des liaisons hydrogène et des interactions électroniques qui maintiennent les brins d'ADN ensemble par rapport à la force des liaisons au sein de chaque brin.
Les méthodes expérimentales qui peuvent mesurer directement les propriétés mécaniques de l'ADN sont relativement nouvelles, et la visualisation à haute résolution en solution est souvent difficile. Néanmoins, les scientifiques ont découvert une grande quantité de données sur les propriétés mécaniques de ce polymère, et les implications des propriétés mécaniques de l'ADN sur les processus cellulaires sont un sujet de recherche en cours.
L'ADN trouvé dans de nombreuses cellules peut être de longueur macroscopique : quelques centimètres de long pour chaque chromosome humain. Par conséquent, les cellules doivent compacter ou emballer l' ADN pour le transporter en elles. Chez les eucaryotes, cela est porté par des protéines en forme de bobine appelées histones , autour desquelles l'ADN s'enroule. C'est le compactage ultérieur de ce complexe ADN-protéine qui produit les chromosomes mitotiques eucaryotes bien connus .
À la fin des années 1970, d'autres modèles non hélicoïdaux de la structure de l'ADN ont été brièvement considérés comme une solution potentielle aux problèmes de réplication de l'ADN dans les plasmides et la chromatine . Cependant, les modèles ont été mis de côté en faveur du modèle à double hélice en raison des avancées expérimentales ultérieures telles que la cristallographie aux rayons X des duplex d'ADN, et plus tard la particule centrale du nucléosome , et la découverte des topoisomérases . De tels modèles non à double hélice ne sont actuellement pas acceptés par la communauté scientifique traditionnelle.
Détermination de la structure de l'ADN à l'aide de la modélisation moléculaire et des modèles de rayons X
Une fois que l'ADN a été séparé et purifié par des méthodes biochimiques standard, on a un échantillon dans un bocal un peu comme dans la figure en haut de cet article. Vous trouverez ci-dessous les principales étapes impliquées dans la génération d'informations structurelles à partir d'études de diffraction des rayons X de fibres d'ADN orientées qui sont tirées de l'échantillon d'ADN hydraté à l'aide de modèles moléculaires d'ADN combinés à une analyse cristallographique et mathématique des modèles de rayons X .
Modèles de réseau paracristallin de structures d'ADN-B
Un réseau paracristallin , ou paracrystal, est un réseau moléculaire ou atomique avec des quantités importantes (par exemple, supérieures à quelques pour cent) de désordre partiel des arrangements moléculaires. Les cas limites du modèle paracrystal sont les nanostructures , telles que les verres , les liquides , etc., qui peuvent ne posséder qu'un ordre local et aucun ordre global. Un exemple simple d'un réseau paracristallin est montré dans la figure suivante pour un verre de silice :
Les cristaux liquides ont également des structures paracristallines plutôt que cristallines .
L'ADN-B hautement hydraté se produit naturellement dans les cellules vivantes dans un tel état paracristallin, qui est dynamique malgré la double hélice d'ADN relativement rigide stabilisée par des liaisons hydrogène parallèles entre les paires de bases de nucléotides dans les deux chaînes d'ADN hélicoïdales complémentaires (voir figures ). Par souci de simplicité, la plupart des modèles moléculaires d'ADN omettent à la fois l'eau et les ions liés dynamiquement à l'ADN-B, et sont donc moins utiles pour comprendre les comportements dynamiques de l'ADN-B in vivo . L'analyse physique et mathématique des données radiographiques et spectroscopiques de l'ADN-B paracristallin est donc bien plus complexe que celle des diagrammes de diffraction des rayons X de l'ADN-A cristallin. Le modèle paracrystal est également important pour les applications technologiques de l'ADN telles que la nanotechnologie de l'ADN . De nouvelles méthodes qui combinent la diffraction des rayons X de l'ADN avec la microscopie aux rayons X dans des cellules vivantes hydratées sont également en cours de développement.
Applications génomiques et biotechnologiques de la modélisation moléculaire de l'ADN

Il existe diverses utilisations de la modélisation moléculaire de l'ADN dans les applications de recherche en génomique et en biotechnologie, de la réparation de l'ADN à la PCR et aux nanostructures d'ADN . Des matrices de jonction d'ADN bidimensionnelles ont été visualisées par microscopie à force atomique .
La modélisation moléculaire de l'ADN a diverses utilisations en génomique et en biotechnologie , avec des applications de recherche allant de la réparation de l'ADN à la PCR et aux nanostructures d'ADN. Ceux-ci incluent des modèles moléculaires informatiques de molécules aussi variées que l'ARN polymérase, un E. coli, un modèle de primase d'ADN bactérien suggérant une dynamique très complexe aux interfaces entre les enzymes et le modèle d'ADN, et des modèles moléculaires de l'interaction chimique mutagène de puissantes molécules cancérigènes. avec l'ADN. Ceux-ci sont tous représentés dans la galerie ci-dessous.
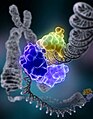
Les applications technologiques comprennent une biopuce à ADN et des nanostructures d'ADN conçues pour le calcul de l'ADN et d'autres applications dynamiques de la nanotechnologie de l' ADN . L'image à droite représente des nanostructures d'ADN auto-assemblées. La structure de « tuile » d'ADN dans cette image se compose de quatre jonctions ramifiées orientées à des angles de 90°. Chaque tuile se compose de neuf oligonucléotides d'ADN comme indiqué ; ces tuiles servent de "bloc de construction" principal pour l'assemblage des nanogrilles d'ADN montrées dans la micrographie AFM.
L'ADN quadruplex peut être impliqué dans certains cancers. Des images d'ADN quadruplex sont dans la galerie ci-dessous.
Galerie de modèles d'ADN
Modèle d'ADN de 15 m de long, Naturalis Biodiversity Center
Voir également
- G-quadruplex
- Cristallographie
- Treillis de cristal
- Base de données sur les propriétés des dinucléotides ( DiProDB ), conçue pour collecter et analyser les traits thermodynamiques, structurels et autres des dinucléotides
- Microscopie à rayons X
- diffusion des rayons X
- Diffusion des neutrons
- Séquence d'acide nucléique
- Dichroïsme circulaire vibratoire (VCD)
- Spectroscopie Raman -microscopie et spectroscopie Raman anti-Stokes cohérente (CARS)
- Sir Lawrence Bragg , FRS
- Comparatif des logiciels de simulation d'acide nucléique
- AMBRE
- CHARMM
- Ormeau (mécanique moléculaire)
- Logiciel de visualisation Sirius
- CMQ@Accueil
- Spectroscopie RMN (FT-RMN)
- Microscopie d' imagerie RMN
- Spectroscopie micro-ondes
- FT- infrarouge (IR)
- FT- spectroscopie proche infrarouge (NIR)
- Imagerie spectrale , l' imagerie hyperspectral , imagerie chimique
- Spectroscopie de corrélation de fluorescence
- Spectroscopie de corrélation croisée de fluorescence et transfert d'énergie de résonance de Förster (FRET)
- Microscopie confocale
- Modèles obsolètes de la structure de l'ADN
Les références
Lectures complémentaires
- Applications de nouvelles techniques aux aliments diététiques, à la biotechnologie médicale et agricole . (Juin 2004) IC Baianu, PR Lozano, VI Prisecaru et HC Lin., q-bio/0406047.
- F. Bessel, Untersuchung des Theils der planetarischen Störungen , Berlin Abhandlungen (1824), article 14.
- Sir Lawrence Bragg, FRS. L'état cristallin, une enquête générale. Londres : G. Bells and Sons, Ltd., vol. 1 et 2., 1966., 2024 pages.
- Cantor, CR et Schimmel, PR Biophysical Chemistry, Parties I et II ., San Franscisco : WH Freeman and Co. 1980. 1800 pages.
- Voet, D. et JG Voet. Biochimie , 2e édition, New York, Toronto, Singapour : John Wiley & Sons, Inc., 1995, ISBN 0-471-58651-X ., 1361 pages.
- Watson, GN Un traité sur la théorie des fonctions de Bessel ., (1995) Cambridge University Press. ISBN 0-521-48391-3 .
- Watson, James D. Biologie moléculaire du gène. New York et Amsterdam : WA Benjamin, Inc. 1965., 494 pages.
- Wentworth, WE Chimie Physique. Un cours court. , Malden (Mass.) : Blackwell Science, Inc. 2000.
- Herbert R. Wilson, FRS. Diffraction des rayons X par les protéines, Nucleic Acids and Viruses ., Londres : Edward Arnold (Publishers) Ltd. 1966.
- Kurt Wuthrich. RMN des protéines et des acides nucléiques. , New York, Brisbane, Chicester, Toronto, Singapour : J. Wiley & Sons. 1986., 292 pages.
- Hallin PF, David Ussery D (2004). « Base de données CBS Genome Atlas : Un stockage dynamique pour les résultats bioinformatiques et les données de séquence d'ADN » . Bioinformatique . 20 (18) : 3682-6. doi : 10.1093/bioinformatique/bth423 . PMID 15256401 .
- Zhang CT, Zhang R, Ou HY (2003). « La base de données courbe Z : une représentation graphique des séquences du génome » . Bioinformatique . 19 (5) : 593-599. doi : 10.1093/bioinformatique/btg041 . PMID 12651717 .
Liens externes
- DNA the Double Helix Game Extrait du site officiel du Prix Nobel
- MDDNA : Bioinformatique structurale de l'ADN
- Double Helix 1953-2003 Centre national d'enseignement en biotechnologie
- DNAlive : une interface web pour calculer les propriétés physiques de l'ADN . Permet également la réticulation des résultats avec le navigateur de génome UCSC et la dynamique de l'ADN.
- Plus de détails sur l'analyse mathématique et moléculaire de la structure de l'ADN sur la base des données radiographiques
- Fonctions de Bessel correspondant aux transformées de Fourier d'hélices atomiques ou moléculaires.
- aperçu des principes STM/AFM/SNOM avec des vidéos éducatives
Bases de données pour les modèles et séquences moléculaires d'ADN
- Diffraction des rayons X
- ID NDB : UD0017 Base de données
- Atlas de rayons X -base de données
- Fichiers PDB de coordonnées pour les structures d'acides nucléiques à partir de la diffraction des rayons X par des cristaux NA (incl. ADN)
- Facteurs de structure fichiers téléchargeables au format CIF
- Diffusion des neutrons
- Source de neutrons ISIS : source de neutrons pulsés ISIS : un centre mondial pour la science avec des neutrons et des muons à Harwell, près d'Oxford, au Royaume-Uni.
- Microscopie à rayons X
- Microscopie électronique
- Bases de données RMN
- Atlas RMN - base de données
- mmcif fichiers de coordonnées téléchargeables d'acides nucléiques en solution à partir de données RMN 2D-FT
- Fichiers de contraintes RMN pour les AN au format PDB
- Bases de données génomiques et structurelles
- Base de données CBS Genome Atlas — contient des exemples d'asymétries de base.
- La base de données de courbes en Z des génomes - un outil de visualisation et d'analyse en 3 dimensions des génomes .
- Modèles moléculaires d'ADN et d'autres acides nucléiques : fichiers de coordonnées de modèles de structure moléculaire d'acides nucléiques aux formats PDB et CIF
- Microscopie à force atomique