Morphée - Morpheein
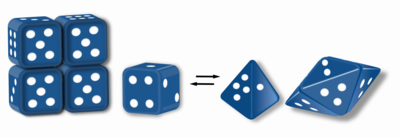
Les morphéines sont des protéines qui peuvent former deux ou plusieurs homo- oligomères différents (formes de morphée), mais doivent se séparer et changer de forme pour passer d'une forme à l'autre. La forme alternative peut se réassembler en un oligomère différent. La forme de la sous-unité dicte quel oligomère est formé. Chaque oligomère a un nombre fini de sous-unités ( stoechiométrie ). Les morphéines peuvent s'interconvertir entre les formes dans des conditions physiologiques et peuvent exister sous forme d'équilibre de différents oligomères. Ces oligomères sont physiologiquement pertinents et ne sont pas des protéines mal repliées ; cela distingue les morphéines des prions et de l' amyloïde . Les différents oligomères ont des fonctionnalités distinctes. L'interconversion des formes morphiniques peut être une base structurelle pour la régulation allostérique . Une mutation qui modifie l'équilibre normal des formes morphiniques peut servir de base à une maladie conformationnelle . Les caractéristiques des morphéines peuvent être exploitées pour la découverte de médicaments . L'image en dés (figure 1) représente un équilibre de morphée contenant deux formes monomères différentes qui dictent l'assemblage à un tétramère ou à un pentamère. La seule protéine qui est établie pour fonctionner comme une morphéine est la porphobilinogène synthase, bien qu'il y ait des suggestions dans la littérature que d'autres protéines peuvent fonctionner comme des morphéines (pour plus d'informations, voir "Tableau des morphéines putatives" ci-dessous).
Implications pour la découverte de médicaments
Les différences de conformation entre les sous-unités de différents oligomères et les différences fonctionnelles associées d'une morphéine constituent un point de départ pour la découverte de médicaments. La fonction des protéines dépend de la forme oligomérique ; par conséquent, la fonction de la protéine peut être régulée en déplaçant l'équilibre des formes. Un composé à petite molécule peut déplacer l'équilibre soit en bloquant soit en favorisant la formation de l'un des oligomères. L'équilibre peut être déplacé en utilisant une petite molécule qui a une affinité de liaison préférentielle pour une seule des formes alternatives de morphéine. Un inhibiteur de la porphobilinogène synthase avec ce mécanisme d'action a été documenté.
Implications pour la régulation allostérique
Le modèle de régulation allostérique de la morphéine présente des similitudes et des différences par rapport aux autres modèles. Le modèle concerté (le modèle Monod, Wyman et Changeux (MWC)) de la régulation allostérique exige que toutes les sous-unités soient dans la même conformation ou état au sein d'un oligomère comme le modèle de la morphée. Cependant, ni ce modèle ni le modèle séquentiel (modèle de Koshland, Nemethy et Filmer) ne prennent en compte le fait que la protéine peut se dissocier pour s'interconvertir entre les oligomères.
Implications pour l'enseignement des relations structure-fonction des protéines
Il est généralement enseigné qu'une séquence d'acides aminés donnée n'aura qu'une seule structure quaternaire physiologiquement pertinente (native) ; les morpheeins remettent en cause ce concept. Le modèle de la morphéine ne nécessite pas de changements bruts dans le repliement de la protéine de base. Les différences de conformation qui accompagnent la conversion entre les oligomères peuvent être similaires aux mouvements protéiques nécessaires au fonctionnement de certaines protéines. Le modèle de la morphée met en évidence l'importance de la flexibilité conformationnelle pour la fonctionnalité des protéines et offre une explication potentielle pour les protéines présentant une cinétique non Michaelis-Menten , une hystérésis et/ou une activité spécifique dépendante de la concentration des protéines .
Implications pour comprendre la base structurelle de la maladie
Le terme « maladie conformationnelle » englobe généralement des mutations qui entraînent des protéines mal repliées qui s'agrègent, telles que les maladies d'Alzheimer et de Creutzfeldt-Jakob. À la lumière de la découverte des morphéines, cependant, cette définition pourrait être élargie pour inclure des mutations qui modifient l'équilibre des formes oligomères alternatives d'une protéine. Un exemple d'une telle maladie conformationnelle est la porphyrie ALAD , qui résulte d'une mutation de la porphobilinogène synthase qui provoque un changement dans son équilibre de morpheeine.
Tableau des protéines dont le comportement publié est cohérent avec celui d'une morphéine
| Protéine | Exemple d'espèces | Numéro EC | Numero CAS | Oligomères alternatifs | Preuve |
|---|---|---|---|---|---|
| Acétyl-CoA carboxylase -1 | Gallus domestique | CE 6.4.1.2 | 9023-93-2 | dimère inactif, dimère actif, plus grand | Les molécules effectrices ont un impact sur la multimérisation, les fonctions de travail au noir multiple/ protéique |
| α-acétylgalactosaminidase | Bos taureau | CE 4.3.2.2 | 9027-81-0 | monomère inactif, tétramère actif | La liaison/le renouvellement du substrat a un impact sur la multimérisation, Activité spécifique dépendante de la concentration en protéines, Différents assemblages ont des activités différentes, Formes oligomères distinctes sur le plan de la conformation. |
| Adénylosuccinate lyase | Bacillus subtilis | CE 4.3.2.2 | 9027-81-0 | monomère, dimère, trimère, tétramère | Des mutations modifient l'équilibre des oligomères, Paramètres cinétiques dépendants des oligomères, Poids moléculaire dépendant de la concentration en protéines |
| Aristolochène synthase | Pénicillium roqueforti | CE 4.2.3.9 | 94185-89-4 | monomère, ordre supérieur | Activité spécifique dépendante de la concentration en protéines |
| L- Asparaginase | Leptosphaeria michotii | CE 3.5.1.1 | 9015-68-3 | dimère, tétramère, octamère inactif | La liaison/le retournement du substrat a un impact sur la multimérisation |
| Aspartokinase | Escherichia coli | CE 2.7.2.4 & CE 1.1.1.3 | 9012-50-4 | monomère, dimère, tétramère | Fonctions de clair de lune multiples/ protéiques , Formes oligomériques distinctes du point de vue conformationnel |
| ATPase du transporteur ABCA1 | Homo sapiens | dimère, tétramère | La liaison/le retournement du substrat a un impact sur la multimérisation | ||
| Biotine—(acétyl-CoA-carboxylase) ligase holoenzyme synthétase | Escherichia coli | CE 6.3.4.15 | 37340-95-7 | monomère, dimère | Fonctions de clair de lune multiples/ protéines , différents assemblages ont des activités différentes |
| Chorismate mutase | Escherichia coli | CE 5.4.99.5 | 9068-30-8 | dimère, trimère, hexamère | Formes oligomères conformationnellement distinctes |
| Citrate synthase | Escherichia coli | CE 2.3.3.1 | 9027-96-7 | monomère, dimère, trimère, tétramère, pentamère, hexamère, dodécamère | La liaison/le renouvellement du substrat a un impact sur la multimérisation, Équilibre caractérisé des oligomères, Activité spécifique dépendante de la concentration en protéines, Équilibre oligomère pH-dépendant |
| Cyanovirine-N | Nostoc ellipsosporum | 918555-82-5 | monomère et dimère à domaine échangé | Équilibre caractérisé des oligomères, Formes oligomères distinctes de conformation | |
| 3-oxoacide CoA-transférase | Sus scrofa domestique | CE 2.8.3.5 | 9027-43-4 | dimère, tétramère | Oligomères séparables par chromatographie, le substrat pourrait de préférence stabiliser une forme |
| Cystathionine bêta-synthase | Homo sapiens | CE 4.2.1.22 | 9023-99-8 | multiple - va du dimère au 16-mer | Les molécules effectrices ont un impact sur la multimérisation, Les mutations modifient l'équilibre des oligomères, Différents assemblages ont des activités différentes, Des mutations causant des maladies sur des sites éloignés du site actif |
| D-aminoacide oxydase | CE 1.4.3.3 | 9000-88-8 | monomères, dimères, oligomères d'ordre supérieur | Paramètres cinétiques dépendants des oligomères | |
| Dihydrolipoamide déshydrogénase | Sus scrofa domestique | CE 1.8.1.4 | 9001-18-7 | monomère, deux formes dimères différentes, tétramère | Fonctions de clair de lune multiples/ protéiques , Différents assemblages ont des activités différentes, Équilibre oligomérique dépendant du pH, Formes oligomériques distinctes du point de vue conformationnel |
| Dopamine bêta-monooxygénase | Bos taureau | CE 1.14.17.1 | 9013-38-1 | dimères, tétramères | Molécules effectrices impact multimérisation, Equilibre caractérisé des oligomères, Paramètres cinétiques dépendant de l'oligomère |
| Géranylgéranyl pyrophosphate synthase / Farnésyltransférase | Homo sapiens | CE 2.5.1.29 | 9032-58-0 | hexamère, octamère | Les molécules effectrices impactent la multimérisation |
| GDP-mannose 6-déshydrogénase | Pseudomonas aeruginosa | CE 1.1.1.132 | 37250-63-8 | trimère, 2 tétramères et hexamère | Activité spécifique dépendante de la concentration protéique, Hystérésis cinétique |
| Glutamate déshydrogénase | Bos taureau | CE 1.4.1.2 | 9001-46-1 | hexamères actifs et inactifs, ordre supérieur | Molécules effectrices impact multimérisation, Equilibre caractérisé des oligomères |
| racémase de glutamate | Mycobacterium tuberculosis, Escherichia coli, Bacillus subtilis, Aquifex pyrophilus | CE 5.1.1.3 | 9024-08-02 | monomère, 2 dimères, tétramère | Fonctions de clair de lune multiples/ protéiques , Équilibre caractérisé des oligomères, Formes oligomères distinctes du point de vue conformationnel |
| Glycéraldéhyde-3-phosphate déshydrogénase | Oryctolagus cuniculas, Sus scrofa domestica | CE 1.2.1.12 | 9001-50-7 | monomère, dimère, tétramère Équilibre caractérisé des oligomères, Différents assemblages ont des activités différentes | |
| Glycérol kinase | Escherichia coli | CE 2.7.1.30 | 9030-66-4 | monomère et 2 tétramères | Équilibre caractérisé des oligomères, Formes oligomères distinctes de conformation, Fonctions effectrices en empêchant le mouvement du domaine |
| VIH- intégrase | Virus de l'immunodéficience humaine-1 | CE 2.7.7.- | monomère, dimère, tétramère, ordre supérieur | Les molécules effectrices ont un impact sur la multimérisation, Fonctions de clair de lune multiples/ protéiques , Différents assemblages ont des activités différentes | |
| HPr-Kinase/phosphatase | Bacillus subtilis, Lactobacillus casei, Mycoplasma pneumoniae, Staphylococcus xylosus | CE 2.7.1.- / CE 3.1.3.- | 9026-43-1 | monomères, dimères, trimères, hexamères | Les molécules effectrices ont un impact sur la multimérisation, Fonctions au noir de la lune multiples/ protéiques , Différents assemblages ont des activités différentes, Équilibre oligomérique dépendant du pH |
| Lactate déshydrogénase | Bacillus stearothermophilus | CE 1.1.1.27 | 9001-60-9 | 2 dimères, tétramère | Molécules effectrices impactant la multimérisation, Équilibre caractérisé des oligomères, Activité spécifique dépendante de la concentration de la protéine, Des mutations modifient l'équilibre des oligomères, Paramètres cinétiques dépendant de l'oligomère, Formes oligomères distinctes de conformation |
| Lon protéase | Escherichia coli, Mycobacterium smegmatis | CE 3.4.21.53 | 79818-35-2 | monomère, dimère, trimère, tétramère | Les molécules effectrices ont un impact sur la multimérisation, la liaison/le renouvellement du substrat a un impact sur la multimérisation, l'activité spécifique dépendante de la concentration de protéines, l'hystérésis cinétique |
| NAD(P)+ mitochondrial Enzyme malique/ malate déshydrogénase (oxaloacétate-décarboxylant) (NADP+) | Homo sapiens | CE 1.1.1.40 | 9028-47-1 | monomère, 2 dimères, tétramère | Les molécules effectrices impactent la multimérisation, Les mutations modifient l'équilibre des oligomères, L'hystérésis cinétique, |
| Peroxiredoxines | Salmonelle typhimurium | CE 1.6.4.- & CE 1.11.1.15 | 207137-51-7 | 2 dimères, décamères | Formes oligomériques distinctes du point de vue conformationnel, Différents assemblages ont des activités différentes |
| Phénylalanine hydroxylase | Homo sapiens | CE 1.14.16.1 | 9029-73-6 | tétramère de haute activité, tétramère de faible activité | La liaison/le renouvellement du substrat a un impact sur la multimérisation, Formes oligomériques distinctes du point de vue conformationnel |
| Phosphoénolpyruvate carboxylase | Escherichia coli, Zea mays | CE 4.1.1.31 | 9067-77-0 | dimère inactif, tétramère actif | Multimérisation d'impact de molécules effectrices, Équilibre caractérisé des oligomères, Hystérésis cinétique, Formes oligomères distinctes par conformation |
| Phosphofructokinase | Bacillus stearothermophilus, Thermus thermophilus | CE 2.7.1.11 | 9001-80-3 | dimère inactif, tétramère actif | Molécules effectrices impact multimérisation, Equilibre caractérisé des oligomères |
| Polyphénol oxydase | Agaricus bisporus, Malus domestica, Lactuca sativa L. | CE 1.10.3.1 | 9002-10-2 | monomère, trimère, tétramère, octamère, dodécamère | Fonctions de moonlighting multiples/ protéiques , Multimérisation d'impacts de liaison/renouvellement du substrat, Différents assemblages ont des activités différentes, Hystérésis cinétique |
| Porphobilinogène synthase | Drosophila melanogaster, Danio rerio | CE 4.2.1.24 | 9036-37-7 | dimère, hexamère, octamère | PBGS est le prototype de la morphée. |
| Pyruvate kinase | Homo sapiens | CE 2.7.1.40 | 9001-59-6 | dimères actifs et inactifs, tétramère actif, monomère, trimère, pentamère | Formes oligomères conformationnellement distinctes |
| Ribonucléase A | Bos taureau | CE 3.1.27.5 | 9901-99-4 | monomère, dimère, trimère, tétramère, hexamère, pentamère, ordre supérieur | Fonctions de clair de lune multiples/ protéiques , Différents assemblages ont des activités différentes, Formes oligomères distinctes du point de vue conformationnel |
| Ribonucléotide réductase | Mus musculus | CE 1.17.4.1 | 9047-64-7 | tétramère, hexamère | Les molécules effectrices impactent la multimérisation |
| S-adénosyl-L-homocystéine hydrolase | Dictyostelium discoideum | CE 3.3.1.1 | 9025-54-1 | tétramère et autres | Les molécules effectrices impactent la multimérisation |
| Thréonine déshydratase biodégradante / thréonine ammoniac-lyase | Escherichia coli | CE 4.3.1.19 | 774231-81-1 | 2 monomères, 2 tétramères | Les molécules effectrices impactent la multimérisation, Equilibre caractérisé des oligomères, Différents assemblages ont des activités différentes |
| - Tryptase | Homo sapiens | CE 3.4.21.59 | 97501-93-4 | monomères actifs et inactifs, tétramères actifs et inactifs | Activité spécifique dépendante de la concentration en protéines, Equilibre caractérisé des oligomères |
| Facteur de nécrose tumorale alpha | Homo sapiens | 94948-61-5 | monomère, dimère, trimère | Différentes assemblées ont différentes activités | |
| Uracil phosphoribosyltransférase | Escherichia coli | CE 2.4.2.9 | 9030-24-4 | trimère, pentamère | Les molécules effectrices impactent la multimérisation, La liaison/le renouvellement du substrat impacte la multimérisation, Différents assemblages ont des activités différentes |
Les références
- ^ A b c d e Jaffe, Eileen K. (2005). « Morpheeins - un nouveau paradigme structurel pour la régulation allostérique ». Tendances en sciences biochimiques . 30 (9) : 490-7. doi : 10.1016/j.tibs.2005.07.003 . PMID 16023348 .
- ^ A b c Breinig, Sabine; Kervinen, Jukka ; Stith, Linda ; Wasson, Andrew S; Fairman, Robert; Wlodawer, Alexandre ; Zdanov, Alexandre ; Jaffe, Eileen K (2003). « Contrôle de la biosynthèse du tétrapyrrole par des formes quaternaires alternatives de la porphobilinogène synthase ». Biologie structurale de la nature . 10 (9) : 757–63. doi : 10.1038/nsb963 . PMID 12897770 . S2CID 24188785 .
- ^ A b c Lawrence, Sarah H .; Ramirez, Ursula D. ; Tang, Lei ; Fazliyez, Farit; Kundrat, Lenka; Markham, George D.; Jaffe, Eileen K. (2008). "Le changement de forme mène à la découverte de médicaments allostériques à petites molécules" . Chimie & Biologie . 15 (6) : 586–96. doi : 10.1016/j.chembiol.2008.04.012 . PMC 2703447 . PMID 18559269 .
- ^ A b c Selwood, Trevor; Jaffe, Eileen K. (2012). "Les homo-oligomères à dissociation dynamique et le contrôle de la fonction protéique" . Archives de biochimie et biophysique . 519 (2) : 131–43. doi : 10.1016/j.abb.2011.11.020 . PMC 3298769 . PMID 22182754 .
- ^ un b Jaffe, Eileen K.; Stith, Linda (2007). "ALAD La porphyrie est une maladie conformationnelle" . Le Journal américain de génétique humaine . 80 (2) : 329-37. doi : 10.1086/511444 . PMC 1785348 . PMID 17236137 .
- ^ Jaffe, Eileen K. (2010). "Morpheeins - Une nouvelle voie pour la découverte de médicaments allostériques" . Le Journal des Actes de la Conférence Ouverte . 1 : 1–6. doi : 10.2174/2210289201001010001 . PMC 3107518 . PMID 21643557 .
- ^ Tang, L.; Stith, L ; Jaffe, EK (2005). "Interconversion induite par le substrat d'isoformes de structure quaternaire de protéines" . Journal de chimie biologique . 280 (16) : 15786-93. doi : 10.1074/jbc.M500218200 . PMID 15710608 .
- ^ Jaffe, Eileen K.; Laurent, Sarah H. (2012). « Allostery et l'oligomérisation dynamique de la porphobilinogène synthase » . Archives de biochimie et biophysique . 519 (2) : 144-53. doi : 10.1016/j.abb.2011.10.010 . PMC 3291741 . PMID 22037356 .
- ^ un b Lawrence, Sarah H.; Jaffe, Eileen K. (2008). "Élargir les concepts dans les relations structure-fonction des protéines et la cinétique enzymatique: Enseignement à l'aide de morpheeins" . Enseignement de la biochimie et de la biologie moléculaire . 36 (4) : 274-283. doi : 10.1002/bmb.20211 . PMC 2575429 . PMID 19578473 .
- ^ un b Monod, Jacques; Changeux, Jean-Pierre ; Jacob, François (1963). « Protéines allostériques et systèmes de contrôle cellulaire ». Journal de biologie moléculaire . 6 (4) : 306-29. doi : 10.1016/S0022-2836(63)80091-1 . PMID 13936070 .
- ^ un b Monod, Jacques; Wyman, Jeffries ; Changeux, Jean-Pierre (1965). « Sur la nature des transitions allostériques : Un modèle plausible ». Journal de biologie moléculaire . 12 : 88-118. doi : 10.1016/S0022-2836(65)80285-6 . PMID 14343300 .
- ^ Koshland, DE (1970). "7 La base moléculaire de la régulation des enzymes" . Les Enzymes Tome 1 . Les Enzymes. 1 . p. 341-396. doi : 10.1016/S1874-6047(08)60170-5 . ISBN 978-0-12-122701-2.
- ^ Koshland, DE; Nemethy, G.; Filmer, D. (1966). "Comparaison des données expérimentales de liaison et des modèles théoriques dans les protéines contenant des sous-unités". Biochimie . 5 (1) : 365-85. doi : 10.1021/bi00865a047 . PMID 5938952 .
- ^ Gerstein, Marc; Echols, Nathaniel (2004). « Explorer la gamme de flexibilité des protéines, du point de vue de la protéomique structurelle ». Opinion actuelle en biologie chimique . 8 (1) : 14-9. doi : 10.1016/j.cbpa.2003.12.006 . PMID 15036151 .
- ^ Carrell, Robin W; Lomas, David A (1997). "Maladie conformationnelle". La Lancette . 350 (9071) : 134-8. doi : 10.1016/S0140-6736(97)02073-4 . PMID 9228977 . S2CID 39124185 .
- ^ un b Boone, AN; Brownsey, RW; Elliott, JE ; Kulpa, JE ; Lee, WM (2006). "Régulation de l'acétyl-CoA carboxylase". Transactions de la société biochimique . 34 (2) : 223-7. doi : 10.1042/BST20060223 . PMID 16545081 .
- ^ Shen, Yang; Volrath, Sandra L.; Weatherly, Stéphanie C.; Elich, Tedd D.; Tong, Liang (2004). "Un mécanisme pour l'inhibition puissante de l'acétyl-coenzyme eucaryote une carboxylase par Soraphen A, un produit naturel polykétide macrocyclique" . Cellule moléculaire . 16 (6) : 881–91. doi : 10.1016/j.molcel.2004.11.034 . PMID 15610732 .
- ^ A b c Weissmann, Bernard; Wang, Ching-Te (1971). « Association-dissociation et cinétique anormale de l'alpha-acétylgalactosaminidase bovine ». Biochimie . 10 (6) : 1067-1072. doi : 10.1021/bi00782a021 . PMID 5550813 .
- ^ A b c Weissmann, Bernard; Hinrichsen, Dorotea F. (1969). « α-acétylgalactosaminidase de mammifère. Occurrence, purification partielle et action sur les liaisons dans les mucines sous-maxillaires ». Biochimie . 8 (5) : 2034-2043. doi : 10.1021/bi00833a038 . PMID 5785223 .
- ^ De Zoysa Ariyananda, Lushanti; Colman, Roberta F. (2008). « Évaluation des types d'interactions dans l'association de sous-unités dans Bacillus subtilis Adenylosuccinate Lyase ». Biochimie . 47 (9) : 2923-34. doi : 10.1021/bi701400c . PMID 18237141 .
- ^ A b c Palenchar, Jennifer Brosius; Colman, Roberta F. (2003). « Caractérisation d'une lyase d'adénylosuccinate mutante de Bacillus subtilis équivalente à une enzyme mutante trouvée dans la carence en adénylosuccinate lyase humaine : l'asparagine 276 joue un rôle structurel important ». Biochimie . 42 (7) : 1831-1841. doi : 10.1021/bi020640+ . PMID 12590570 .
- ^ Hohn, Thomas M.; Plattner, Ronald D. (1989). « Purification et caractérisation de la sesquiterpène cyclase aristolochène synthase de Penicillium roqueforti ». Archives de biochimie et biophysique . 272 (1) : 137-43. doi : 10.1016/0003-9861(89)90204-X . PMID 2544140 .
- ^ Caruthers, JM; Kang, moi ; Rynkiewicz, MJ; Canne, DE; Christianson, DW (2000). "Détermination de la structure cristalline de la synthèse d'aristolochène à partir du moule de fromage bleu, Penicillium roqueforti" . Journal de chimie biologique . 275 (33): 25533-9. doi : 10.1074/jbc.M000433200 . PMID 10825154 .
- ^ Jerebzoff-Quintin, Simonne; Jerebzoff, Stéphane (1985). "L'activité L-asparaginase dans Leptosphaeria michotii. Isolement et propriétés de deux formes de l'enzyme". Physiologie plantaire . 64 : 74-80. doi : 10.1111/j.1399-3054.1985.tb01215.x .
- ^ Yun, Mi-Kyung; Nourrice, Amanda; Blanc, Stephen W. ; Roche, Charles O. ; Heath, Richard J. (2007). "Structure cristalline et régulation allostérique de la l-asparaginase I cytoplasmique d'Escherichia coli" . Journal de biologie moléculaire . 369 (3) : 794-811. doi : 10.1016/j.jmb.2007.03.061 . PMC 1991333 . PMID 17451745 .
- ^ Garel, J.-R. (1980). "Pliage séquentiel d'une protéine allostérique bifonctionnelle" . Actes de l'Académie nationale des sciences . 77 (6) : 3379-3383. Bibcode : 1980PNAS ... 77.3379G . doi : 10.1073/pnas.77.6.3379 . JSTOR 8892 . PMC 349619 . PMID 6774337 .
- ^ un b Kotaka, M.; Ren, J.; Lockyer, M. ; Hawkins, AR ; Stammers, DK (2006). "Structures de l'aspartokinase III d'Escherichia coli à l'état R et T : MÉCANISMES DE LA TRANSITION ALLOSTÉRIQUE ET DE L'INHIBITION PAR LA LYSINE" . Journal de chimie biologique . 281 (42) : 31544-52. doi : 10.1074/jbc.M605886200 . PMID 16905770 .
- ^ Ogilvie, JW; Vickers, LP; Clark, RB; Jones, MM (1975). "Aspartokinase I-homosérine déshydrogénase I d'Escherichia coli K12 (lambda). Activation par des cations monovalents et une analyse de l'effet du complexe adénosine triphosphate-ion magnésium sur ce processus d'activation" . Le Journal de Chimie Biologique . 250 (4) : 1242-1250. doi : 10.1016/S0021-9258 (19) 41805-X . PMID 163250 .
- ^ un b Trompier, D.; Alibert, M; Davanture, S; Hamon, Y ; Pierres, M; Chimini, G (2006). "La transition des dimères aux formes oligomériques supérieures se produit pendant le cycle ATPase du transporteur ABCA1" . Journal de chimie biologique . 281 (29) : 20283-90. doi : 10.1074/jbc.M601072200 . PMID 16709568 .
- ^ un b Eisenstein, Edward; Beckett, Dorothée (1999). « Dimérisation du répresseur de biotine d'Escherichiacoli : fonction de corépresseur dans l'assemblage de protéines ». Biochimie . 38 (40) : 13077-84. doi : 10.1021/bi991241q . PMID 10529178 .
- ^ Streaker, Emily D.; Beckett, Dorothée (1998). « Couplage de la liaison d'ADN spécifique au site à la dimérisation des protéines dans l'assemblage du complexe répresseur de biotine-opérateur de biotine ». Biochimie . 37 (9) : 3210–9. doi : 10.1021/bi9715019 . PMID 9485476 .
- ^ Vamvaca, Katherina; Butz, Maren ; Walter, Kai U.; Taylor, Sean V. ; Hilvert, Donald (2005). "Optimisation simultanée de l'activité enzymatique et de la structure quaternaire par évolution dirigée" . Sciences des protéines . 14 (8) : 2103–14. doi : 10.1110/ps.051431605 . PMC 2279322 . PMID 15987889 .
- ^ A b c d e Tong, EK; Duckworth, Harry W. (1975). « Structure quaternaire de citrate synthase d'Escherichia coli K 12 ». Biochimie . 14 (2) : 235-41. doi : 10.1021/bi00673a007 . PMID 1091285 .
- ^ Bewley, Carole A.; Gustafson, Kirk R.; Boyd, Michael R.; Covell, David G.; Bax, annonce ; Clore, G. Marius ; Gronenborn, Angela M. (1998). « Structure de la solution de la cyanovirine-N, une puissante protéine inactivant le VIH ». Biologie structurale de la nature . 5 (7) : 571-8. doi : 10.1038/828 . PMID 9665171 . S2CID 11367037 .
- ^ Yang, Fan; Bewley, Carole A; Louis, John M; Gustafson, Kirk R; Boyd, Michael R; Gronenborn, Angela M; Clore, G. Marius ; Wlodawer, Alexandre (1999). "La structure cristalline de la cyanovirine-N, une puissante protéine inactivant le VIH, montre un échange de domaine inattendu" . Journal de biologie moléculaire . 288 (3) : 403-12. doi : 10.1006/jmbi.1999.2693 . PMID 10329150 . S2CID 308708 .
- ^ un b Barrientos, LG; Gronenborn, AM (2005). « La protéine de liaison aux glucides hautement spécifique, la cyanovirine-N : structure, activité anti-VIH/Ebola et possibilités thérapeutiques ». Mini revues en chimie médicinale . 5 (1) : 21-31. doi : 10.2174/1389557053402783 . PMID 15638789 .
- ^ un b Barrientos, LG; Louis, JM; Botos, moi ; Mori, T; Han, Z; O'Keefe, BR ; Boyd, M. ; Wlodawer, A; et al. (2002). "Le dimère à échange de domaine de la cyanovirine-N est dans un état replié métastable : Réconciliation des structures aux rayons X et RMN" . Structurer . 10 (5) : 673-86. doi : 10.1016/S0969-2126(02)00758-X . PMID 12015150 .
- ^ A b c Rochet, Jean-Christophe; Brownie, Edward R.; Oikawa, Kim ; Hicks, Leslie D.; Fraser, Marie E.; James, Michael NG; Kay, Cyril M. ; Bridger, William A.; et al. (2000). "La transférase CoA de coeur de porc existe sous forme de deux formes oligomériques séparées par une grande barrière cinétique". Biochimie . 39 (37) : 11291-302. doi : 10.1021/bi0003184 . PMID 10985774 .
- ^ Franck, Nina; Kery, Vladimir ; MacLean, Kenneth N.; Kraus, Jan P. (2006). « Cystéines accessibles aux solvants dans la cystathionine β-synthase humaine : rôle crucial de la cystéine 431 dans la liaison S-adénosyl-l-méthionine ». Biochimie . 45 (36) : 11021-9. doi : 10.1021/bi060737m . PMID 16953589 .
- ^ un b Sen, Suvajit; Banerjee, Ruma (2007). "Une mutation liée à un pathogène dans le noyau catalytique de la cystathionine β-synthase humaine perturbe la régulation allostérique et permet la caractérisation cinétique d'un dimère de pleine longueur" . Biochimie . 46 (13) : 4110-6. doi : 10.1021/bi602617f . PMC 3204387 . PMID 17352495 .
- ^ Kery, Vladimir; Poneleit, Loelle; Kraus, Jan P. (1998). « Clivage de la trypsine de la cystathionine β-synthase humaine dans un noyau actif conservé de manière évolutive : conséquences structurelles et fonctionnelles ». Archives de biochimie et biophysique . 355 (2) : 222-32. doi : 10.1006/abbi.1998.0723 . PMID 9675031 .
- ^ Shan, Xiaoyin; Kruger, Warren D. (1998). « Correction des mutations CBS causant la maladie dans la levure ». Génétique de la nature . 19 (1) : 91-3. doi : 10.1038/ng0598-91 . PMID 9590298 . S2CID 47102642 .
- ^ un b Antonini, E; Brunori, M; Bruzzesi, R; Chiancone, E; Massey, V (1966). "Phénomènes d'association-dissociation de la D-aminoacide oxydase" . Le Journal de Chimie Biologique . 241 (10) : 2358-66. doi : 10.1016/S0021-9258 (18) 96629-9 . PMID 4380380 .
- ^ un b Massey, V; Curti, B; Ganther, H (1966). « Un changement de conformation dépendant de la température de la D-aminoacide oxydase et son effet sur la catalyse » . Le Journal de Chimie Biologique . 241 (10) : 2347-57. doi : 10.1016/S0021-9258 (18) 96628-7 . PMID 5911617 .
- ^ A b c d Babady, NE; Pang, Y.-P.; Elpeleg, O. ; Isaya, G. (2007). "Activité protéolytique cryptique de la dihydrolipoamide déshydrogénase" . Actes de l'Académie nationale des sciences . 104 (15) : 6158-63. Bibcode : 2007PNAS..104.6158B . doi : 10.1073/pnas.0610618104 . PMC 1851069 . PMID 17404228 .
- ^ Muiswinkel-Voetberg, H.; Visser, Jaap ; Veeger, Cornelis (1973). "Études conformationnelles sur la lipoamide déshydrogénase du cœur de porc. 1. Interconversion des formes dissociables et non dissociables" . Journal Européen de Biochimie . 33 (2) : 265-70. doi : 10.1111/j.1432-1033.1973.tb02679.x . PMID 4348439 .
- ^ Klyachko, Pays-Bas; Shchedrina, Virginie ; Efimov, AV ; Kazakov, SV; Gazarian, IG; Kristal, BS; Brown, AM (2005). « La préférence de substrat PH-dépendante de la lipoamide déshydrogénase de cœur de porc varie selon l'état oligomérique : RÉPONSE À L'ACIDIFICATION DE LA MATRICE MITOCHONDRIALE » . Journal de chimie biologique . 280 (16) : 16106–14. doi : 10.1074/jbc.M414285200 . PMID 15710613 .
- ^ Muiswinkel-Voetberg, H.; Veeger, Cornelis (1973). "Études conformationnelles sur la lipoamide déshydrogénase du cœur de porc. 2. Études spectroscopiques sur l'apoenzyme et les formes monomères et dimères" . Journal Européen de Biochimie . 33 (2) : 271–8. doi : 10.1111/j.1432-1033.1973.tb02680.x . PMID 4348440 .
- ^ A b c d Saxena, Ashima; Hensley, Preston; Osborne, James C. ; Fleming, Patrick J. (1985). "La dissociation des sous-unités dépendante du pH et l'activité catalytique de la dopamine -hydroxylase bovine" . Journal de chimie biologique . 260 (6) : 3386-92. doi : 10.1016/S0021-9258 (19) 83633-5 . PMID 3972830 .
- ^ A b c d Dhawan, S; Hensley, P; Osborne Jr, JC; Fleming, PJ (1986). "Dissociation de la sous-unité dépendante de l'adénosine 5'-diphosphate de la dopamine bêta-hydroxylase bovine" . Le Journal de Chimie Biologique . 261 (17) : 7680–4. doi : 10.1016/S0021-9258 (19)57453-1 . PMID 3711102 .
- ^ A b c d Stewart, LC; Klinman, JP (1988). « Dopamine bêta-hydroxylase des granules de chromaffine surrénale : structure et fonction ». Revue annuelle de biochimie . 57 : 551–92. doi : 10.1146/annurev.bi.57.070188.003003 . PMID 3052283 .
- ^ Kuzuguchi, T.; Morita, Y; Sagami, moi ; Sagami, H; Ogura, K (1999). "Human Geranylgeranyl Diphosphate Synthase. CLONAGE ET EXPRESSION D'ADNc" . Journal de chimie biologique . 274 (9) : 5888–94. doi : 10.1074/jbc.274.9.5888 . PMID 10026212 .
- ^ un b Kavanagh, KL; Dunford, JE; Bunkoczi, G; Russell, RG; Oppermann, U (2006). "La structure cristalline de la géranylgéranyl pyrophosphate synthase humaine révèle un nouvel arrangement hexamère et une liaison au produit inhibiteur" . Journal de chimie biologique . 281 (31) : 22004-12. doi : 10.1074/jbc.M602603200 . PMID 16698791 .
- ^ Miyagi, Y.; Matsumura, Y. ; Sagami, H. (2007). "Human Geranylgeranyl Diphosphate Synthase est un octamère en solution". Journal de biochimie . 142 (3) : 377-81. doi : 10.1093/jb/mvm144 . PMID 17646172 .
- ^ Snook, Christopher F.; Tipton, Peter A.; Beamer, Lesa J. (2003). « Structure cristalline de GDP-mannose déshydrogénase : une enzyme clé de la biosynthèse d'alginate dans P. Aeruginosa ». Biochimie . 42 (16) : 4658-68. doi : 10.1021/bi027328k . PMID 12705829 .
- ^ Roychoudhury, S; mai, tuberculose ; Gill, JF; Singh, Saskatchewan ; Feingold, DS ; Chakrabarty, AM (1989). "Purification et caractérisation de la guanosine diphospho-D-mannose déshydrogénase. Une enzyme clé dans la biosynthèse de l'alginate par Pseudomonas aeruginosa" . Le Journal de Chimie Biologique . 264 (16) : 9380-5. doi : 10.1016/S0021-9258 (18) 60542-3 . PMID 2470755 .
- ^ un b Rien, Laura E.; Gilbert, ensoleillé ; Imhoff, Rébecca ; Snook, Christophe ; Beamer, Lesa; Tipton, Peter (2002). « Allosterism et Coopérativité dans Pseudomonas aeruginosaGDP-Mannose Dehydrogenase ». Biochimie . 41 (30) : 9637-45. doi : 10.1021/bi025862m . PMID 12135385 .
- ^ un pêcheur b , Harvey F. (2006). « Complexes de glutamate déshydrogénase-ligand et leur relation avec le mécanisme de la réaction » . Avancées en enzymologie et domaines connexes de la biologie moléculaire . Avancées en enzymologie - et domaines connexes de la biologie moléculaire. 39 . p. 369-417 . doi : 10.1002/9780470122846.ch6 . ISBN 978-0-470-12284-6. PMID 4147773 .
- ^ Huang, CY; Frieden, C (1972). "Le mécanisme des changements structurels induits par le ligand dans la glutamate déshydrogénase. Études de la vitesse de dépolymérisation et d'isomérisation effectuées par les coenzymes et les nucléotides de guanine" . Le Journal de Chimie Biologique . 247 (11) : 3638-46. doi : 10.1016/S0021-9258 (19) 45188-0 . PMID 4402280 .
- ^ un b Kim, Sang Suk; Choi, I.-G.; Kim, Sung-Hou ; Yu, YG (1999). « Clonage moléculaire, expression et caractérisation d'une racémase de glutamate thermostable à partir d'une bactérie hyperthermophile, Aquifex pyrophilus ». extrêmophiles . 3 (3) : 175–83. doi : 10.1007/s007920050114 . PMID 10484173 . S2CID 709039 .
- ^ un b Lundqvist, Tomas; Fisher, Stewart L.; Kern, Gunther ; Folmer, Rutger HA; Xue, Yafeng ; Newton, D. Trevor ; Keating, Thomas A.; Alm, Richard A.; et al. (2007). « Exploitation de la diversité structurelle et régulatrice dans les racémases de glutamate ». Nature . 447 (7146) : 817–22. Bibcode : 2007Natur.447..817L . doi : 10.1038/nature05689 . PMID 17568739 . S2CID 4408683 .
- ^ un b mai, Melissa; Mehboob, Shahila; Mulhearn, Debbie C.; Wang, Zhiqiang ; Yu, Huidong ; Thatcher, Gregory RJ; Santarsiero, Bernard D.; Johnson, Michael E.; et al. (2007). "Analyse structurelle et fonctionnelle de deux isozymes de racémase glutamate de Bacillus anthracis et implications pour la conception d'inhibiteurs" . Journal de biologie moléculaire . 371 (5) : 1219-1237. doi : 10.1016/j.jmb.2007.05.093 . PMC 2736553 . PMID 17610893 .
- ^ un b Taal, Makie A.; Sedelnikova, Svetlana E.; Ruzheinikov, Sergueï N.; Boulanger, Patrick J. ; Rice, David W. (2004). "Expression, purification et analyse préliminaire aux rayons X de cristaux de racémase de Bacillus subtilisglutamate" . Acta Crystallographica Section D . 60 (11) : 2031-34. doi : 10.1107/S0907444904021134 . PMID 15502318 .
- ^ un b Kim, Kook-Han; Bong, Young-Jong ; Parc, Joon Kyu; Shin, Key-Jung ; Hwang, Kwang Yeon ; Kim, Eunice Eunkyeong (2007). "Base structurelle pour l'inhibition du glutamate racémase". Journal de biologie moléculaire . 372 (2) : 434–43. doi : 10.1016/j.jmb.2007.05.003 . PMID 17658548 .
- ^ Ashiuchi, M.; Kuwana, E; Yamamoto, T; Komatsu, K; Soude, K; Misono, H (2002). "Le glutamate racémase est un inhibiteur endogène de l'ADN gyrase" . Journal de chimie biologique . 277 (42) : 39070-3. doi : 10.1074/jbc.C200253200 . PMID 12213801 .
- ^ Ashiuchi, M.; Tani, K.; Soda, K.; Misono, H. (1998). "Propriétés de la racémase glutamate de Bacillus subtilis IFO 3336 produisant du poly-glutamate". Journal de biochimie . 123 (6) : 1156–63. doi : 10.1093/oxfordjournals.jbchem.a022055 . PMID 9604005 .
- ^ Sengupta, S.; Ghosh, S.; Nagaraja, V. (2008). « Fonction au noir de la glutamate racémase de Mycobacterium tuberculosis : la racémisation et l'inhibition de l'ADN gyrase sont deux activités indépendantes de l'enzyme » . Microbiologie . 154 (9) : 2796–803. doi : 10.1099/mic.0.2008/020933-0 . PMID 18757813 .
- ^ Sirover, Michael A (1999). « De nouvelles connaissances sur une ancienne protéine : la diversité fonctionnelle de la glycéraldéhyde-3-phosphate déshydrogénase des mammifères ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 1432 (2) : 159-84. doi : 10.1016/S0167-4838(99)00119-3 . PMID 10407139 .
- ^ Constantinide, SM; Deal Jr, WC (1969). "Dissociation réversible de la glycéraldéhyde 3-phosphate déshydrogénase tétramère de muscle de lapin en dimères ou monomères par l'adénosine triphosphate" . Le Journal de Chimie Biologique . 244 (20) : 5695–702. doi : 10.1016/S0021-9258 (18) 63615-4 . PMID 4312250 .
- ^ Kumagai, H; Sakai, H (1983). « Une protéine de cerveau porcin (protéine 35 K) qui regroupe des microtubules et son identification en tant que glycéraldéhyde 3-phosphate déshydrogénase ». Journal de biochimie . 93 (5) : 1259-1269. doi : 10.1093/oxfordjournals.jbchem.a134260 . PMID 6885722 .
- ^ un b De Riel, Jon K.; Paulus, Henri (1978). « La dissociation des sous-unités dans la régulation allostérique de la glycérol kinase d'Escherichia coli. 2. Preuve physique ». Biochimie . 17 (24) : 5141-6. doi : 10.1021/bi00617a011 . PMID 215195 .
- ^ un b De Riel, Jon K.; Paulus, Henri (1978). "Dissociation des sous-unités dans la régulation allostérique de la glycérol kinase d'Escherichia coli. 1. Preuve cinétique". Biochimie . 17 (24) : 5134–40. doi : 10.1021/bi00617a010 . PMID 215194 .
- ^ un b De Riel, Jon K.; Paulus, Henri (1978). "Dissociation des sous-unités dans la régulation allostérique de la glycérol kinase d'Escherichia coli. 3. Rôle dans la désensibilisation". Biochimie . 17 (24) : 5146-50. doi : 10.1021/bi00617a012 . PMID 31903 .
- ^ a b Feese, Michael D; Faber, H Rick ; Bystrom, Cory E; Pettigrew, Donald W; Remington, S James (1998). « Glycérol kinase d'Escherichia coli et un mutant Ala65 → Thr : les structures cristallines révèlent des changements conformationnels avec des implications pour la régulation allostérique » . Structurer . 6 (11) : 1407–18. doi : 10.1016/S0969-2126(98)00140-3 . PMID 9817843 .
- ^ un b Bystrom, Cory E.; Pettigrew, Donald W.; Branchaud, Bruce P.; O'Brien, Patrick ; Remington, S. James (1999). "Les structures cristallines de la variante S58→W de la glycérol kinase d'Escherichia coli dans un complexe avec des analogues d'ATP non hydrolysables révèlent une conformation active putative de l'enzyme à la suite d'un mouvement de domaine". Biochimie . 38 (12) : 3508–18. doi : 10.1021/bi982460z . PMID 10090737 .
- ^ un b Deprez, Eric; Tauc, Patrick ; Leh, Hervé ; Mouscadet, Jean-François ; Auclair, Christian ; Brochon, Jean-Claude (2000). « États oligomères de l'intégrase du VIH-1 tels que mesurés par l'anisotropie de fluorescence résolue dans le temps ». Biochimie . 39 (31) : 9275-84. doi : 10.1021/bi000397j . PMID 10924120 .
- ^ un b Deprez, E.; Tauc, P.; Leh, H.; Mouscadet, J.-F. ; Auclair, C.; Hawkins, moi ; Brochon, J.-C. (2001). « La liaison à l'ADN induit la dissociation de la forme multimérique de l'intégrase du VIH-1 : une étude d'anisotropie de fluorescence résolue en temps » . Actes de l'Académie nationale des sciences . 98 (18) : 10090-5. Bibcode : 2001PNAS ... 9810090D . doi : 10.1073/pnas.181024498 . PMC 56920 . PMID 11504911 .
- ^ A b c Faure, A. L .; Calmels, C; Desjobert, C; Castroviejo, M; Caumont-Sarcos, A; Tarrago-Litvak, L; Litvak, S; Parissi, V (2005). "Les oligomères réticulés par l'intégrase du VIH-1 sont actifs in vitro" . Recherche sur les acides nucléiques . 33 (3) : 977-86. doi : 10.1093/nar/gki241 . PMC 549407 . PMID 15718297 .
- ^ un b Guiot, E.; Carayon, K; Delelis, O; Simon, F; Tauc, P; Zubin, E; Gottikh, M; Mouscadet, JF ; et al. (2006). "Relation entre le statut oligomérique de l'intégrase du VIH-1 sur l'ADN et l'activité enzymatique" . Journal de chimie biologique . 281 (32) : 22707-19. doi : 10.1074/jbc.M602198200 . PMID 16774912 .
- ^ Fieulaine, S.; Morera, S; Poncet, S ; Monedero, V; Guéguen-Chaignon, V ; Galinier, A; Janin, J; Deutscher, J; et al. (2001). « Structure aux rayons X de la kinase HPr : Une protéine kinase bactérienne avec un domaine de liaison aux nucléotides de la boucle P » . Le Journal de l'EMBO . 20 (15) : 3917-27. doi : 10.1093/emboj/20.15.3917 . PMC 149164 . PMID 11483495 .
- ^ Marquez, José Antonio; Hasenbein, Sonja; Koch, Brigitte; Fieulaine, Sonia; Nessler, Sylvie ; Russell, Robert B.; Hengstenberg, Wolfgang ; Scheffzek, Klaus (2002). "Structure de la kinase/phosphatase HPr pleine longueur de Staphylococcus xylosus à une résolution de 1,95 : imitant le produit/substrat des réactions de transfert de phospho" . Actes de l'Académie nationale des sciences . 99 (6) : 3458-63. Bibcode : 2002PNAS ... 99.3458M . doi : 10.1073/pnas.052461499 . JSTOR 3058148 . PMC 122545 . PMID 11904409 .
- ^ Allen, Gregory S.; Steinhauer, Katrin; Hillen, Wolfgang ; Stülke, Jörg; Brennan, Richard G. (2003). « Structure cristalline de HPr Kinase/Phosphatase de Mycoplasma pneumoniae ». Journal de biologie moléculaire . 326 (4) : 1203–17. doi : 10.1016/S0022-2836(02)01378-5 . PMID 12589763 .
- ^ Poncet, Sandrine ; Mijakovic, Ivan; Nessler, Sylvie ; Guéguen-Chaignon, Virginie ; Chaptal, Vincent; Galinier, Anne; Boël, Grégory ; Mazé, Alain; et al. (2004). « HPr kinase/phosphorylase, une enzyme de capteur bifonctionnelle contenant un motif de Walker contrôlant la répression des catabolites dans les bactéries Gram-positives ». Biochimica et Biophysica Acta (BBA) - Protéines et Protéomique . 1697 (1–2) : 123–35. doi : 10.1016/j.bbapap.200311.018 . PMID 15023355 .
- ^ A b c d e Ramström, H .; Sanglier, S ; Leize-Wagner, E; Philippe, C; Van Dorsselaer, A; Haiech, J (2002). "Propriétés et régulation de l'enzyme bifonctionnelle HPr Kinase/Phosphatase dans Bacillus subtilis" . Journal de chimie biologique . 278 (2) : 1174–85. doi : 10.1074/jbc.M209052200 . PMID 12411438 .
- ^ Jault, J.-M. ; Fieulaine, S; Nessler, S ; Gonzalo, P; Di Pietro, A; Deutscher, J; Galinier, A (2000). "La kinase HPr de Bacillus subtilis est une enzyme homo-oligomère qui présente une forte coopérativité positive pour la liaison aux nucléotides et au fructose 1,6-bisphosphate" . Journal de chimie biologique . 275 (3) : 1773-1780. doi : 10.1074/jbc.275.3.1773 . PMID 10636874 .
- ^ Clarke, Anthony R.; Waldman, Adam DB ; Munro, Ian; Holbrook, J. John (1985). « Les changements dans l'état d'association de sous-unités de lactate déshydrogénase de Bacillus stearothermophilus ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 828 (3) : 375-9. doi : 10.1016/0167-4838(85)90319-X . PMID 3986214 .
- ^ A b c d e Clarke, Anthony R .; Waldman, Adam DB ; Hart, Keith W.; John Holbrook, J. (1985). « Les taux de changements définis dans la structure des protéines au cours du cycle catalytique de la lactate déshydrogénase ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 829 (3) : 397-407. doi : 10.1016/0167-4838(85)90250-X . PMID 4005269 .
- ^ Clarke, Anthony R.; Wigley, Dale B.; Barstow, David A.; Chia, William N. ; Atkinson, Tony ; Holbrook, J. John (1987). « Une seule substitution d'acide aminé dérégule une lactate déshydrogénase bactérienne et stabilise sa structure tétramère ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 913 (1) : 72-80. doi : 10.1016/0167-4838(87)90234-2 . PMID 3580377 .
- ^ Cameron, Alexandre D.; Roper, David I. ; Moreton, Kathleen M.; Muirhead, Hilary; Holbrook, J. John; Wigley, Dale B. (1994). "Activation allostérique dans Bacillus stearothermophilus Lactate Dehydrogenase étudiée par une analyse cristallographique aux rayons X d'un mutant conçu pour empêcher la tétramérisation de l'enzyme". Journal de biologie moléculaire . 238 (4) : 615-25. doi : 10.1006/jmbi.1994.1318 . PMID 8176749 .
- ^ A b c Roudiak, Stanislav G .; Shrader, Thomas E. (1998). « Rôle fonctionnel de la région N-terminale de la protéase Lon de Mycobacterium smegmatis ». Biochimie . 37 (32) : 11255–63. doi : 10.1021/bi980945h . PMID 9698372 .
- ^ A b c Rudyak, Stanislav G .; Brenowitz, Michel ; Shrader, Thomas E. (2001). « L'oligomérisation liée au Mg2 + module l'activité catalytique de la protéase Lon (La) de Mycobacterium smegmatis ». Biochimie . 40 (31) : 9317-23. doi : 10.1021/bi0102508 . PMID 11478899 .
- ^ a b Vignoble, Diana; Patterson-Ward, Jessica; Lee, Irène (2006). "Les expériences cinétiques à un seul chiffre d'affaires confirment l'existence de sites d'ATPase de haute et basse affinité dans la protéase d'Escherichia coliLon" . Biochimie . 45 (14) : 4602-10. doi : 10.1021/bi052377t . PMC 2515378 . PMID 16584195 .
- ^ un b Yang, Zhiru; Lanks, Charles W. ; Tong, Liang (2002). "Mécanisme moléculaire pour la régulation de l'enzyme malique NAD(P)+-dépendante des mitochondries humaines par l'ATP et le fumarate" . Structurer . 10 (7) : 951-60. doi : 10.1016/S0969-2126(02)00788-8 . PMID 12121650 .
- ^ un b Gerald e, Edwards; Carlos s, Andreo (1992). « Enzyme NADP-malique des plantes ». Phytochimie . 31 (6) : 1845-1857. doi : 10.1016/0031-9422(92)80322-6 . PMID 1368216 .
- ^ Hsieh, J.-Y.; Chen, S.-H.; Hung, H.-C. (2009). "Rôles fonctionnels de l'organisation tétramère de l'enzyme malique" . Journal de chimie biologique . 284 (27) : 18096-105. doi : 10.1074/jbc.M109.005082 . PMC 2709377 . PMID 19416979 .
- ^ Poole, Leslie B. (2005). « Défenses bactériennes contre les oxydants : caractéristiques mécaniques des peroxydases à base de cystéine et de leurs flavoprotéines réductases ». Archives de biochimie et biophysique . 433 (1) : 240-54. doi : 10.1016/j.abb.2004.09.006 . PMID 15581580 .
- ^ Aran, Martin; Ferrero, Diego S.; Pagano, Eduardo; Wolosiuk, Ricardo A. (2009). "Peroxiredoxines 2-Cys typiques - modulation par des transformations covalentes et des interactions non covalentes". Revue FEBS . 276 (9) : 2478–93. doi : 10.1111/j.1742-4658.2009.06984.x . PMID 19476489 . S2CID 1698327 .
- ^ Bjørgo, Elisa; De Carvalho, Raquel Margarida Negrão; Flatmark, Torgeir (2001). « Une comparaison des propriétés cinétiques et régulatrices des formes tétramères et dimères de la phénylalanine hydroxylase humaine de type sauvage et Thr427 → Pro mutant ». Journal Européen de Biochimie . 268 (4) : 997–1005. doi : 10.1046/j.1432-1327.2001.01958.x . PMID 11179966 .
- ^ Martinez, Aurora; Knappskog, Per M. ; Olafsdottir, Sigridur ; Døskeland, Anne P.; Eiken, Hans Geir; Svebak, Randi Myrseth ; Bozzini, MeriLisa; Apold, Jaran ; et al. (1995). "L'expression de la phénylalanine hydroxylase humaine recombinante en tant que protéine de fusion dans Escherichia coli contourne la dégradation protéolytique par les protéases de la cellule hôte. Isolement et caractérisation de l'enzyme de type sauvage" . Le Journal Biochimique . 306 (2) : 589–97. doi : 10.1042/bj3060589 . PMC 1136558 . PMID 7887915 .
- ^ Knappskog, Par M.; Flatmark, Torgeir ; Aarden, Johanna M.; Haavik, janvier ; Martinez, Aurore (1996). "Relations structure/fonction dans la phénylalanine hydroxylase humaine. Effet des suppressions terminales sur l'oligomérisation, l'activation et la coopérativité de la liaison du substrat à l'enzyme". Journal Européen de Biochimie . 242 (3) : 813–21. doi : 10.1111/j.1432-1033.1996.0813r.x . PMID 9022714 .
- ^ Phillips, Robert S.; Parniak, Michael A.; Kaufman, Seymour (1984). « Enquête spectroscopique de l'interaction du ligand avec la phénylalanine hydroxylase hépatique : Preuve d'un changement conformationnel associé à l'activation ». Biochimie . 23 (17) : 3836-42. doi : 10.1021/bi00312a007 . PMID 6487579 .
- ^ Fusetti, F.; Erlandsen, H; Marque plate, T ; Stevens, RC (1998). "Structure de la phénylalanine hydroxylase humaine tétramère et ses implications pour la phénylcétonurie" . Journal de chimie biologique . 273 (27) : 16962–7. doi : 10.1074/jbc.273.27.16962 . PMID 9642259 .
- ^ A b c d e f Wohl, RC; Markus, G (1972). "Phosphoenolpyruvate carboxylase d'Escherichia coli. Purification et quelques propriétés" . Le Journal de Chimie Biologique . 247 (18) : 5785-92. doi : 10.1016/S0021-9258 (19) 44827-8 . PMID 4560418 .
- ^ Kai, Yasushi; Matsumura, Hiroyoshi ; Izui, Katsura (2003). « Phosphoenolpyruvate carboxylase : structure tridimensionnelle et mécanismes moléculaires ». Archives de biochimie et biophysique . 414 (2) : 170-9. doi : 10.1016/S0003-9861(03)00170-X . PMID 12781768 .
- ^ A b c Xu, Jing; Oshima, Tairo ; Yoshida, Masasuke (1990). « Conversion tétramère-dimère de phosphofructokinase de Thermus thermophilus induite par ses effecteurs allostériques ». Journal de biologie moléculaire . 215 (4) : 597-606. doi : 10.1016/S0022-2836(05)80171-8 . PMID 2146397 .
- ^ Jolley Jr, RL; Mason, HS (1965). "Les multiples formes de champignon Tyrosinase. Interconversion" . Le Journal de Chimie Biologique . 240 : PC1489-91. doi : 10.1016/S0021-9258(18)97603-9 . PMID 14284774 .
- ^ Jolley Jr, RL; Robb, DA; Mason, HS (1969). "Les formes multiples de la tyrosinase des champignons. Phénomènes d'association-dissociation" . Le Journal de Chimie Biologique . 244 (6) : 1593–9. doi : 10.1016/S0021-9258 (18) 91800-4 . PMID 4975157 .
- ^ Mallette, MF; Dawson, CR (1949). « Sur la nature des préparations de tyrosinase de champignon hautement purifiées ». Archives de biochimie . 23 (1) : 29-44. PMID 18135760 .
- ^ un b Chazarra, Soledad; García-Carmona, Francisco; Cabanes, Juana (2001). « Hystérésis et coopérativité positive de la laitue iceberg polyphénol oxydase ». Communications de recherche biochimique et biophysique . 289 (3) : 769–75. doi : 10.1006/bbrc.2001.6014 . PMID 11726215 .
- ^ Harel, E.; Mayer, AM (1968). « Interconversion de sous-unités de catéchol oxydase de chloroplastes de pomme ». Phytochimie . 7 (2) : 199-204. doi : 10.1016/S0031-9422(00)86315-3 .
- ^ un b Jaffe EK, Lawrence SH (mars 2012). « Allostery et l'oligomérisation dynamique de la porphobilinogène synthase » . Cambre. Biochimie. Biophysique . 519 (2) : 144-53. doi : 10.1016/j.abb.2011.10.010 . PMC 3291741 . PMID 22037356 .
- ^ Breinig S, Kervinen J, Stith L, Wasson AS, Fairman R, Wlodawer A, Zdanov A, Jaffe EK (septembre 2003). « Contrôle de la biosynthèse du tétrapyrrole par des formes quaternaires alternatives de la porphobilinogène synthase ». Nat. Structurer. Biol . 10 (9) : 757–63. doi : 10.1038/nsb963 . PMID 12897770 . S2CID 24188785 .
- ^ un b Schulz, Ju¨Rgen; Sparmann, Gisèle ; Hofmann, Eberhard (1975). "Inactivation réversible médiée par l'alanine de la pyruvate kinase tumorale causée par une transition tétramère-dimère" . Lettres FEBS . 50 (3) : 346-50. doi : 10.1016/0014-5793 (75) 90064-2 . PMID 1116605 . S2CID 5665440 .
- ^ un b Ibsen, KH; Schiller, KW ; Haas, TA (1971). « Formes cinétiques et physiques interconvertibles de la pyruvate kinase des érythrocytes humains » . Le Journal de Chimie Biologique . 246 (5) : 1233-1240. doi : 10.1016/S0021-9258 (19) 76963-4 . PMID 5545066 .
- ^ Liu, Yanshun; Gotte, Giovanni ; Libonati, Massimo ; Eisenberg, David (2009). "Structures des deux trimères 3D de RNase a à échange de domaine" . Sciences des protéines . 11 (2) : 371-80. doi : 10.1110/ps.36602 . PMC 2373430 . PMID 11790847 .
- ^ un b Gotte, Giovanni; Bertoldi, Mariarita; Libonati, Massimo (1999). "Polyvalence structurelle de la ribonucléase bovine A. conformères distincts d'agrégats trimériques et tétramériques de l'enzyme" . Journal Européen de Biochimie . 265 (2) : 680-7. doi : 10.1046/j.1432-1327.1999.00761.x . PMID 10504400 .
- ^ Gotte, Giovanni; Laurents, Douglas V. ; Libonati, Massimo (2006). « Oligomères tridimensionnels à domaine échangé de la ribonucléase A : identification d'un cinquième tétramère, pentamères et hexamères, et détection de traces d'espèces heptamériques, octamériques et nonamériques ». Biochimica et Biophysica Acta (BBA) - Protéines et Protéomique . 1764 (1) : 44-54. doi : 10.1016/j.bbapap.2005.10.011 . PMID 16310422 .
- ^ un b Gotte, Giovanni; Libonati, Massimo (1998). « Deux formes différentes de dimères agrégés de ribonucléase A ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 1386 (1) : 106-112. doi : 10.1016/S0167-4838(98)00087-9 . PMID 9675255 .
- ^ un b Libonati, Massimo; Gotte, Giovanni (2004). "Oligomérisation de la ribonucléase bovine A : caractéristiques structurelles et fonctionnelles de ses multimères" . Journal biochimique . 380 (2) : 311-27. doi : 10.1042/BJ20031922 . PMC 1224197 . PMID 15104538 .
- ^ un b Libonati, M. (2004). « Actions biologiques des oligomères de la ribonucléase A ». Sciences de la vie cellulaire et moléculaire . 61 (19-20): 2431-6. doi : 10.1007/s00018-004-4302-x . PMID 15526151 . S2CID 8769502 .
- ^ un b Libonati, M; Bertoldi, M; Sorrentino, S (1996). "L'activité sur l'ARN double brin d'agrégats de ribonucléase plus élevée que les dimères augmente en fonction de la taille des agrégats" . Le Journal Biochimique . 318 (1) : 287-90. doi : 10.1042/bj3180287 . PMC 1217620 . PMID 8761484 .
- ^ un b Libonati, M.; Gotte, G.; Vottariello, F. (2008). « Une nouvelle action biologique acquise par la ribonucléase grâce à l'oligomérisation ». Biotechnologie pharmaceutique actuelle . 9 (3) : 200-9. doi : 10.2174/138920108784567308 . PMID 18673285 .
- ^ Kashlan, Ossama B.; Cooperman, Barry S. (2003). "Modèle complet pour la régulation allostérique de la ribonucléotide réductase de mammifère: raffinements et conséquences†". Biochimie . 42 (6) : 1696-706. doi : 10.1021/bi020634d . PMID 12578384 .
- ^ Kashlan, Ossama B.; Scott, Charles P. ; Lear, James D.; Cooperman, Barry S. (2002). "Un modèle complet pour la régulation allostérique de la ribonucléotide réductase des mammifères. Conséquences fonctionnelles de l'oligomérisation induite par l'ATP et le dATP de la grande sous-unité†". Biochimie . 41 (2) : 462–74. doi : 10.1021/bi011653a . PMID 11781084 .
- ^ Eriksson, Mathias; Uhlin, Ulla; Ramaswamy, S; Ekberg, Monique ; Regnström, Karin; Sjöberg, Britt-Marie; Eklund, Hans (1997). « Liaison des effecteurs allostériques à la protéine ribonucléotide réductase R1 : la réduction des cystéines du site actif favorise la liaison au substrat » . Structurer . 5 (8) : 1077-1092. doi : 10.1016/S0969-2126(97)00259-1 . PMID 9309223 .
- ^ un Fairman b , James Wesley; Wijerathna, Sanath Ranjan; Ahmad, Md Faiz ; Xu, Haï ; Nakano, Ryo ; Jha, Shalini ; Prendergast, Jay ; Welin, R. Martin ; et al. (2011). "Base structurelle pour la régulation allostérique de la ribonucléotide réductase humaine par oligomérisation induite par les nucléotides" . Nature Biologie structurale et moléculaire . 18 (3) : 316–22. doi : 10.1038/nsmb.2007 . PMC 3101628 . PMID 21336276 .
- ^ un b Hohman, RJ; Guitton, MC ; Véron, M. (1984). « Purification de la S-adénosyl-l-homocystéine hydrolase de Dictyostelium discoideum : inactivation réversible par l'AMPc et la 2′-désoxyadénosine ». Archives de biochimie et biophysique . 233 (2) : 785–95. doi : 10.1016/0003-9861(84)90507-1 . PMID 6091559 .
- ^ Guranowski, Andrzej; Pawelkiewicz, Jerzy (1977). « Adénosylhomocystéinase des graines de lupin jaune. Purification et propriétés ». Journal Européen de Biochimie . 80 (2) : 517–23. doi : 10.1111/j.1432-1033.1977.tb11907.x . PMID 923592 .
- ^ Kajander, EO; Raina, AM (1981). "Purification par chromatographie d'affinité de la S-adénosyl-L-homocystéine hydrolase. Certaines propriétés de l'enzyme du foie de rat" . Le Journal Biochimique . 193 (2) : 503-12. doi : 10.1042/bj1930503 . PMC 1162632 . PMID 7305945 .
- ^ A b c Saeki, Y; Ito, S; Shizuta, Y ; Hayaishi, O; Kagamiyama, H; Wada, H (1977). "Structure de sous-unité de la thréonine désaminase biodégradative" . Le Journal de Chimie Biologique . 252 (7) : 2206–8. doi : 10.1016/S0021-9258 (17) 40542-4 . PMID 321452 .
- ^ A b c Phillips, AT; Bois, WA (1964). « Base pour l'activation de l'AMP de la thréonine déshydrase « biodégradable » à partir de ». Communications de recherche biochimique et biophysique . 15 (6) : 530-535. doi : 10.1016/0006-291X(64)90499-1 .
- ^ A b c Gerlt, JA; Rabinowitz, KW; Dunne, CP; Bois, WA (1973). "Le mécanisme d'action de la thréonine déshydrase activée par l'acide 5'-adénylique. V. Relation entre l'activation allostérique induite par le ligand et l'interconversion des protoméroligomères" . Le Journal de Chimie Biologique . 248 (23): 8200-6. doi : 10.1016/S0021-9258 (19) 43214-6 . PMID 4584826 .
- ^ Addington, Adele K.; Johnson, David A. (1996). « Inactivation de la tryptase pulmonaire humaine : Preuve d'un intermédiaire tétramère réactivable et des monomères actifs ». Biochimie . 35 (42) : 13511-8. doi : 10.1021/bi960042t . PMID 8885830 .
- ^ Fajardo, Ignacio; Pejler, Gunnar (2003). "Formation de monomères actifs à partir de β-tryptase humaine tétramère" . Journal biochimique . 369 (3) : 603-10. doi : 10.1042/BJ20021418 . PMC 1223112 . PMID 12387726 .
- ^ Fukuoka, Yoshihiro; Schwartz, Lawrence B. (2004). « β-Tryptase humaine : détection et caractérisation du monomère actif et prévention de la reconstitution du tétramère par les inhibiteurs de la protéase ». Biochimie . 43 (33): 10757-64. doi : 10.1021/bi049486c . PMID 15311937 .
- ^ Fukuoka, Y; Schwartz, LB (2006). "L'anticorps monoclonal anti-tryptase B12 perturbe la structure tétramérique de la bêta-tryptase stabilisée par l'héparine pour former des monomères inactifs à pH neutre et actifs à pH acide" . Journal d'immunologie . 176 (5) : 3165-72. doi : 10.4049/jimmunol.176.5.3165 . PMC 1810230 . PMID 16493076 .
- ^ Fukuoka, Yoshihiro; Schwartz, Lawrence B. (2007). "Les monomères actifs de la β-tryptase humaine ont des spécificités de substrat étendues" . Immunopharmacologie internationale . 7 (14) : 1900–8. doi : 10.1016/j.intimp.2007.07.007 . PMC 2278033 . PMID 18039527 .
- ^ Hallgren, J.; Spillmann, D; Pejler, G (2001). "Exigences structurelles et mécanisme pour l'activation induite par l'héparine d'une tryptase des mastocytes de souris recombinante, protéase des mastocytes de souris-6. FORMATION DE MONOMÈRES DE TRYPTASE ACTIFS EN PRÉSENCE D'HÉPARINE DE FAIBLE POIDS MOLÉCULAIRE" . Journal de chimie biologique . 276 (46) : 42774-81. doi : 10.1074/jbc.M105531200 . PMID 11533057 .
- ^ Schechter, Norman M.; Choi, Eun-Jung ; Selwood, Trevor ; McCaslin, Darrell R. (2007). « Caractérisation de trois formes catalytiques distinctes de tryptase humaine-β : leurs interrelations et pertinence ». Biochimie . 46 (33) : 9615-29. doi : 10.1021/bi7004625 . PMID 17655281 .
- ^ Schechter, Norman M.; Ing, Grace Y. ; Selwood, Trevor ; McCaslin, Darrell R. (1995). "Changements structurels associés à l'inactivation spontanée de la tryptase humaine à sérine protéinase". Biochimie . 34 (33): 10628-38. doi : 10.1021/bi00033a038 . PMID 7654717 .
- ^ Schwartz, Lawrence B. (1994). "[6] Tryptase : Une sérine protéase de mastocyte". Enzymes protéolytiques : sérine et cystéine peptidases . Méthodes en enzymologie. 244 . p. 88-100 . doi : 10.1016/0076-6879(94)44008-5 . ISBN 978-0-12-182145-6. PMID 7845247 .
- ^ Strik, Merel CM; Wolbink, Angela; Wouters, Dorine ; Bladergroen, Bellinda A.; Verlaan, Angélique R.; van Houdt, Inge S.; Hijlkema, Sanne ; Hack, C. Erik; et al. (2004). "La serpine intracellulaire SERPINB6 (PI6) est abondamment exprimée par les mastocytes humains et forme des complexes avec les monomères β-tryptase" . Du sang . 103 (7) : 2710-7. doi : 10.1182/sang-2003-08-2981 . PMID 14670919 .
- ^ un b Kozik, Andrzej; Potempa, Jan ; Travis, James (1998). « L'inactivation spontanée de la tryptase pulmonaire humaine telle que sondée par chromatographie d'exclusion stérique et réticulation chimique : la dissociation de l'enzyme tétramère active en monomères inactifs est l'événement principal de l'ensemble du processus ». Biochimica et Biophysica Acta (BBA) - Structure des protéines et enzymologie moléculaire . 1385 (1) : 139-48. doi : 10.1016/S0167-4838(98)00053-3 . PMID 9630576 .
- ^ Alzani, R.; Cozzi, E.; Corti, A.; Temponi, M.; Trizio, D.; Gigli, M. ; Rizzo, V. (1995). « Mécanisme de désoligomérisation induite par la suramine du facteur de nécrose tumorale .alpha ». Biochimie . 34 (19) : 6344-50. doi : 10.1021/bi00019a012 . PMID 7756262 .
- ^ Corti, A; Fassina, G; Marcucci, F; Barbanti, E; Cassani, G (1992). "Le facteur de nécrose tumorale oligomère alpha se transforme lentement en formes inactives à des niveaux bioactifs" . Le Journal Biochimique . 284 (3) : 905-10. doi : 10.1042/bj2840905 . PMC 1132625 . PMID 1622406 .
- ^ Hlodan, romain; Douleur, Roger H. (1995). « La voie de pliage et d'assemblage du facteur de nécrose tumorale TNFalpha, une protéine trimère globulaire ». Journal Européen de Biochimie . 231 (2) : 381–7. doi : 10.1111/j.1432-1033.1995.tb20710.x . PMID 7635149 .
- ^ A b c d Jensen, Kaj Frank; Mygind, Bente (1996). "Différents états oligomères sont impliqués dans le comportement allostérique de l'uracile phosphoribosyltransférase d'Escherichia Coli". Journal Européen de Biochimie . 240 (3) : 637–45. doi : 10.1111/j.1432-1033.1996.0637h.x . PMID 8856065 .
