Ribonucléotide réductase - Ribonucleotide reductase
| ribonucléoside-diphosphate réductase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
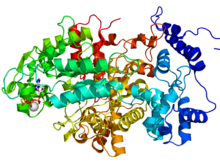
 Hétérooctamère ribonucléoside-diphosphate réductase, E.Coli
| |||||||||
| Identifiants | |||||||||
| CE n° | 1.17.4.1 | ||||||||
| N ° CAS. | 9047-64-7sy | ||||||||
| Bases de données | |||||||||
| IntEnz | Vue IntEnz | ||||||||
| BRENDA | Entrée BRENDA | ||||||||
| ExPASy | Vue NiceZyme | ||||||||
| KEGG | Entrée KEGG | ||||||||
| MétaCycle | voie métabolique | ||||||||
| PRIAM | profil | ||||||||
| Structures de l' APB | RCSB PDB PDBe PDBsum | ||||||||
| Ontologie des gènes | AmiGO / QuickGO | ||||||||
| |||||||||
La ribonucléotide réductase ( RNR ), également connue sous le nom de ribonucléoside diphosphate réductase ( rNDP ), est une enzyme qui catalyse la formation de désoxyribonucléotides à partir de ribonucléotides . Il catalyse cette formation en éliminant le groupe 2'-hydroxyle du cycle ribose des nucléosides diphosphates. Cette réduction produit des désoxyribonucléotides. Les désoxyribonucléotides sont à leur tour utilisés dans la synthèse de l' ADN . La réaction catalysée par RNR est strictement conservée dans tous les organismes vivants. De plus, le RNR joue un rôle essentiel dans la régulation du taux total de synthèse d'ADN de sorte que l'ADN par rapport à la masse cellulaire soit maintenu à un rapport constant pendant la division cellulaire et la réparation de l'ADN . Une caractéristique quelque peu inhabituelle de l'enzyme RNR est qu'elle catalyse une réaction qui se déroule via un mécanisme d'action radicalaire . Les substrats pour RNR sont ADP , GDP , CDP et UDP . Le dTDP (désoxythymidine diphosphate) est synthétisé par une autre enzyme ( thymidylate kinase ) à partir du dTMP (désoxythymidine monophosphate).
Structure
Les ribonucléotides réductases sont divisées en trois classes. Les enzymes RNR de classe I sont construites à partir de grandes sous-unités alpha et de petites sous-unités bêta qui s'associent pour former un tétramère hétérodimère actif . En réduisant les NDP en 2'-dNDP, l'enzyme catalyse la synthèse de novo des désoxyribonucléotides (dNTP), précurseurs de la synthèse de l'ADN et essentiels à la prolifération cellulaire . Les RNR de classe II produisent un radical 5'-désoxyadénosyle par clivage homolytique de la liaison C-Co dans l'adénosylcobalamine. De plus, les RNR de classe III contiennent un radical glycyle stable.
Les humains portent des RNR de classe I. La sous-unité alpha est codée par le gène RRM1 alors qu'il existe deux isoformes de la sous-unité bêta, codées par les gènes RRM2 et RRM2B :
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Chaque monomère alpha de classe I se compose de trois domaines :
- un domaine principalement hélicoïdal comprenant les 220 résidus N-terminaux ,
- une deuxième grande structure α/β à dix brins comprenant 480 résidus,
- et une troisième petite structure α/β à cinq brins comprenant 70 résidus.
Dans Pfam , le deuxième domaine a été interprété comme deux domaines distincts :
- un domaine N-terminal tout alpha plus court,
- et un domaine C-terminal plus long.
|
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
La sous-unité bêta de classe I contient généralement un centre di-métal et un radical tyrosyle stable . Chez l'homme, la sous-unité bêta repose sur un cofacteur di-fer. Dans E. coli , le radical tyrosyle est situé à la position 122 (Y122) fournissant le radical stable pour les sous-unités RNR2 de classe I. Chez A. aegypti , ce radical tyrosyle est situé en position 184 (Y184). Le radical tyrosyle est profondément enfoui à l'intérieur de la protéine dans un environnement hydrophobe, situé à proximité du centre de fer qui est utilisé dans la stabilisation d'un radical tyrosyle. La structure de deux fers -oxo-liés est dominée par des ligands qui servent de sites de liaison au fer : quatre carboxylates [ aspartate (D146), glutamate (E177, E240 et E274)] et deux histidines (H180 et H277). L'association se produit entre l'extrémité C-terminale de RNR2 et l' extrémité C-terminale de RNR1. L'activité enzymatique dépend de l'association des sous-unités RNR1 et RNR2. Le site actif est constitué des groupes dithiol actifs du RNR1 ainsi que du centre diferrique et du radical tyrosyle de la sous-unité RNR2.
D'autres résidus de RNR2, tels que l'aspartate (D273), le tryptophane (W48) et la tyrosine (Y356) stabilisent davantage le radical tyrosyle du site actif permettant ainsi le transfert d'électrons. Ces résidus aident au transfert de l'électron radical de la tyrosine (Y122) de RNR2 à la cystéine (C439) de RNR1. Le transfert d'électrons commence sur la tyrosine RNR2 (Y122) et se poursuit dans la RNR2 vers le tryptophane (W48), qui est séparé de la tyrosine RNR1 (Y731) de 2,5 nanomètres . Le transfert d'électrons de RNR2 à RNR1 se produit via la tyrosine (Y356 à Y731) et se poursuit via la tyrosine (Y730) à la cystéine (C439) dans le site actif. Les mutations dirigées vers le site de la structure primaire RNR indiquent que tous les résidus cités ci-dessus participent au transfert à longue distance du radical libre vers le site actif.
Chez les moustiques A. aegypti , RNR1 retient la plupart des résidus d'acides aminés cruciaux, y compris l'aspartate (D64) et la valine (V292 ou V284), qui sont nécessaires à la régulation allostérique ; les résidus proline (P210 et P610), leucine (L453 et L473) et méthionine (M603) qui sont situés dans le site actif hydrophobe; les résidus de cystéine (C225, C436 et C451) qui sont impliqués dans l'élimination d'un atome d'hydrogène et le transfert de l'électron radicalaire au site actif ; les résidus de cystéine (C225 et C436), d' asparagine (N434) et de glutamate (E441) qui se lient au substrat ribonucléotidique ; les résidus tyrosine (Y723 et Y743) qui dictent le transfert radicalaire ; et les résidus cystéine (C838 et C841) qui sont utilisés dans la régénération des groupes dithiol dans le site actif.
Fonction
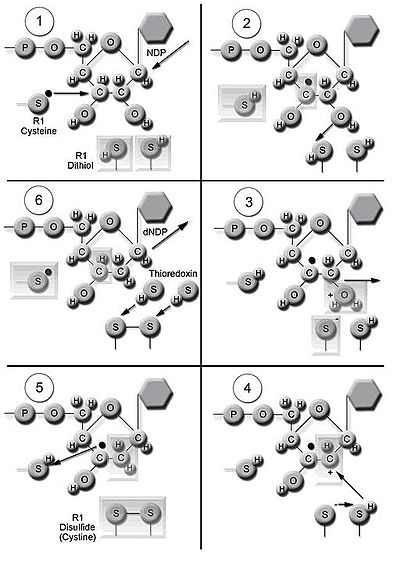
L'enzyme ribonucléotide réductase (RNR) catalyse la synthèse de novo des dNDP. La catalyse des ribonucléosides 5'-diphosphates (NDP) implique une réduction au niveau du carbone 2' du ribose 5-phosphate pour former les 2'-désoxyribonucléoside 5'-diphosphates réduits par un dérivé 2'-désoxy (dNDP). Cette réduction est initiée avec la génération d'un radical libre. Après une seule réduction, RNR nécessite des électrons donnés par les groupes dithiol de la protéine thiorédoxine . La régénération de la thiorédoxine se produit lorsque le phosphate de nicotinamide adénine dinucléotide ( NADPH ) fournit deux atomes d'hydrogène qui sont utilisés pour réduire les groupes disulfure de la thiorédoxine.
Trois classes de RNR ont des mécanismes similaires pour la réduction des NDP, mais diffèrent dans le domaine qui génère le radical libre, le métal spécifique dans la structure métalloprotéique et les donneurs d'électrons. Toutes les classes utilisent la chimie des radicaux libres. Les réductases de classe I utilisent un centre de fer avec une conversion ferreux en ferrique pour générer un radical libre tyrosyle. La réduction des substrats NDP se produit dans des conditions aérobies. Les réductases de classe I sont divisées en IA et IB en raison de différences de réglementation. Les réductases de classe IA sont distribuées chez les eucaryotes , les eubactéries , les bactériophages et les virus . Les réductases de classe IB se trouvent dans les eubactéries. Les réductases de classe IB peuvent également utiliser un radical généré avec la stabilisation d'un centre de manganèse binucléaire . Les réductases de classe II génèrent le radical libre 5'-désoxyadénosyle à partir de la cobalamine (coenzyme B12) et ont une structure plus simple que les réductases de classe I et de classe III. La réduction des NDP ou des ribonucléotides 5'-triphosphates (NTP) se produit dans des conditions aérobies ou anaérobies. Les réductases de classe II sont distribuées dans les archaebactéries , les eubactéries et les bactériophages. Les réductases de classe III utilisent un radical glycine généré à l'aide d'une S-adénosyl méthionine et d'un centre fer-soufre. La réduction des PNT est limitée aux conditions anaérobies. Les réductases de classe III sont distribuées dans les archaebactéries, les eubactéries et les bactériophages. Les organismes ne sont pas limités à avoir une seule classe d'enzymes. Par exemple, E. coli a à la fois un RNR de classe I et de classe III.
Mécanisme de réduction catalytique
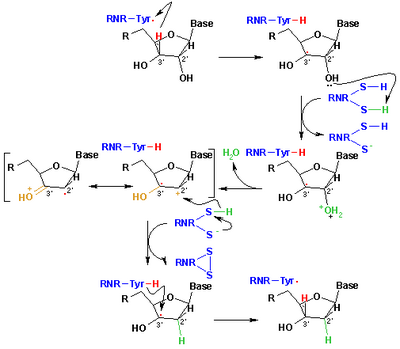
Le mécanisme actuellement accepté pour la réduction des ribonucléotides en désoxyribonucléotides est représenté dans le schéma suivant. La première étape consiste en l'abstraction du 3'-H du substrat 1 par le radical Cys439. Par la suite, la réaction implique l'élimination d'une molécule d'eau du carbone C-2' du ribonucléotide, catalysée par Cys225 et Glu441. Dans la troisième étape, il y a un transfert d'atomes d'hydrogène de Cys225 au carbone C-2' du radical 2'-cétyle 3, après un précédent transfert de protons de Cys462 à Cys225. A l'issue de cette étape, on obtient un pont disulfure anionique radicalaire et l'intermédiaire 4 cétone à coque fermée. Cet intermédiaire a été identifié lors de la conversion de plusieurs analogues de substrats substitués en 2', ainsi qu'avec le substrat naturel interagissant avec des mutants enzymatiques. L'étape suivante est l'oxydation du pont disulfure anionique, avec réduction concomitante du substrat, générant 5. La densité de spin passe des atomes de soufre à l'atome C-3' du substrat, avec transfert simultané de protons de Glu441 au carbone C -3'. La dernière étape est l'inverse de la première étape et implique un transfert d'hydrogène de Cys439 à C-3', régénérant le radical initial et aboutissant au produit final 6.
Des modèles théoriques de certaines étapes de ces mécanismes utilisant le modèle complet de la protéine R1 peuvent être trouvés dans les études réalisées par Cerqueira et al. .
Régulation
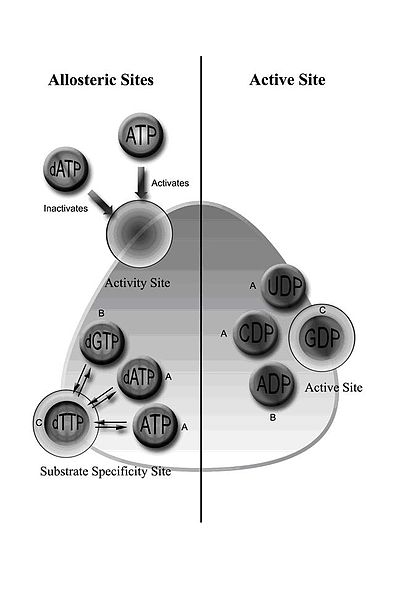
Le RNR de classe I comprend les sous-unités RNR1 et RNR2, qui peuvent s'associer pour former un tétramère hétérodimère. RNR1 contient les deux sites allostériques, médiateurs de la régulation de la spécificité et de l'activité du substrat. Selon la configuration allostérique, l'un des quatre ribonucléotides se lie au site actif.
La régulation du RNR est conçue pour maintenir des quantités équilibrées de dNTP. La liaison des molécules effectrices augmente ou diminue l'activité RNR. Lorsque l'ATP se lie au site d'activité allostérique, il active le RNR. En revanche, lorsque le dATP se lie à ce site, il désactive le RNR. En plus de contrôler l'activité, le mécanisme allostérique régule également la spécificité du substrat et garantit que l'enzyme produit une quantité égale de chaque dNTP pour la synthèse de l'ADN. Dans toutes les classes, la liaison de l'ATP ou du dATP au site allostérique induit une réduction de la cytidine 5'-diphosphate (CDP) et de l'uridine 5'-diphosphate (UDP) ; le 2'-désoxyguanosine 5'-triphosphate (dGTP) induit une réduction de l'adénosine 5'-diphosphate (ADP) ; et le 2'-désoxythymidine 5'-triphosphate (dTTP) induit une réduction de la guanosine 5'-diphosphate (GDP) (figure 1).
Les réductases de classe IB ne sont pas inhibées par le dATP car il leur manque environ 50 acides aminés N-terminaux requis pour le site d'activité allostérique. De plus, il est important que l'activité de la ribonucléotide réductase soit sous contrôle transcriptionnel et post-transcriptionnel car la synthèse d'ADN sans dommage repose sur un pool équilibré de désoxyribonucléotides. Les cellules eucaryotes avec des réductases de classe IA ont un mécanisme de contrôle négatif pour désactiver la synthèse des dNTP lorsqu'ils s'accumulent. Ce mécanisme protège la cellule des effets toxiques et mutagènes qui peuvent résulter de la surproduction de dNTP, car des changements dans les pools de dNTP équilibrés entraînent des dommages à l'ADN et la mort cellulaire. Bien que la surproduction de dNTP ou un apport déséquilibré de ceux-ci puisse conduire à une mauvaise incorporation de nucléotides dans l'ADN, l'apport de dNTP peut permettre la réparation de l'ADN. p53R2 est une petite sous-unité de la ribonucléotide réductase qui peut induire une telle réparation. Des changements au sein de cet homologue R2 induit par p53 peuvent entraîner un épuisement de l'ADN mitochondrial et, par conséquent, p53R2 sert un facteur majeur dans l'approvisionnement en dNTP.
RNR peut utiliser le modèle morpheein de régulation allostérique .
Inhibiteurs de RNR1 et RNR2
Généralement, les inhibiteurs de RNR de classe I peuvent être divisés en trois groupes principaux : les inhibiteurs de traduction, qui bloquent la synthèse de l'enzyme ; les inhibiteurs de dimérisation qui empêchent l'association des deux sous-unités RNR (R1 et R2) ; et des inhibiteurs catalytiques qui inactivent la sous-unité R1 et/ou la sous-unité R2.
Le RNR de classe I peut être inhibé par des peptides similaires à l' extrémité C-terminale du RNR2. Ces peptides peuvent entrer en compétition avec RNR2 pour se lier à RNR1 et, par conséquent, RNR1 ne forme pas de complexe enzymatiquement actif avec RNR2. Bien que l'extrémité C-terminale des protéines RNR2 soit différente d'une espèce à l'autre, RNR2 peut interagir avec RNR1 d'une espèce à l'autre. Lorsque l'extrémité C-terminale du RNR2 de la souris a été remplacée par les résidus d'acides aminés E. coli RNR2 C-terminaux (7 ou 33), la sous-unité RNR2 chimérique se lie toujours aux sous-unités RNR1 de la souris. Cependant, ils manquent d'activité enzymatique en raison probablement de l'élimination des résidus impliqués dans le transfert de l'électron radicalaire du RNR2 vers la sous-unité RNR1.
Les petits peptides peuvent spécifiquement inhiber la liaison des sous-unités RNR2 avec RNR1 lorsqu'ils partagent une similitude significative avec l'extrémité C-terminale normale de RNR2. Cette inhibition de la liaison de RNR2 à RNR1 a été testée avec succès dans le RNR du virus de l'herpès simplex (HSV). Lorsqu'un oligomère de 7 acides aminés (GAVVNDL) tronqué à partir de l'extrémité C-terminale de la sous-unité RNR2 a été utilisé dans des tests de compétition, il a empêché le RNR2 normal de former un complexe enzymatiquement actif avec RNR1. D'autres petits inhibiteurs peptidiques similaires à l'extrémité C-terminale de RNR2 ont également été utilisés avec succès pour inhiber l'activité enzymatique de HSV RNR et donc la réplication de HSV. Dans des modèles murins de kératite stromale et de néovascularisation cornéenne ( maladie oculaire à HSV ), il a été rapporté qu'un petit analogue RNR2 C-terminal BILD 1263 inhibe la RNR et est efficace pour prévenir ces maladies. Dans certains cas, bien que le traitement avec de petits analogues C-terminaux ne puisse pas arrêter la propagation de la maladie, ils peuvent quand même aider à la guérison. Dans le HSV résistant à l' acyclovir (PAAr5), un petit inhibiteur peptidique BILD 1633 a été rapporté comme étant 5 à 10 fois plus puissant que BILD 1263 contre l'infection cutanée PAAr5. Une approche de thérapie combinée (BILD 1633 et acyclovir) est plus efficace pour guérir les lésions topiques chez la souris. Ces données suggèrent que les petits inhibiteurs peptidiques qui entrent en compétition avec RNR2 pour se lier à RNR1 sont utiles pour empêcher la propagation du HSV.
Le gallium inhibe RNR2 en se substituant à Fe 3+ dans le site actif. Le maltolate de gallium est une forme de gallium biodisponible par voie orale qui exploite cette activité inhibitrice pour traiter le cancer, les infections et d'autres maladies.
Les médicaments hydroxyurée et Motexafin gadolinium interfèrent avec l'action de cette enzyme.
Les références
Liens externes
- Ribonucléotide+réductases à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis
- La base de données de la ribonucléotide réductase (RNRdb)