Spectrométrie de masse en tandem - Tandem mass spectrometry
La spectrométrie de masse en tandem , également connue sous le nom de MS/MS ou MS 2 , est une technique d' analyse instrumentale dans laquelle deux ou plusieurs analyseurs de masse sont couplés en utilisant une étape de réaction supplémentaire pour augmenter leurs capacités à analyser des échantillons chimiques. Une utilisation courante de la SM en tandem est l'analyse de biomolécules , telles que les protéines et les peptides .
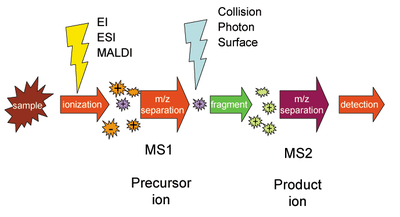
Les molécules d'un échantillon donné sont ionisées et le premier spectromètre (désigné MS1 ) sépare ces ions par leur rapport masse sur charge (souvent exprimé en m/z ou m/Q). Les ions d'un rapport m/z particulier provenant de MS1 sont sélectionnés puis divisés en ions fragments plus petits , par exemple par dissociation induite par collision , réaction ion-molécule ou photodissociation . Ces fragments sont ensuite introduits dans le deuxième spectromètre de masse ( MS2 ), qui à son tour sépare les fragments par leur rapport m/z et les détecte . L'étape de fragmentation permet d'identifier et de séparer les ions qui ont des rapports m/z très similaires dans les spectromètres de masse ordinaires.
Structure
La spectrométrie de masse en tandem comprend un spectromètre de masse à triple quadripôle (QqQ), un spectromètre de masse multisectoriel , un quadripôle à temps de vol (Q-TOF) et un spectromètre de masse hybride.
Spectromètre de masse triple quadripôle
Les spectromètres de masse à triple quadripôle utilisent les premier et troisième quadripôles comme filtres de masse. Lorsque les analytes passent le deuxième quadripôle, la fragmentation se produit par collision avec le gaz.
Quadrupôle–temps de vol (Q-TOF)
Le spectromètre de masse Q-TOF combine des instruments TOF et quadripolaires, ce qui permet une précision de masse élevée pour les ions produits, une capacité de quantification précise et une applicabilité aux expériences de fragmentation. Il s'agit d'une méthode de spectrométrie de masse dont le rapport de fragmentation ionique ( m / z ) est déterminé grâce à une mesure du temps de vol.
Spectromètre de masse hybride
Le spectromètre de masse hybride se compose de plus de deux analyseurs de masse.
Instrumentation
Plusieurs étapes de séparation par analyse de masse peuvent être accomplies avec des éléments de spectromètre de masse individuels séparés dans l'espace ou en utilisant un seul spectromètre de masse avec les étapes MS séparées dans le temps. Pour la spectrométrie de masse en tandem dans l'espace, les différents éléments sont souvent notés de manière abrégée, donnant le type de sélecteur de masse utilisé.
Tandem dans l'espace
Dans la spectrométrie de masse en tandem dans l'espace , les éléments de séparation sont physiquement séparés et distincts, bien qu'il existe une connexion physique entre les éléments pour maintenir un vide poussé . Ces éléments peuvent être des secteurs , des quadripôles de transmission , ou des temps de vol. Lors de l'utilisation de plusieurs quadripôles, ils peuvent servir à la fois d'analyseurs de masse et de chambres de collision.
La notation courante pour les analyseurs de masse est Q – analyseur de masse quadripolaire ; q – quadripôle de collision radiofréquence ; TOF – analyseur de masse à temps de vol ; B – secteur magnétique et E – secteur électrique. La notation peut être combinée pour indiquer divers instruments hybrides, par exemple QqQ' – spectromètre de masse triple quadripôle ; QTOF – spectromètre de masse à temps de vol quadripolaire (également QqTOF ); et BEBE – spectromètre de masse à quatre secteurs (géométrie inverse).
Tandem dans le temps
En faisant de la spectrométrie de masse en tandem dans le temps , la séparation est réalisée avec des ions piégés au même endroit, plusieurs étapes de séparation ayant lieu au fil du temps. Un piège à ions quadripolaire ou un instrument à résonance cyclotron ionique à transformée de Fourier (FTICR) peut être utilisé pour une telle analyse. Les instruments de piégeage peuvent effectuer plusieurs étapes d'analyse, parfois appelées MS n (MS to the n ). Souvent, le nombre d'étapes, n , n'est pas indiqué, mais parfois la valeur est spécifiée ; par exemple MS 3 indique trois étapes de séparation. Les instruments MS en tandem dans le temps n'utilisent pas les modes décrits ci-après, mais collectent généralement toutes les informations à partir d'un balayage d'ions précurseurs et d'un balayage d'ions parents de l'ensemble du spectre. Chaque configuration instrumentale utilise un mode unique d'identification de masse.
Tandem dans les modes MS/MS spatiaux
Lorsque la MS en tandem est effectuée avec une conception dans l'espace, l'instrument doit fonctionner dans l'un des divers modes. Il existe un certain nombre de configurations expérimentales MS/MS en tandem différentes et chaque mode a ses propres applications et fournit des informations différentes. Le tandem MS dans l'espace utilise le couplage de deux composants d'instruments qui mesurent la même gamme de spectre de masse mais avec un fractionnement contrôlé entre eux dans l'espace, tandis que le tandem MS dans le temps implique l'utilisation d'un piège à ions .
Il existe quatre expériences de balayage principales possibles à l'aide de MS/MS : balayage d'ions précurseurs, balayage d'ions produits, balayage de perte neutre et surveillance de réaction sélectionnée.
Pour un balayage d'ions précurseurs, l'ion produit est sélectionné dans le deuxième analyseur de masse, et les masses précurseurs sont balayées dans le premier analyseur de masse. Notez que l'ion précurseur est synonyme d'ion parent et l'ion produit d'ion fille ; cependant, l'utilisation de ces termes anthropomorphes est déconseillée.
Dans un balayage d'ions produits, un ion précurseur est sélectionné dans la première étape, laissé se fragmenter, puis toutes les masses résultantes sont balayées dans le deuxième analyseur de masse et détectées dans le détecteur qui est positionné après le deuxième analyseur de masse. Cette expérience est couramment réalisée pour identifier les transitions utilisées pour la quantification par MS en tandem.
Dans un balayage à perte neutre, le premier analyseur de masse balaie toutes les masses. Le deuxième analyseur de masse effectue également un balayage, mais à un décalage défini par rapport au premier analyseur de masse. Ce décalage correspond à une perte neutre qui est couramment observée pour la classe de composés. Dans un balayage à perte de neutre constant, tous les précurseurs qui subissent la perte d'un neutre commun spécifié sont surveillés. Pour obtenir ces informations, les deux analyseurs de masse sont scannés simultanément, mais avec un décalage de masse qui correspond à la masse du neutre spécifié. Semblable au balayage d'ions précurseurs, cette technique est également utile dans l'identification sélective d'une classe étroitement liée de composés dans un mélange.
Dans la surveillance de réaction sélectionnée, les deux analyseurs de masse sont réglés sur une masse sélectionnée. Ce mode est analogue à la surveillance des ions sélectionnés pour les expériences MS. Un mode d'analyse sélective, qui peut augmenter la sensibilité.
Fragmentation
La fragmentation des ions en phase gazeuse est essentielle à la spectrométrie de masse en tandem et se produit entre les différentes étapes de l'analyse de masse. Il existe de nombreuses méthodes utilisées pour fragmenter les ions et celles-ci peuvent entraîner différents types de fragmentation et donc différentes informations sur la structure et la composition de la molécule.
Fragmentation à la source
Souvent, le processus d' ionisation est suffisamment violent pour laisser les ions résultants avec une énergie interne suffisante pour se fragmenter dans le spectromètre de masse. Si les ions produits persistent dans leur état de non-équilibre pendant une durée modérée avant l'auto-dissociation, ce processus est appelé fragmentation métastable . La fragmentation buse-écrémeur fait référence à l'induction délibérée de la fragmentation dans la source en augmentant le potentiel buse-écrémeur sur des instruments généralement basés sur l' électrospray . Bien que la fragmentation à la source permette l'analyse de fragmentation, il ne s'agit pas techniquement de spectrométrie de masse en tandem à moins que les ions métastables ne soient analysés en masse ou sélectionnés avant l'auto-dissociation et qu'une deuxième étape d'analyse soit effectuée sur les fragments résultants. La fragmentation en source peut être utilisée à la place de la spectrométrie de masse en tandem grâce à l'utilisation de la technologie EISA (Enhanced in-Source Fragmentation Annotation) qui génère une fragmentation qui correspond directement aux données de spectrométrie de masse en tandem. Les fragments observés par EISA ont une intensité de signal plus élevée que les fragments traditionnels qui subissent des pertes dans les cellules de collision des spectromètres de masse en tandem. L'EISA permet l'acquisition de données de fragmentation sur les analyseurs de masse MS1 tels que les instruments à temps de vol et à quadripôle unique. La fragmentation en source est souvent utilisée en plus de la spectrométrie de masse en tandem (avec fragmentation post-source) pour permettre deux étapes de fragmentation dans une expérience de type pseudo MS 3 .
Dissociation induite par collision
La fragmentation post-source est le plus souvent ce qui est utilisé dans une expérience de spectrométrie de masse en tandem. De l'énergie peut également être ajoutée aux ions, qui sont généralement déjà excités vibratoirement, par des collisions post-source avec des atomes ou des molécules neutres, l'absorption de rayonnement, ou le transfert ou la capture d'un électron par un ion à charges multiples. La dissociation induite par collision (CID), également appelée dissociation activée par collision (CAD), implique la collision d'un ion avec un atome ou une molécule neutre en phase gazeuse et la dissociation ultérieure de l'ion. Par exemple, considérez
où l'ion AB + entre en collision avec l'espèce neutre M et se sépare ensuite. Les détails de ce processus sont décrits par la théorie des collisions . En raison de la configuration instrumentale différente, deux types principaux de CID sont possibles : (i) le type à faisceau (dans lequel les ions précurseurs sont fragmentés en vol) et (ii) le type à piège à ions (dans lequel les ions précurseurs sont d'abord piégés , puis fragmenté).
Un troisième type de fragmentation CID, plus récent, est la dissociation collisionnelle à haute énergie (HCD). Le HCD est une technique CID spécifique aux spectromètres de masse orbitrap dans lesquels la fragmentation a lieu à l'extérieur du piège à ions, elle se produit dans la cellule HCD (dans certains instruments nommés « ion routing multipole »). Le HCD est une fragmentation de type piège qui a des caractéristiques de type faisceau. Il existe des bases de données de spectrométrie de masse en tandem à haute résolution et à grande échelle disponibles gratuitement (par exemple METLIN avec 850 000 étalons moléculaires chacun avec des données expérimentales CID MS/MS), et sont généralement utilisées pour faciliter l'identification de petites molécules.
Méthodes de capture et de transfert d'électrons
L'énergie libérée lorsqu'un électron est transféré ou capturé par un ion à charges multiples peut induire une fragmentation.
Dissociation par capture d'électrons
Si un électron est ajouté à un ion positif chargé plusieurs fois, l' énergie coulombienne est libérée. L'ajout d'un électron libre est appelé dissociation par capture d'électrons (ECD) et est représenté par
pour une molécule multiprotonée M.
Dissociation par transfert d'électrons
L'ajout d'un électron via une réaction ion-ion est appelé dissociation par transfert d'électrons (ETD). Semblable à la dissociation par capture d'électrons, l'ETD induit une fragmentation des cations (par exemple des peptides ou des protéines ) en leur transférant des électrons . Il a été inventé par Donald F. Hunt , Joshua Coon , John EP Syka et Jarrod Marto à l' Université de Virginie .
ETD n'utilise pas d'électrons libres mais emploie des anions radicaux (par exemple l' anthracène ou l' azobenzène ) à cette fin :
où A est l'anion.
ETD se clive de manière aléatoire le long du squelette peptidique (ions c et z) tandis que les chaînes latérales et les modifications telles que la phosphorylation sont laissées intactes. La technique ne fonctionne bien que pour les ions à état de charge plus élevé (z> 2), mais par rapport à la dissociation induite par collision (CID), l'ETD est avantageux pour la fragmentation de peptides plus longs ou même de protéines entières. Cela rend la technique importante pour la protéomique descendante . Tout comme l'ECD, l'ETD est efficace pour les peptides avec des modifications telles que la phosphorylation.
Le transfert d'électrons et la dissociation par collision à haute énergie (EThcD) sont une combinaison ETD et HCD où le précurseur peptidique est initialement soumis à une réaction ion/ion avec des anions fluoranthène dans un piège à ions linéaire , qui génère des ions c et z. Dans la deuxième étape, la fragmentation de tous les ions HCD est appliquée à tous les ions dérivés de l'ETD pour générer des ions b et y avant l'analyse finale dans l'analyseur orbitrap. Cette méthode utilise une double fragmentation pour générer des spectres MS/MS riches en ions et donc en données pour le séquençage des peptides et la localisation PTM .
Dissociation négative par transfert d'électrons
La fragmentation peut également se produire avec une espèce déprotonée, dans laquelle un électron est transféré de l'espèce à un réactif cationique dans une dissociation négative par transfert d'électrons (NETD) :
Suite à cet événement de transfert, l'anion déficient en électrons subit un réarrangement interne et des fragments . NETD est l'analogue ion/ion de la dissociation par détachement d' électrons (EDD).
NETD est compatible avec la fragmentation des peptides et des protéines le long du squelette au niveau de la liaison C α -C. Les fragments résultants sont généralement des ions produits de type • - et x.
Dissociation par détachement d'électrons
La dissociation par détachement d'électrons (EDD) est une méthode de fragmentation d'espèces anioniques en spectrométrie de masse. Il sert de mode de compteur négatif à la dissociation de capture d'électrons. Les ions chargés négativement sont activés par irradiation avec des électrons d'énergie cinétique modérée. Le résultat est l'éjection d'électrons de la molécule ionique parente , ce qui provoque une dissociation par recombinaison.
Dissociation de transfert de charge
La réaction entre les peptides chargés positivement et les réactifs cationiques, également connue sous le nom de dissociation par transfert de charge (CTD), a récemment été démontrée comme une voie alternative de fragmentation à haute énergie pour les peptides à faible charge (1+ ou 2+). Le mécanisme proposé de CTD utilisant des cations d'hélium comme réactif est :
Les rapports initiaux indiquent que le CTD provoque le clivage de la liaison C α -C du squelette des peptides et fournit des ions produits de type • - et x.
Photodissociation
L'énergie nécessaire à la dissociation peut être ajoutée par absorption de photons , ce qui entraîne une photodissociation ionique et représentée par
où représente le photon absorbé par l'ion. Les lasers ultraviolets peuvent être utilisés, mais peuvent conduire à une fragmentation excessive des biomolécules.
Dissociation multiphotonique infrarouge
Les photons infrarouges chaufferont les ions et provoqueront une dissociation si suffisamment d'entre eux sont absorbés. Ce processus est appelé dissociation multiphotonique infrarouge (IRMPD) et est souvent accompli avec un laser au dioxyde de carbone et un spectromètre de masse à piégeage d'ions tel qu'un FTMS .
Dissociation radiative infrarouge du corps noir
Le rayonnement du corps noir peut être utilisé pour la photodissociation dans une technique connue sous le nom de dissociation radiative infrarouge du corps noir (BIRD). Dans la méthode BIRD, toute la chambre à vide du spectromètre de masse est chauffée pour créer une lumière infrarouge . BIRD utilise ce rayonnement pour exciter des vibrations de plus en plus énergétiques des ions, jusqu'à ce qu'une liaison se brise, créant des fragments. Ceci est similaire à la dissociation multiphotonique infrarouge qui utilise également la lumière infrarouge, mais à partir d'une source différente. BIRD est le plus souvent utilisé avec la spectrométrie de masse à résonance cyclotron ionique à transformée de Fourier .
Dissociation induite par la surface
Avec la dissociation induite par la surface (SID), la fragmentation est le résultat de la collision d'un ion avec une surface sous vide poussé. Aujourd'hui, le SID est utilisé pour fragmenter une large gamme d'ions. Il y a des années, il n'était courant d'utiliser le SID que sur des espèces à faible masse et à charge unique, car les méthodes d'ionisation et les technologies d'analyse de masse n'étaient pas assez avancées pour former, transmettre ou caractériser correctement des ions de m/z élevé. Au fil du temps, les surfaces monocouches auto-assemblées (SAM) composées de CF3(CF2)10CH2CH2S sur l'or ont été les surfaces de collision les plus utilisées pour le SID dans un spectromètre en tandem. Les SAM ont agi comme les cibles de collision les plus souhaitables en raison de leurs masses efficaces caractéristiquement élevées pour la collision des ions entrants. De plus, ces surfaces sont composées de chaînes fluorocarbonées rigides, qui n'atténuent pas de manière significative l'énergie des ions du projectile. Les chaînes fluorocarbonées sont également bénéfiques en raison de leur capacité à résister à un transfert d'électrons facile de la surface métallique aux ions entrants. La capacité de SID à produire des sous-complexes qui restent stables et fournissent des informations précieuses sur la connectivité est inégalée par aucune autre technique de dissociation. Étant donné que les complexes produits à partir de SID sont stables et conservent une distribution de charge sur le fragment, cela produit un spectre unique que le complexe centre autour d'une distribution m/z plus étroite. Les produits SID et l'énergie à laquelle ils se forment reflètent les forces et la topologie du complexe. Les modèles de dissociation uniques aident à découvrir la structure quaternaire du complexe. La distribution de charge symétrique et la dépendance à la dissociation sont uniques au SID et rendent les spectres produits distinctifs de toute autre technique de dissociation.
La technique SID est également applicable à la spectrométrie de masse à mobilité ionique (IM-MS). Trois méthodes différentes pour cette technique comprennent l'analyse de la caractérisation de la topologie, de la connectivité intersous-unité et du degré de déploiement de la structure des protéines. L'analyse du dépliement de la structure des protéines est l'application la plus couramment utilisée de la technique SID. Pour la spectrométrie de masse à mobilité ionique (IM-MS), le SID est utilisé pour la dissociation des précurseurs activés par la source de trois types différents de complexes protéiques : la protéine C réactive (CRP), la transthyrétine (TTR) et la concanavaline A (Con A) . Cette méthode permet d'observer le degré de dépliement de chacun de ces complexes. Pour cette observation, SID a montré les structures des ions précurseurs qui existent avant la collision avec la surface. IM-MS utilise le SID comme mesure directe de la conformation pour chaque sous-unité de protéines.
La résonance cyclotron ionique à transformée de Fourier (FTICR) est capable de fournir une résolution ultra-élevée et une précision de masse élevée aux instruments qui prennent des mesures de masse. Ces caractéristiques font des spectromètres de masse FTICR un outil utile pour une grande variété d'applications telles que plusieurs expériences de dissociation telles que la dissociation induite par collision (CID, dissociation par transfert d'électrons (ETD) et autres. De plus, la dissociation induite par la surface a été mise en œuvre avec cet instrument pour l'étude de la fragmentation fondamentale des peptides. Spécifiquement, SID a été appliqué à l'étude de l'énergétique et de la cinétique de la fragmentation en phase gazeuse au sein d'un instrument ICR. Cette approche a été utilisée pour comprendre la fragmentation en phase gazeuse des peptides protonés, des ions peptidiques à électrons impairs, des complexes ligand-peptide non covalents et des amas métalliques ligaturés.
Protéomique quantitative
La protéomique quantitative est utilisée pour déterminer la quantité relative ou absolue de protéines dans un échantillon. Plusieurs méthodes de protéomique quantitative sont basées sur la spectrométrie de masse en tandem. MS/MS est devenu une procédure de référence pour l'élucidation structurale de biomolécules complexes.
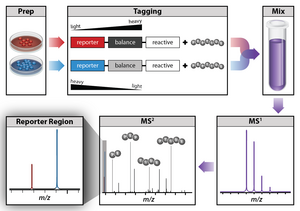
Une méthode couramment utilisée pour la protéomique quantitative est l'étiquetage isobare. L'étiquetage isobare permet l'identification et la quantification simultanées des protéines de plusieurs échantillons en une seule analyse. Pour quantifier les protéines, les peptides sont marqués avec des étiquettes chimiques qui ont la même structure et la même masse nominale, mais varient dans la distribution des isotopes lourds dans leur structure. Ces balises, communément appelées balises de masse en tandem, sont conçues de manière à ce que la balise de masse soit clivée au niveau d'une région de liaison spécifique lors d'une dissociation induite par collision (HCD) à haute énergie au cours de la spectrométrie de masse en tandem, produisant des ions rapporteurs de masses différentes. La quantification des protéines est réalisée en comparant les intensités des ions rapporteurs dans les spectres MS/MS. Deux marqueurs isobares disponibles dans le commerce sont les réactifs iTRAQ et TMT.
Balises isobares pour la quantification relative et absolue (iTRAQ)
Une étiquette isobare pour la quantification relative et absolue (iTRAQ) est un réactif pour la spectrométrie de masse en tandem qui est utilisé pour déterminer la quantité de protéines provenant de différentes sources dans une seule expérience. Il utilise des molécules marquées par des isotopes stables qui peuvent former une liaison covalente avec l'extrémité N-terminale et les amines de la chaîne latérale des protéines. Les réactifs iTRAQ sont utilisés pour marquer les peptides de différents échantillons qui sont regroupés et analysés par chromatographie liquide et spectrométrie de masse en tandem. La fragmentation de l'étiquette attachée génère un ion rapporteur de faible masse moléculaire qui peut être utilisé pour quantifier relativement les peptides et les protéines dont ils sont issus.
Balise de masse en tandem (TMT)
Une étiquette de masse en tandem (TMT) est une étiquette chimique d'étiquette de masse isobare utilisée pour la quantification et l'identification des protéines. Les balises contiennent quatre régions : rapporteur de masse, lieur clivable, normalisation de masse et groupe réactif aux protéines. Les réactifs TMT peuvent être utilisés pour analyser simultanément de 2 à 11 échantillons peptidiques différents préparés à partir de cellules, de tissus ou de fluides biologiques. Trois types de réactifs TMT sont disponibles avec différentes réactivités chimiques : (1) un groupe fonctionnel ester NHS réactif pour le marquage des amines primaires (TMTduplex, TMTsixplex, TMT10plex plus TMT11-131C), (2) un groupe fonctionnel réactif iodoacétyle pour le marquage des sulfhydryles libres ( iodoTMT) et (3) une fonction alcoxyamine réactive pour le marquage des carbonyles (aminoxyTMT).
Applications
Peptides
La spectrométrie de masse en tandem peut être utilisée pour le séquençage des protéines . Lorsque des protéines intactes sont introduites dans un analyseur de masse, cela s'appelle " protéomique descendante " et lorsque les protéines sont digérées en peptides plus petits et ensuite introduites dans le spectromètre de masse, cela s'appelle " protéomique ascendante ". La protéomique shotgun est une variante de la protéomique ascendante dans laquelle les protéines d'un mélange sont digérées avant la séparation et la spectrométrie de masse en tandem.
La spectrométrie de masse en tandem peut produire une étiquette de séquence peptidique qui peut être utilisée pour identifier un peptide dans une base de données de protéines. Une notation a été développée pour indiquer les fragments peptidiques qui proviennent d'un spectre de masse en tandem. Les ions fragments peptidiques sont indiqués par a, b ou c si la charge est conservée sur l' extrémité N-terminale et par x, y ou z si la charge est maintenue sur l' extrémité C-terminale . L'indice indique le nombre de résidus d'acides aminés dans le fragment. Des exposants sont parfois utilisés pour indiquer des pertes neutres en plus de la fragmentation du squelette, * pour la perte d'ammoniac et ° pour la perte d'eau. Bien que le clivage du squelette peptidique soit le plus utile pour le séquençage et l'identification des peptides, d'autres ions fragments peuvent être observés dans des conditions de dissociation à haute énergie. Ceux-ci comprennent les ions de perte de chaîne latérale d, v, w et les ions ammonium et des ions fragments supplémentaires spécifiques à la séquence associés à des résidus d'acides aminés particuliers.
Oligosaccharides
Les oligosaccharides peuvent être séquencés en utilisant la spectrométrie de masse en tandem d'une manière similaire au séquençage peptidique. La fragmentation se produit généralement de part et d'autre de la liaison glycosidique (ions b, c, y et z) mais aussi dans des conditions plus énergétiques à travers la structure du cycle sucre dans un clivage en anneau croisé (ions x). Encore une fois, des indices de fin sont utilisés pour indiquer la position du clivage le long de la chaîne. Pour les ions de clivage de cycle croisé, la nature du clivage de cycle croisé est indiquée par les exposants précédents.
Oligonucléotides
La spectrométrie de masse en tandem a été appliquée au séquençage de l' ADN et de l' ARN . Une notation pour la fragmentation en phase gazeuse des ions oligonucléotides a été proposée.
Dépistage néonatal
Le dépistage néonatal est le processus de dépistage des nouveau-nés de maladies génétiques , endocrinologiques , métaboliques et hématologiques traitables . Le développement du dépistage par spectrométrie de masse en tandem au début des années 1990 a conduit à une large expansion des maladies métaboliques congénitales potentiellement détectables qui affectent les taux sanguins d'acides organiques.
Limitation
La spectrométrie de masse en tandem ne peut pas être appliquée aux analyses monocellulaires car elle est insensible à l'analyse de si petites quantités d'une cellule. Ces limitations sont principalement dues à une combinaison de production d'ions inefficace et de pertes d'ions dans les instruments dues aux sources de bruit chimique des solvants.
Perspectives d'avenir
La spectrométrie de masse en tandem sera un outil utile pour la caractérisation des protéines, des complexes nucléoprotéiques et d'autres structures biologiques. Cependant, certains défis restaient tels que l'analyse quantitative et qualitative de la caractérisation du protéome.
Voir également
- Accélérateur de spectrométrie de masse
- Coupe transversale (physique)
- Spectrométrie d'énergie ionique-cinétique analysée en masse
- Décomposition d'ions unimoléculaires
Les références
Bibliographie
- McLuckey SA , Busch KL, Glish GL (1988). Spectrométrie de masse/spectrométrie de masse : techniques et applications de la spectrométrie de masse en tandem . New York, NY : Éditeurs VCH. ISBN 978-0-89573-275-0.
- McLuckey SA , Glish GL. Spectrométrie de masse/Spectrométrie de masse : Techniques et applications du tandem . Chichester : John Wiley & Fils. ISBN 978-0-471-18699-1.
- McLafferty FW (1983). Spectrométrie de masse en tandem . New York : Wiley. ISBN 978-0-471-86597-1.
- Sherman NE, Kinter M (2000). Séquençage et identification de protéines par spectrométrie de masse en tandem . New York : John Wiley. ISBN 978-0-471-32249-8.





![{\displaystyle [{\ce {M}}+n{\ce {H}}]^{n+}+{\ce {e^{-}->}}\left[[{\ce {M}} +(n-1){\ce {H}}]^{(n-1)+}\right]^{*}{\ce {->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/80535d501352b7854827d81bd70a7811d203d639)
![{\displaystyle [{\ce {M}}+n{\ce {H}}]^{n+}+{\ce {A^{-}->}}\left[[{\ce {M}} +(n-1){\ce {H}}]^{(n-1)+}\right]^{*}+{\ce {A->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a0f39c0e76fd3f9d3862b8b49cb144a43c1cd907)
![{\displaystyle [{\ce {M}}-n{\ce {H}}]^{n-}+{\ce {A+->}}\left[[{\ce {M}}-n{ \ce {H}}]^{(n+1)-}\right]^{*}+{\ce {A->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/383244602d9ce4607f68c0696b8c55fb10951a3f)
![{\displaystyle {\ce {{[{M}+H]^{1}+}+He+->}}\left[{\ce {[{M}+H]^{2}+}}\right ]^{*}+{\ce {Il^{0}->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a8ccd097f31299c546cd6e591efb6ed4f8556e2e)