Remodelage de la chromatine - Chromatin remodeling
Le remodelage de la chromatine est la modification dynamique de l' architecture de la chromatine pour permettre l'accès de l'ADN génomique condensé aux protéines de la machinerie régulatrice de la transcription et ainsi contrôler l'expression des gènes. Un tel remodelage est principalement effectué par 1) des modifications covalentes des histones par des enzymes spécifiques, par exemple les histones acétyltransférases (HAT), des désacétylases, des méthyltransférases et des kinases, et 2) des complexes de remodelage de la chromatine dépendant de l'ATP qui déplacent, éjectent ou restructurent les nucléosomes.. En plus de réguler activement l'expression des gènes, le remodelage dynamique de la chromatine confère un rôle régulateur épigénétique dans plusieurs processus biologiques clés, la réplication et la réparation de l'ADN des ovules ; apoptose; la ségrégation chromosomique ainsi que le développement et la pluripotence. Les aberrations dans les protéines de remodelage de la chromatine sont associées à des maladies humaines, y compris le cancer. Le ciblage des voies de remodelage de la chromatine évolue actuellement comme une stratégie thérapeutique majeure dans le traitement de plusieurs cancers.
Aperçu

La régulation transcriptionnelle du génome est contrôlée principalement au stade de pré - initiation par la liaison des protéines de la machinerie transcriptionnelle centrale (à savoir, l'ARN polymérase, les facteurs de transcription, les activateurs et les répresseurs) à la séquence du promoteur central sur la région codante de l'ADN. Cependant, l'ADN est étroitement emballé dans le noyau à l'aide de protéines d'emballage, principalement des protéines histones, pour former des unités répétitives de nucléosomes qui se regroupent pour former une structure de chromatine condensée. Une telle structure condensée obstrue de nombreuses régions régulatrices de l'ADN, ne leur permettant pas d'interagir avec les protéines de la machinerie transcriptionnelle et de réguler l'expression des gènes. Pour surmonter ce problème et permettre un accès dynamique à l'ADN condensé, un processus connu sous le nom de remodelage de la chromatine modifie l'architecture des nucléosomes pour exposer ou masquer des régions d'ADN pour la régulation transcriptionnelle.
Par définition , le remodelage de la chromatine est le processus assisté par une enzyme pour faciliter l'accès à l'ADN nucléosomique en remodelant la structure, la composition et le positionnement des nucléosomes.
Classification
L'accès à l'ADN nucléosomique est régi par deux grandes classes de complexes protéiques :
- Complexes covalents modificateurs d'histones.
- Complexes de remodelage de la chromatine dépendant de l'ATP.
Complexes covalents modificateurs d'histones
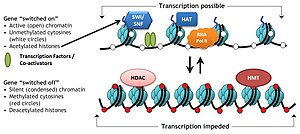
Des complexes protéiques spécifiques, appelés complexes modificateurs d'histones, catalysent l'ajout ou l'élimination de divers éléments chimiques sur les histones. Ces modifications enzymatiques comprennent l' acétylation , la méthylation , la phosphorylation et l' ubiquitination et se produisent principalement au niveau des queues d'histone N-terminales. De telles modifications affectent l'affinité de liaison entre les histones et l'ADN, et ainsi le relâchement ou le resserrement de l'ADN condensé enroulé autour des histones, par exemple, la méthylation de résidus de lysine spécifiques dans H3 et H4 provoque une condensation supplémentaire de l'ADN autour des histones, et empêche ainsi la liaison des facteurs de transcription à l'ADN qui conduit à la répression des gènes. Au contraire, l'acétylation des histones détend la condensation de la chromatine et expose l'ADN à la liaison du TF, entraînant une augmentation de l'expression des gènes.
Modifications connues
Les modifications bien caractérisées des histones comprennent :
Les résidus lysine et arginine sont connus pour être méthylés. Les lysines méthylées sont les marques les mieux comprises du code des histones, car les lysines méthylées spécifiques correspondent bien aux états d'expression des gènes. La méthylation des lysines H3K4 et H3K36 est corrélée à l'activation transcriptionnelle tandis que la déméthylation de H3K4 est corrélée à l'extinction de la région génomique. La méthylation des lysines H3K9 et H3K27 est corrélée à la répression transcriptionnelle. En particulier, H3K9me3 est fortement corrélé avec l'hétérochromatine constitutive.
- Acétylation - par HAT (histone acétyl transférase) ; désacétylation - par HDAC (histone désacétylase)
L'acétylation a tendance à définir "l'ouverture" de la chromatine, car les histones acétylées ne peuvent pas s'emballer aussi bien que les histones désacétylées.
Cependant, il existe de nombreuses autres modifications des histones et les approches de spectrométrie de masse sensible ont récemment considérablement élargi le catalogue.
Hypothèse du code histone
Le code histone est une hypothèse selon laquelle la transcription de l'information génétique codée dans l'ADN est en partie régulée par des modifications chimiques des protéines histones, principalement à leurs extrémités non structurées. Avec des modifications similaires telles que la méthylation de l'ADN, il fait partie du code épigénétique .
Des preuves cumulatives suggèrent qu'un tel code est écrit par des enzymes spécifiques qui peuvent (par exemple) méthyler ou acétyler l'ADN (« écrivains »), éliminés par d'autres enzymes ayant une activité de déméthylase ou de désacétylase (« gommes »), et finalement facilement identifiés par des protéines ( » lecteurs») qui sont recrutés pour de telles modifications d'histones et se lient via des domaines spécifiques, par exemple, bromodomaine, chromodomaine. Cette triple action d'« écriture », de « lecture » et d'« effacement » établit l'environnement local favorable à la régulation transcriptionnelle, à la réparation des dommages à l'ADN, etc.
Le concept critique de l' hypothèse du code des histones est que les modifications des histones servent à recruter d'autres protéines par reconnaissance spécifique de l'histone modifiée via des domaines protéiques spécialisés à ces fins, plutôt que par la simple stabilisation ou déstabilisation de l'interaction entre les histones et l'ADN sous-jacent. Ces protéines recrutées agissent alors pour modifier activement la structure de la chromatine ou pour favoriser la transcription.
Un résumé très basique du code histone pour le statut d'expression génique est donné ci-dessous (la nomenclature des histones est décrite ici ):
| Type de modification |
Histone | ||||||
|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H4K20 | H2BK5 | |
| mono- méthylation | Activation | Activation | Activation | Activation | Activation | Activation | |
| diméthylation | répression | répression | Activation | ||||
| triméthylation | Activation | répression | répression | activation, répression |
répression | ||
| acétylation | Activation | Activation | |||||
Remodelage de la chromatine dépendant de l'ATP
Les complexes de remodelage de la chromatine dépendant de l'ATP régulent l'expression des gènes en déplaçant, en éjectant ou en restructurant les nucléosomes. Ces complexes protéiques ont un domaine ATPase commun et l'énergie provenant de l'hydrolyse de l'ATP permet à ces complexes de remodelage de repositionner les nucléosomes (souvent appelés « glissement de nucléosome ») le long de l'ADN, d'éjecter ou d'assembler des histones on/off de l'ADN ou de faciliter l'échange d'histone variants, et créant ainsi des régions d'ADN sans nucléosome pour l'activation des gènes. En outre, plusieurs remodeleurs ont une activité de translocation d'ADN pour effectuer des tâches de remodelage spécifiques.
Tous les complexes de remodelage de la chromatine dépendant de l'ATP possèdent une sous-unité d'ATPase qui appartient à la superfamille des protéines SNF2. En association avec l'identité de la sous-unité, deux groupes principaux ont été classés pour ces protéines. Ceux-ci sont connus sous le nom de groupe SWI2/SNF2 et de groupe imitation SWI (ISWI). La troisième classe de complexes ATP-dépendants qui a été récemment décrite contient une ATPase de type Snf2 et démontre également une activité désacétylase.
Complexes connus de remodelage de la chromatine
Il existe au moins cinq familles de remodeleurs de la chromatine chez les eucaryotes : SWI/SNF , ISWI , NuRD /Mi-2/ CHD , INO80 et SWR1 avec les deux premiers remodeleurs étant très bien étudiés jusqu'à présent, en particulier dans le modèle de la levure. Bien que tous les remodeleurs partagent un domaine ATPase commun, leurs fonctions sont spécifiques en fonction de plusieurs processus biologiques (réparation de l'ADN, apoptose, etc.). Cela est dû au fait que chaque complexe de remodelage possède des domaines protéiques uniques ( Hélicase , bromodomaine , etc.) dans leur région ATPase catalytique et possède également différentes sous-unités recrutées.
Fonctions spécifiques
- Plusieurs expériences in vitro suggèrent que les remodeleurs ISWI organisent le nucléosome sous une forme de faisceau appropriée et créent un espacement égal entre les nucléosomes, tandis que les remodeleurs SWI/SNF troublent les nucléosomes.
- Il a été démontré que les remodeleurs de la famille ISWI jouent un rôle central dans l'assemblage de la chromatine après la réplication de l'ADN et le maintien des structures de la chromatine d'ordre supérieur.
- Les remodeleurs de la famille INO80 et SWI/SNF participent à la réparation des cassures double brin (DSB) de l'ADN et à la réparation par excision des nucléotides (NER) et jouent ainsi un rôle crucial dans la réponse aux dommages à l'ADN induite par TP53.
- Les complexes de remodelage NuRD/Mi-2/ CHD médient principalement la répression transcriptionnelle dans le noyau et sont nécessaires au maintien de la pluripotence des cellules souches embryonnaires.
Importance
Dans les processus biologiques normaux
Le remodelage de la chromatine joue un rôle central dans la régulation de l'expression des gènes en fournissant à la machinerie de transcription un accès dynamique à un génome autrement étroitement emballé. En outre, le mouvement des nucléosomes par les remodeleurs de la chromatine est essentiel à plusieurs processus biologiques importants, notamment l'assemblage et la ségrégation des chromosomes, la réplication et la réparation de l'ADN, le développement embryonnaire et la pluripotence et la progression du cycle cellulaire. La dérégulation du remodelage de la chromatine entraîne une perte de la régulation transcriptionnelle à ces points de contrôle critiques nécessaires au bon fonctionnement cellulaire, et provoque ainsi divers syndromes pathologiques, y compris le cancer.
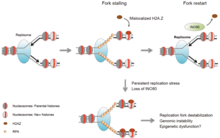
Réponse aux dommages à l'ADN
La relaxation de la chromatine est l'une des premières réponses cellulaires aux dommages de l'ADN. La relaxation semble être initiée par PARP1 , dont l'accumulation au niveau des dommages à l'ADN est à moitié complète 1,6 seconde après que les dommages à l'ADN se soient produits. Ceci est rapidement suivi par l'accumulation du remodeleur de la chromatine Alc1 , qui possède un domaine de liaison à l' ADP-ribose , lui permettant d'être rapidement attiré par le produit de PARP1. Le recrutement maximal d'Alc1 se produit dans les 10 secondes suivant les dommages à l'ADN. Environ la moitié de la relaxation maximale de la chromatine, probablement due à l'action d'Alc1, se produit en 10 secondes. L'action de PARP1 au site d'une cassure double brin permet le recrutement des deux enzymes de réparation de l'ADN MRE11 et NBS1 . La moitié du recrutement maximum de ces deux enzymes de réparation de l'ADN prend 13 secondes pour MRE11 et 28 secondes pour NBS1.
Un autre processus de relaxation de la chromatine, après formation d'une cassure double brin d'ADN, utilise γH2AX, la forme phosphorylée de la protéine H2AX . La variante d' histone H2AX constitue environ 10 % des histones H2A de la chromatine humaine. Le γH2AX (phosphorylé sur la sérine 139 de H2AX) a été détecté 20 secondes après l'irradiation des cellules (avec formation de cassures double brin de l'ADN), et une accumulation à moitié maximale de γH2AX s'est produite en une minute. L'étendue de la chromatine avec le γH2AX phosphorylé est d'environ deux millions de paires de bases au site d'une cassure double brin de l'ADN.
Le H2AX ne provoque pas en lui-même la décondensation de la chromatine, mais quelques secondes après l'irradiation, la protéine « Mediator of the DNA Damage Checkpoint 1 » ( MDC1 ) s'attache spécifiquement au γH2AX. Ceci s'accompagne d'une accumulation simultanée de la protéine RNF8 et de la protéine de réparation de l'ADN NBS1 qui se lient à MDC1 alors que MDC1 se fixe à γH2AX. RNF8 médie une décondensation étendue de la chromatine, par son interaction ultérieure avec la protéine CHD4 , un composant du complexe de remodelage nucléosomique et de désacétylase NuRD . L'accumulation de CHD4 au site de la cassure double brin est rapide, avec une accumulation à moitié maximale se produisant 40 secondes après l'irradiation.
La relaxation initiale rapide de la chromatine lors des dommages à l'ADN (avec une initiation rapide de la réparation de l'ADN) est suivie d'une recondensation lente, la chromatine recouvrant un état de compactage proche de son niveau d'avant les dommages en ∼ 20 min.
Cancer
Le remodelage de la chromatine permet d'affiner les étapes cruciales de la croissance et de la division cellulaires, telles que la progression du cycle cellulaire, la réparation de l'ADN et la ségrégation des chromosomes, et exerce donc une fonction antitumorale. Des mutations dans de tels remodeleurs de la chromatine et des modifications d'histones covalentes dérégulées favorisent potentiellement l'autosuffisance dans la croissance cellulaire et échappent aux signaux cellulaires régulateurs de croissance - deux caractéristiques importantes du cancer .
- Des mutations inactivantes dans SMARCB1 , anciennement connu sous le nom de hSNF5/INI1 et un composant du complexe de remodelage SWI/SNF humain ont été trouvées dans un grand nombre de tumeurs rhabdoïdes , affectant généralement la population pédiatrique. Des mutations similaires sont également présentes dans d'autres cancers infantiles, tels que le carcinome du plexus choroïde , le médulloblastome et dans certaines leucémies aiguës. De plus, des études de knock-out chez la souris soutiennent fortement SMARCB1 en tant que protéine suppresseur de tumeur. Depuis l'observation initiale des mutations SMARCB1 dans les tumeurs rhabdoïdes, plusieurs autres sous-unités du complexe de remodelage de la chromatine humaine SWI/SNF ont été trouvées mutées dans un large éventail de néoplasmes.
- La SWI/SNF ATPase BRG1 (ou SMARCA4 ) est l'ATPase de remodelage de la chromatine la plus fréquemment mutée dans le cancer. Les mutations de ce gène ont d'abord été reconnues dans des lignées cellulaires cancéreuses humaines dérivées de la glande surrénale et du poumon. Dans le cancer, les mutations dans BRG1 montrent une préférence inhabituellement élevée pour les mutations faux-sens qui ciblent le domaine ATPase. Les mutations sont enrichies en séquences d'ATPase hautement conservées, qui se trouvent sur des surfaces fonctionnelles importantes telles que la poche d'ATP ou la surface de liaison à l'ADN. Ces mutations agissent de manière génétiquement dominante pour altérer la fonction régulatrice de la chromatine au niveau des amplificateurs et des promoteurs.
- La protéine de fusion PML-RAR dans la leucémie myéloïde aiguë recrute des histones désacétylases. Cela conduit à la répression du gène responsable de la différenciation des myélocytes, conduisant à la leucémie.
- La protéine Rb suppresseur de tumeur fonctionne par le recrutement des homologues humains des enzymes SWI/SNF BRG1, histone désacétylase et ADN méthyltransférase. Des mutations dans BRG1 sont rapportées dans plusieurs cancers provoquant une perte de l'action suppresseur de tumeur de Rb.
- Des rapports récents indiquent une hyperméthylation de l'ADN dans la région promotrice des principaux gènes suppresseurs de tumeurs dans plusieurs cancers. Bien que peu de mutations soient encore rapportées dans les histones méthyltransférases, une corrélation entre l'hyperméthylation de l'ADN et la méthylation de l'histone H3 lysine-9 a été rapportée dans plusieurs cancers, principalement dans les cancers colorectaux et du sein.
- Les mutations des histones acétyl transférases (HAT) p300 (de type faux-sens et tronquant) sont le plus souvent rapportées dans les carcinomes colorectaux, pancréatiques, mammaires et gastriques. La perte d'hétérozygotie dans la région codante de p300 (chromosome 22q13) est présente dans un grand nombre de glioblastomes.
- En outre, les HAT ont un rôle divers en tant que facteurs de transcription en plus d'avoir une activité d'histone acétylase, par exemple, la sous-unité HAT, hADA3 peut agir comme une protéine adaptatrice liant les facteurs de transcription avec d'autres complexes HAT. En l'absence de hADA3, l'activité transcriptionnelle de TP53 est significativement réduite, suggérant le rôle de hADA3 dans l'activation de la fonction de TP53 en réponse à des dommages à l'ADN.
- De même, TRRAP, l'homologue humain de la levure Tra1, s'est avéré interagir directement avec c-Myc et E2F1 - oncoprotéines connues.
Génomique du cancer
Les progrès rapides de la génomique du cancer et les méthodes de séquençage à haut débit ChIP-chip , ChIP-Seq et Bisulfite permettent de mieux comprendre le rôle du remodelage de la chromatine dans la régulation transcriptionnelle et le rôle dans le cancer.
Intervention thérapeutique
L'instabilité épigénétique causée par la dérégulation du remodelage de la chromatine est étudiée dans plusieurs cancers, dont le cancer du sein, le cancer colorectal, le cancer du pancréas. Une telle instabilité provoque en grande partie un silence généralisé des gènes avec un impact primaire sur les gènes suppresseurs de tumeurs. Par conséquent, des stratégies sont maintenant essayées pour surmonter le silençage épigénétique avec une combinaison synergique d' inhibiteurs d'HDAC ou d' agents HDI et déméthylants de l'ADN . Les IDH sont principalement utilisés comme traitement d'appoint dans plusieurs types de cancer. Les inhibiteurs d'HDAC peuvent induire l' expression de p21 (WAF1), un régulateur de l'activité suppresseur de tumeur de p53 . Les HDAC sont impliquées dans la voie par laquelle la protéine du rétinoblastome (pRb) supprime la prolifération cellulaire . L'œstrogène est bien établi en tant que facteur mitogène impliqué dans la tumorigenèse et la progression du cancer du sein via sa liaison au récepteur des œstrogènes alpha (ERα). Des données récentes indiquent que l'inactivation de la chromatine médiée par l'HDAC et la méthylation de l'ADN est un élément essentiel de l'inhibition de l'ERα dans les cellules cancéreuses du sein humaines.
- Utilisation approuvée :
- Vorinostat a été autorisé par la FDA des États-Unis en octobre 2006 pour le traitement du lymphome cutané à cellules T (CTCL).
- La romidepsine (nom commercial Istodax) a été autorisée par la FDA américaine en novembre 2009 pour le lymphome cutané à cellules T (CTCL).
- Essais cliniques de phase III :
- Le panobinostat (LBH589) fait l'objet d'essais cliniques pour divers cancers, y compris un essai de phase III pour le lymphome cutané à cellules T (CTCL).
- Acide valproïque (sous forme de valproate de magnésium) dans les essais de phase III pour le cancer du col de l'utérus et le cancer de l'ovaire .
- Début des essais cliniques pivots de phase II
- Belinostat (PXD101) a eu un essai de phase II pour rechuté cancer de l' ovaire , et rapporté de bons résultats pour le lymphome des lymphocytes T .
- Inhibiteurs HDAC
Les candidats de premier plan actuels pour de nouvelles cibles médicamenteuses sont les histones lysine méthyltransférases (KMT) et les protéines arginine méthyltransférases (PRMT).
Autres syndromes pathologiques
- Le syndrome d'ATRX (retard mental lié à l'X de la -thalassémie) et le syndrome de la myélodysplasie de la -thalassémie sont dus à des mutations de l' ATRX , une ATPase liée à SNF2 avec un PHD.
- Le syndrome de CHARGE , une maladie autosomique dominante, a été récemment lié à une haploinsuffisance de CHD7 , qui code pour l' ATPase CHD7 de la famille CHD .
Sénescence
Le remodelage architectural de la chromatine est impliqué dans le processus de sénescence cellulaire , qui est lié au, et pourtant distinct, du vieillissement de l'organisme . La sénescence cellulaire réplicative fait référence à un arrêt permanent du cycle cellulaire où les cellules post- mitotiques continuent d'exister en tant que cellules métaboliquement actives mais ne parviennent pas à proliférer. La sénescence peut survenir en raison de la dégradation associée à l' âge , de l' attrition des télomères , des progérias , des pré-cancéreuses et d'autres formes de dommages ou de maladies. Les cellules sénescentes subissent des changements phénotypiques répressifs distincts, potentiellement pour empêcher la prolifération de cellules endommagées ou cancéreuses, avec une organisation de la chromatine modifiée , des fluctuations dans l'abondance des remodeleurs et des changements dans les modifications épigénétiques. Les cellules sénescentes subissent des modifications du paysage de la chromatine lorsque l' hétérochromatine constitutive migre vers le centre du noyau et déplace l' euchromatine et l'hétérochromatine facultative vers les régions situées à la périphérie du noyau. Cela perturbe les interactions chromatine- lamine et inverse le schéma typiquement observé dans une cellule mitotiquement active. Les domaines individuels associés aux lamines (LAD) et les domaines d'association topologique (TAD) sont perturbés par cette migration qui peut affecter les interactions cis à travers le génome. De plus, il existe un schéma général de perte d' histones canoniques , en particulier en ce qui concerne les histones nucléosomiques H3 et H4 et l'histone de liaison H1. Les variants d'histone avec deux exons sont régulés à la hausse dans les cellules sénescentes pour produire un assemblage de nucléosomes modifié qui contribue à la permissivité de la chromatine aux changements sénescents. Bien que la transcription des protéines histones variantes puisse être élevée, les protéines histones canoniques ne sont pas exprimées car elles ne sont produites que pendant la phase S du cycle cellulaire et les cellules sénescentes sont post-mitotiques. Au cours de la sénescence, des portions de chromosomes peuvent être exportées du noyau pour une dégradation lysosomale, ce qui entraîne un plus grand désarroi organisationnel et une perturbation des interactions de la chromatine.
L'abondance du remodeleur de la chromatine peut être impliquée dans la sénescence cellulaire, car le knockdown ou le knock - out des remodeleurs dépendants de l'ATP tels que NuRD, ACF1 et SWI/SNP peut entraîner des dommages à l'ADN et des phénotypes sénescents chez la levure, C. elegans, les souris et les cultures de cellules humaines. ACF1 et NuRD sont régulés à la baisse dans les cellules sénescentes, ce qui suggère que le remodelage de la chromatine est essentiel pour maintenir un phénotype mitotique. Les gènes impliqués dans la signalisation de la sénescence peuvent être réduits au silence par la confirmation de la chromatine et les complexes répressifs polycomb comme on le voit dans le silence PRC1/PCR2 de p16 . L'épuisement du remodeleur spécifique entraîne l'activation de gènes prolifératifs par incapacité à maintenir le silence. Certains remodeleurs agissent sur les régions amplificatrices des gènes plutôt que sur les loci spécifiques pour empêcher la rentrée dans le cycle cellulaire en formant des régions d'hétérochromatine dense autour des régions régulatrices.
Les cellules sénescentes subissent des fluctuations généralisées des modifications épigénétiques dans des régions spécifiques de la chromatine par rapport aux cellules mitotiques. Les cellules humaines et murines subissant une sénescence réplicative subissent une diminution globale générale de la méthylation ; cependant, des loci spécifiques peuvent différer de la tendance générale. Des régions spécifiques de la chromatine, en particulier celles autour des promoteurs ou des amplificateurs des loci prolifératifs, peuvent présenter des états de méthylation élevés avec un déséquilibre global des modifications des histones répressives et activatrices. Les gènes prolifératifs peuvent montrer des augmentations de la marque répressive H3K27me3 tandis que les gènes impliqués dans le silence ou les produits histones aberrants peuvent être enrichis avec la modification activatrice H3K4me3 . De plus, la régulation positive des histones désacétylases, telles que les membres de la famille des sirtuines , peut retarder la sénescence en éliminant les groupes acétyle qui contribuent à une plus grande accessibilité à la chromatine. La perte générale de méthylation, combinée à l'ajout de groupes acétyle, entraîne une conformation de la chromatine plus accessible avec une propension à la désorganisation par rapport aux cellules mitotiquement actives. La perte générale d'histones empêche l'ajout de modifications d'histones et contribue à des changements d'enrichissement dans certaines régions de la chromatine pendant la sénescence.
Voir également
- Épigénétique
- Histone
- Nucléosomes
- Chromatine
- Histone acétyltransférase
- Facteurs de transcription
- CAF-1 (Chromatin assembly factor-1) - chaperon d'histones qui joue un rôle de coordination dans le remodelage de la chromatine.
Les références
Lectures complémentaires
- Chen T, Dent SY (février 2014). "Modificateurs et remodeleurs de la chromatine : régulateurs de la différenciation cellulaire" . Nature Avis Génétique . 15 (2) : 93-106. doi : 10.1038/nrg3607 . PMC 3999985 . PMID 24366184 .
Liens externes
- MBInfo - Chromatine
- MBInfo - Emballage ADN
- YouTube - Chromatine, Histones et Modifications
- YouTube – Présentation de l'épigénétique
- Chromatine + remodelage à la National Library of Medicine Medical Subject Headings (MeSH) des États-Unis