Nucléosome - Nucleosome

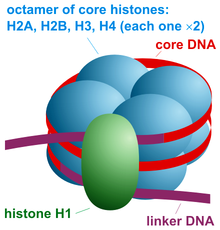
Un nucléosome est l'unité structurelle de base de l' emballage de l' ADN chez les eucaryotes . La structure d'un nucléosome consiste en un segment d'ADN enroulé autour de huit protéines histones et ressemble à un fil enroulé autour d'une bobine. Le nucléosome est la sous-unité fondamentale de la chromatine . Chaque nucléosome est composé d'un peu moins de deux tours d'ADN enroulés autour d'un ensemble de huit protéines appelées histones, connues sous le nom d' octamère d'histones . Chaque octamère d'histone est composé de deux copies chacune des protéines d'histone H2A , H2B , H3 et H4 .
L'ADN doit être compacté en nucléosomes pour s'adapter au noyau cellulaire . En plus de l'enveloppement des nucléosomes, la chromatine eucaryote est encore compactée en étant repliée en une série de structures plus complexes, formant finalement un chromosome . Chaque cellule humaine contient environ 30 millions de nucléosomes.
On pense que les nucléosomes sont porteurs d' informations héritées épigénétiquement sous la forme de modifications covalentes de leurs histones centrales . Les positions des nucléosomes dans le génome ne sont pas aléatoires, et il est important de savoir où se trouve chaque nucléosome car cela détermine l'accessibilité de l'ADN aux protéines régulatrices .
Les nucléosomes ont été observés pour la première fois sous forme de particules au microscope électronique par Don et Ada Olins en 1974, et leur existence et leur structure (en tant qu'octamères d'histones entourés d'environ 200 paires de bases d'ADN) ont été proposées par Roger Kornberg . Le rôle du nucléosome en tant que répresseur général des gènes a été démontré par Lorch et al. in vitro, et par Han et Grunstein in vivo en 1987 et 1988, respectivement.
La particule centrale du nucléosome se compose d'environ 146 paires de bases (pb) d' ADN enveloppées dans 1,67 tours superhélicoïdaux gauchers autour d'un octamère d' histones , composé de 2 copies chacune des histones centrales H2A , H2B , H3 et H4 . Les particules centrales sont reliées par des tronçons d' ADN de liaison , qui peuvent mesurer jusqu'à 80 pb de long. Techniquement, un nucléosome est défini comme la particule centrale plus une de ces régions de liaison ; cependant, le mot est souvent synonyme de particule centrale. Des cartes de positionnement des nucléosomes à l'échelle du génome sont désormais disponibles pour de nombreux organismes modèles, notamment le foie et le cerveau de souris.
Les histones de liaison telles que H1 et ses isoformes sont impliquées dans la compaction de la chromatine et se trouvent à la base du nucléosome près de l'entrée et de la sortie de l'ADN se liant à la région de liaison de l'ADN. Les nucléosomes non condensés sans l'histone de liaison ressemblent à des « billes sur une chaîne d'ADN » au microscope électronique .
Contrairement à la plupart des cellules eucaryotes, les spermatozoïdes matures utilisent largement des protamines pour emballer leur ADN génomique, très probablement pour atteindre un rapport d'encapsidation encore plus élevé. Des équivalents d'histones et une structure de chromatine simplifiée ont également été trouvés chez Archaea , suggérant que les eucaryotes ne sont pas les seuls organismes à utiliser des nucléosomes.
Structure
Structure de la particule centrale
Aperçu
Des études structurelles pionnières menées dans les années 1980 par le groupe d'Aaron Klug ont fourni la première preuve qu'un octamère de protéines histones enroule l'ADN autour de lui en environ 1,7 tour d'une superhélice gauche. En 1997, la première structure cristalline à résolution atomique du nucléosome a été résolue par le groupe de Richmond, montrant les détails les plus importants de la particule. L' ADN palindromique du satellite alpha humain essentiel à la réalisation de la structure cristalline du nucléosome de 1997 a été développé par le groupe Bunick du Oak Ridge National Laboratory dans le Tennessee. Les structures de plus de 20 particules de noyau de nucléosomes différentes ont été résolues à ce jour, y compris celles contenant des variants d'histones et des histones de différentes espèces. La structure de la particule centrale du nucléosome est remarquablement conservée, et même un changement de plus de 100 résidus entre les histones de grenouille et de levure donne des cartes de densité électronique avec un écart quadratique moyen global de seulement 1,6 Å.
La particule centrale du nucléosome (NCP)
La particule centrale du nucléosome (illustrée sur la figure) se compose d'environ 146 paires de bases d'ADN enveloppées dans 1,67 tours superhélicoïdaux gauchers autour de l' octamère d'histone , composé de 2 copies chacune des histones centrales H2A , H2B , H3 et H4 . Les nucléosomes adjacents sont reliés par un tronçon d'ADN libre appelé ADN de liaison (qui varie de 10 à 80 pb de longueur selon l'espèce et le type de tissu). L'ensemble de la structure génère un cylindre de 11 nm de diamètre et de 5,5 nm de hauteur.

Des particules de noyau de nucléosome sont observées lorsque la chromatine en interphase est traitée pour provoquer le déploiement partiel de la chromatine. L'image résultante, via un microscope électronique, est "des perles sur une ficelle". La chaîne est l'ADN, tandis que chaque perle du nucléosome est une particule centrale. La particule centrale du nucléosome est composée d'ADN et de protéines histones.
La digestion partielle de la chromatine par la DNAse révèle sa structure nucléosomique. Parce que les portions d'ADN des particules centrales du nucléosome sont moins accessibles pour la DNAse que les sections de liaison, l'ADN est digéré en fragments de longueurs égales à la multiplicité de la distance entre les nucléosomes (180, 360, 540 paires de bases, etc.). Par conséquent, un motif très caractéristique similaire à une échelle est visible lors de l'électrophorèse sur gel de cet ADN. Une telle digestion peut également se produire dans des conditions naturelles lors de l' apoptose ("suicide cellulaire" ou mort cellulaire programmée), car l' autodestruction de l'ADN est typiquement son rôle.
Interactions protéiques au sein du nucléosome
Les protéines histones centrales contiennent un motif structurel caractéristique appelé "pli d'histone", qui se compose de trois hélices alpha (α1-3) séparées par deux boucles (L1-2). En solution, les histones forment des hétérodimères H2A-H2B et des hétérotétramères H3-H4. Les histones se dimérisent autour de leurs longues hélices α2 dans une orientation antiparallèle et, dans le cas de H3 et H4, deux de ces dimères forment un faisceau à 4 hélices stabilisé par une interaction étendue H3-H3'. Le dimère H2A/H2B se lie au tétramère H3/H4 en raison des interactions entre H4 et H2B, qui incluent la formation d'un amas hydrophobe. L'octamère d'histone est formé d'un tétramère central H3/H4 pris en sandwich entre deux dimères H2A/H2B. En raison de la charge hautement basique des quatre histones centrales, l'octamère d'histone n'est stable qu'en présence d'ADN ou de concentrations de sel très élevées.
Interactions Histone - ADN
Le nucléosome contient plus de 120 interactions directes protéine-ADN et plusieurs centaines d'interactions médiées par l'eau. Les interactions directes protéine-ADN ne sont pas réparties uniformément sur la surface de l'octamère mais plutôt localisées sur des sites discrets. Ceux-ci sont dus à la formation de deux types de sites de liaison à l'ADN au sein de l'octamère ; le site α1α1, qui utilise l'hélice 1 de deux histones adjacentes, et le site L1L2 formé par les boucles L1 et L2. Les liaisons salines et les liaisons hydrogène entre les groupes basiques et hydroxyles de la chaîne latérale et les amides de la chaîne principale avec les phosphates du squelette de l'ADN forment la majeure partie des interactions avec l'ADN. Ceci est important, étant donné que la distribution omniprésente des nucléosomes le long des génomes exige qu'il s'agisse d'un facteur de liaison à l'ADN non spécifique à la séquence. Bien que les nucléosomes aient tendance à préférer certaines séquences d'ADN à d'autres, ils sont capables de se lier pratiquement à n'importe quelle séquence, ce qui serait dû à la flexibilité dans la formation de ces interactions médiées par l'eau. De plus, des interactions non polaires sont faites entre les chaînes latérales des protéines et les groupes désoxyribose, et une chaîne latérale d'arginine s'intercale dans le petit sillon de l'ADN sur les 14 sites où elle fait face à la surface de l'octamère. La distribution et la force des sites de liaison à l'ADN autour de la surface de l'octamère déforment l'ADN dans le noyau du nucléosome. L'ADN est courbé de manière non uniforme et contient également des défauts de torsion. La torsion de l'ADN de forme B libre en solution est de 10,5 pb par tour. Cependant, la torsion globale de l'ADN nucléosomique n'est que de 10,2 pb par tour, variant d'une valeur de 9,4 à 10,9 pb par tour.
Domaines de queue d'histone
Les extensions de queue d'histone constituent jusqu'à 30% en masse d'histones, mais ne sont pas visibles dans les structures cristallines des nucléosomes en raison de leur grande flexibilité intrinsèque, et ont été considérées comme largement non structurées. Les queues N-terminales des histones H3 et H2B traversent un canal formé par les petits sillons des deux brins d'ADN, dépassant de l'ADN toutes les 20 pb. La queue N-terminale de l'histone H4, d'autre part, a une région d'acides aminés très basiques (16-25), qui, dans la structure cristalline, forme une interaction avec la région de surface très acide d'un dimère H2A-H2B d'un autre nucléosome, étant potentiellement pertinent pour la structure d'ordre supérieur des nucléosomes. On pense que cette interaction se produit également dans des conditions physiologiques et suggère que l' acétylation de la queue H4 déforme la structure d'ordre supérieur de la chromatine.
Structure d'ordre supérieur
L'organisation de l'ADN réalisée par le nucléosome ne permet pas d'expliquer pleinement l'encapsidation de l'ADN observée dans le noyau cellulaire. Un compactage supplémentaire de la chromatine dans le noyau cellulaire est nécessaire, mais il n'est pas encore bien compris. La compréhension actuelle est que les nucléosomes répétés avec un ADN "de liaison" intermédiaire forment une fibre de 10 nm , décrite comme des "perles sur une chaîne", et ont un rapport d'emballage d'environ cinq à dix. Une chaîne de nucléosomes peut être disposée dans une fibre de 30 nm , une structure compactée avec un rapport de tassement de ~50 et dont la formation dépend de la présence de l' histone H1 .
Une structure cristalline d'un tétranucléosome a été présentée et utilisée pour construire une structure proposée de la fibre de 30 nm sous la forme d'une hélice à deux départs. Il existe encore un certain nombre de controverses concernant ce modèle, car il est incompatible avec les données récentes de microscopie électronique . Au-delà de cela, la structure de la chromatine est mal comprise, mais il est classiquement suggéré que la fibre de 30 nm est arrangée en boucles le long d'un échafaudage protéique central pour former de l' euchromatine transcriptionnellement active . Un compactage supplémentaire conduit à une hétérochromatine transcriptionnellement inactive .
Dynamique
Bien que le nucléosome soit un complexe protéine-ADN très stable, il n'est pas statique et il a été démontré qu'il subit un certain nombre de réarrangements structurels différents, notamment le glissement du nucléosome et l'exposition au site de l'ADN. Selon le contexte, les nucléosomes peuvent inhiber ou faciliter la liaison aux facteurs de transcription. Les positions des nucléosomes sont contrôlées par trois contributions majeures : Premièrement, l'affinité de liaison intrinsèque de l'octamère d'histone dépend de la séquence d'ADN. Deuxièmement, le nucléosome peut être déplacé ou recruté par la liaison compétitive ou coopérative d'autres facteurs protéiques. Troisièmement, le nucléosome peut être activement transloqué par des complexes de remodelage ATP-dépendants.
Glissement des nucléosomes
Les travaux réalisés au laboratoire de Bradbury ont montré que les nucléosomes reconstitués sur la séquence de positionnement de l'ADN 5S étaient capables de se repositionner traductionnellement sur des séquences adjacentes lorsqu'ils étaient incubés thermiquement. Des travaux ultérieurs ont montré que ce repositionnement ne nécessitait pas de rupture de l'octamère d'histone, mais était compatible avec le fait que les nucléosomes pouvaient "glisser" le long de l'ADN en cis . En 2008, il a en outre été révélé que les sites de liaison CTCF agissent comme des ancres de positionnement des nucléosomes de sorte que, lorsqu'ils sont utilisés pour aligner divers signaux génomiques, plusieurs nucléosomes flanquants peuvent être facilement identifiés. Bien que les nucléosomes soient intrinsèquement mobiles, les eucaryotes ont développé une grande famille d'enzymes de remodelage de la chromatine dépendantes de l'ATP pour modifier la structure de la chromatine, dont beaucoup le font via le glissement des nucléosomes. En 2012, le laboratoire de Beena Pillai a démontré que le glissement des nucléosomes est l'un des mécanismes possibles pour l'expression à grande échelle de gènes spécifiques aux tissus. Le travail montre que le site de démarrage de la transcription pour les gènes exprimés dans un tissu particulier est appauvri en nucléosomes tandis que le même ensemble de gènes dans d'autres tissus où ils ne sont pas exprimés sont liés aux nucléosomes.
Exposition de sites d'ADN
Les travaux du laboratoire Widom ont montré que l'ADN nucléosomique est en équilibre entre un état emballé et un état non emballé. Les mesures de ces taux à l'aide du FRET à résolution temporelle ont révélé que l'ADN dans le nucléosome reste entièrement enveloppé pendant seulement 250 ms avant d'être déballé pendant 10 à 50 ms, puis rapidement réemballé. Cela implique que l'ADN n'a pas besoin d'être activement dissocié du nucléosome, mais qu'il y a une fraction de temps significative pendant laquelle il est pleinement accessible. En effet, cela peut être étendu à l'observation que l'introduction d'une séquence de liaison à l'ADN au sein du nucléosome augmente l'accessibilité des régions adjacentes de l'ADN lorsqu'il est lié. Cette propension de l'ADN dans le nucléosome à « respirer » a des conséquences fonctionnelles importantes pour toutes les protéines de liaison à l'ADN qui opèrent dans un environnement de chromatine. En particulier, la respiration dynamique des nucléosomes joue un rôle important dans la restriction de l'avancement de l' ARN polymérase II lors de l'élongation de la transcription.
Région exempte de nucléosomes
Les promoteurs de gènes actifs ont des régions libres de nucléosomes (NFR). Cela permet l'accessibilité de l'ADN du promoteur à diverses protéines, telles que les facteurs de transcription. La région sans nucléosome s'étend généralement sur 200 nucléotides dans S. cerevisae. Les nucléosomes bien positionnés forment les limites de la NFR. Ces nucléosomes sont appelés nucléosome +1 et nucléosome -1 et sont situés respectivement à des distances canoniques en aval et en amont du site de début de la transcription. Le nucléosome +1 et plusieurs nucléosomes en aval ont également tendance à incorporer la variante d'histone H2A.Z.
Structure modulatrice du nucléosome
Les génomes eucaryotes sont ubiquitairement associés à la chromatine ; cependant, les cellules doivent réguler spatialement et temporellement des loci spécifiques indépendamment de la chromatine en vrac. Afin d'atteindre le niveau élevé de contrôle requis pour coordonner les processus nucléaires tels que la réplication, la réparation et la transcription de l'ADN, les cellules ont développé une variété de moyens pour moduler localement et spécifiquement la structure et la fonction de la chromatine. Cela peut impliquer une modification covalente des histones, l'incorporation de variantes d'histones et un remodelage non covalent par des enzymes de remodelage ATP-dépendantes.
Modifications post-traductionnelles des histones
Depuis leur découverte au milieu des années 1960, on a prédit que les modifications des histones affecteraient la transcription. Le fait que la plupart des premières modifications post-traductionnelles trouvées étaient concentrées dans les extensions de la queue qui dépassent du noyau du nucléosome a conduit à deux théories principales concernant le mécanisme de modification des histones. La première des théories a suggéré qu'elles peuvent affecter les interactions électrostatiques entre les queues d'histone et l'ADN pour « desserrer » la structure de la chromatine. Plus tard, il a été proposé que des combinaisons de ces modifications puissent créer des épitopes de liaison avec lesquels recruter d'autres protéines. Récemment, étant donné que davantage de modifications ont été trouvées dans les régions structurées des histones, il a été avancé que ces modifications pourraient affecter les interactions histone-ADN et histone-histone au sein du noyau du nucléosome. Les modifications (telles que l'acétylation ou la phosphorylation) qui abaissent la charge du noyau d'histone globulaire devraient « desserrer » l'association noyau-ADN ; la force de l'effet dépend de l'emplacement de la modification dans le noyau. Certaines modifications se sont avérées corrélées avec le silençage génique ; d'autres semblent être corrélées à l'activation des gènes. Les modifications courantes incluent l' acétylation , la méthylation ou l' ubiquitination de la lysine ; méthylation de l' arginine ; et phosphorylation de la serine . Les informations stockées de cette manière sont considérées comme épigénétiques , car elles ne sont pas codées dans l'ADN mais sont toujours héritées des cellules filles. Le maintien d'un statut réprimé ou activé d'un gène est souvent nécessaire à la différenciation cellulaire .
Variantes d'histones
Bien que les histones soient remarquablement conservées tout au long de l'évolution, plusieurs formes variantes ont été identifiées. Cette diversification de la fonction histone est limitée à H2A et H3, H2B et H4 étant pour la plupart invariants. H2A peut être remplacé par H2AZ (qui conduit à une stabilité réduite des nucléosomes) ou H2AX (qui est associé à la réparation de l'ADN et à la différenciation des cellules T ), tandis que les chromosomes X inactifs chez les mammifères sont enrichis en macroH2A. H3 peut être remplacé par H3.3 (qui est en corrélation avec les gènes d'activation et les éléments régulateurs) et dans les centromères, H3 est remplacé par CENPA .
Remodelage nucléosomique dépendant de l'ATP
Un certain nombre de réactions distinctes sont associées au terme de remodelage de la chromatine dépendant de l'ATP . Il a été démontré que les enzymes de remodelage font glisser les nucléosomes le long de l'ADN, perturbent les contacts histone-ADN au point de déstabiliser le dimère H2A/H2B et génèrent une torsion superhélicoïdale négative dans l'ADN et la chromatine. Récemment, il a été démontré que l'enzyme de remodelage Swr1 introduisait la variante d'histone H2A.Z dans les nucléosomes. À l'heure actuelle, il n'est pas clair si tous ces éléments représentent des réactions distinctes ou simplement des résultats alternatifs d'un mécanisme commun. Ce qui est partagé entre tous, et en fait la caractéristique du remodelage de la chromatine dépendant de l'ATP, c'est qu'ils entraînent tous une accessibilité de l'ADN altérée.
Des études portant sur l'activation des gènes in vivo et, plus étonnant encore, le remodelage in vitro ont révélé que les événements de remodelage de la chromatine et la liaison aux facteurs de transcription sont de nature cyclique et périodique. Bien que les conséquences de cela sur le mécanisme de réaction du remodelage de la chromatine ne soient pas connues, la nature dynamique du système peut lui permettre de répondre plus rapidement aux stimuli externes. Une étude récente indique que les positions des nucléosomes changent de manière significative au cours du développement des cellules souches embryonnaires de souris, et ces changements sont liés à la liaison des facteurs de transcription du développement.
Remodelage dynamique des nucléosomes à travers le génome de la levure
Des études menées en 2007 ont catalogué les positions des nucléosomes chez la levure et montré que les nucléosomes sont épuisés dans les régions promotrices et les origines de réplication . Environ 80% du génome de la levure semble être recouvert de nucléosomes et le modèle de positionnement des nucléosomes est clairement lié aux régions d'ADN qui régulent la transcription , aux régions qui sont transcrites et aux régions qui initient la réplication de l'ADN. Plus récemment, une nouvelle étude a examiné les changements dynamiques dans le repositionnement des nucléosomes lors d'un événement de reprogrammation transcriptionnelle globale afin d'élucider les effets sur le déplacement des nucléosomes lors de modifications transcriptionnelles à l'échelle du génome chez la levure ( Saccharomyces cerevisiae ). Les résultats suggèrent que les nucléosomes localisés dans les régions promotrices sont déplacés en réponse au stress (comme le choc thermique ). De plus, l'élimination des nucléosomes correspond généralement à une activation transcriptionnelle et le remplacement des nucléosomes correspond généralement à une répression transcriptionnelle, probablement parce que les sites de liaison des facteurs de transcription sont devenus plus ou moins accessibles, respectivement. En général, seulement un ou deux nucléosomes ont été repositionnés au niveau du promoteur pour effectuer ces changements transcriptionnels. Cependant, même dans les régions chromosomiques qui n'étaient pas associées à des changements transcriptionnels, un repositionnement des nucléosomes a été observé, suggérant que la couverture et la découverte de l'ADN transcriptionnel ne produisent pas nécessairement un événement transcriptionnel. Après la transcription, la région de l'ADNr doit être protégée de tout dommage, cela suggère que les protéines HMGB jouent un rôle majeur dans la protection de la région sans nucléosome.
Assemblage de nucléosomes in vitro
Les nucléosomes peuvent être assemblés in vitro en utilisant des histones purifiées natives ou recombinantes. Une technique standard de chargement de l'ADN autour des histones implique l'utilisation de la dialyse au sel . Une réaction composée d'octamères d'histones et d'une matrice d'ADN nue peut être incubée ensemble à une concentration en sel de 2 M. En diminuant régulièrement la concentration en sel, l'ADN s'équilibrera jusqu'à une position où il est enroulé autour des octamères d'histones, formant des nucléosomes. Dans des conditions appropriées, ce processus de reconstitution permet de cartographier expérimentalement l'affinité de positionnement des nucléosomes d'une séquence donnée.
Particules de noyau de nucléosome réticulé au disulfure
Une avancée récente dans la production de particules de noyau de nucléosome avec une stabilité améliorée implique des réticulations disulfure spécifiques au site . Deux réticulations différentes peuvent être introduites dans la particule centrale du nucléosome. Un premier réticule les deux copies de H2A via une cystéine introduite (N38C) résultant en un octamère d'histones qui est stable contre la perte de dimères H2A/H2B lors de la reconstitution du nucléosome. Une seconde réticulation peut être introduite entre la queue d'histone H3 N-terminale et les extrémités d'ADN du nucléosome via un nucléotide convertible incorporé. La réticulation ADN-histone octamère stabilise la particule centrale du nucléosome contre la dissociation de l'ADN à de très faibles concentrations de particules et à des concentrations élevées de sel.
Assemblage de nucléosomes in vivo
Les nucléosomes sont l'unité d'emballage de base de l'ADN construit à partir de protéines histones autour desquelles l'ADN est enroulé. Ils servent d'échafaudage pour la formation d'une structure de chromatine d'ordre supérieur ainsi que pour une couche de contrôle régulateur de l'expression des gènes. Les nucléosomes sont rapidement assemblés sur l'ADN nouvellement synthétisé derrière la fourche de réplication.
H3 et H4
Les histones H3 et H4 d'anciens nucléosomes désassemblés sont conservées à proximité et distribuées de manière aléatoire sur l'ADN nouvellement synthétisé. Ils sont assemblés par le complexe du facteur d'assemblage de la chromatine-1 (CAF-1), qui se compose de trois sous-unités (p150, p60 et p48). H3 et H4 nouvellement synthétisés sont assemblés par le facteur d'assemblage de couplage de réplication (RCAF). RCAF contient la sous-unité Asf1, qui se lie aux protéines H3 et H4 nouvellement synthétisées. Les anciennes protéines H3 et H4 conservent leurs modifications chimiques ce qui contribue à la transmission de la signature épigénétique. Les protéines H3 et H4 nouvellement synthétisées sont progressivement acétylées au niveau de différents résidus de lysine dans le cadre du processus de maturation de la chromatine. On pense également que les anciennes protéines H3 et H4 des nouveaux nucléosomes recrutent des enzymes modifiant les histones qui marquent les nouvelles histones, contribuant ainsi à la mémoire épigénétique.
H2A et H2B
Contrairement aux anciennes H3 et H4, les anciennes protéines histones H2A et H2B sont libérées et dégradées ; par conséquent, les protéines H2A et H2B nouvellement assemblées sont incorporées dans de nouveaux nucléosomes. H2A et H2B sont assemblés en dimères qui sont ensuite chargés sur les nucléosomes par la protéine d'assemblage des nucléosomes-1 (NAP-1) qui aide également au glissement des nucléosomes. Les nucléosomes sont également espacés par des complexes de remodelage des nucléosomes dépendant de l'ATP contenant des enzymes telles que Isw1 Ino80 et Chd1, puis assemblés dans une structure d'ordre supérieur.
Galerie
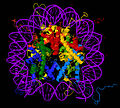
La structure cristalline de la particule centrale du nucléosome ( PDB : 1EQZ ) - différentes vues montrant les détails du repliement et de l'organisation des histones. Les histones H2A , H2B , H3 , H4 et l' ADN sont colorées.
Voir également
Les références
Liens externes
- MBInfo - Que sont les nucléosomes
- Nucléosomes sur le site du Richmond Lab
- Nucléosomes de Proteopedia
- Nucléosome à l'APB
- Remodelage dynamique de nucléosomes individuels à travers un génome eucaryote en réponse à une perturbation transcriptionnelle
- Données et outils de positionnement des nucléosomes en ligne (liste annotée, constamment mise à jour)
- Structure de la protéine histone
- HistoneDB 2.0 - Base de données des histones et variantes au NCBI