Réplication de l'ADN eucaryote - Eukaryotic DNA replication
La réplication de l'ADN eucaryote est un mécanisme conservé qui limite la réplication de l'ADN à une fois par cycle cellulaire. Réplication de l' ADN eucaryote chromosomique d' ADN est essentielle pour la duplication d'une cellule et est nécessaire pour le maintien de la eucaryote génome .
La réplication de l'ADN est l'action des ADN polymérases synthétisant un brin d'ADN complémentaire du brin matrice d'origine. Pour synthétiser l'ADN, l'ADN double brin est déroulé par des hélicases à ADN avant les polymérases, formant une fourche de réplication contenant deux matrices simple brin. Les processus de réplication permettent la copie d'une seule double hélice d'ADN en deux hélices d'ADN, qui sont divisées en cellules filles lors de la mitose . Les principales fonctions enzymatiques réalisées au niveau de la fourche de réplication sont bien conservées des procaryotes aux eucaryotes , mais la machinerie de réplication dans la réplication de l'ADN eucaryote est un complexe beaucoup plus vaste, coordonnant de nombreuses protéines sur le site de réplication, formant le réplisome .
Le réplisome est responsable de la copie de l'intégralité de l' ADN génomique dans chaque cellule proliférative. Ce processus permet le passage haute fidélité des informations héréditaires/génétiques de la cellule parentale à la cellule fille et est donc essentiel à tous les organismes. Une grande partie du cycle cellulaire consiste à garantir que la réplication de l'ADN se déroule sans erreur.
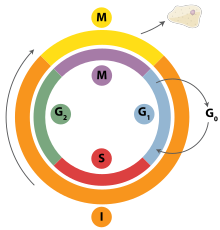
Dans la phase G 1 du cycle cellulaire, de nombreux processus de régulation de la réplication de l'ADN sont initiés. Chez les eucaryotes, la grande majorité de la synthèse de l' ADN se produit pendant la phase S du cycle cellulaire, et le génome entier doit être déroulé et dupliqué pour former deux copies filles. Pendant G 2 , toute erreur d'ADN endommagé ou de réplication est corrigée. Enfin, une copie des génomes est ségréguée dans chaque cellule fille à la mitose ou à la phase M. Ces copies filles contiennent chacune un brin de l'ADN duplex parental et un brin antiparallèle naissant.
Ce mécanisme est conservé des procaryotes aux eucaryotes et est connu sous le nom de réplication semi-conservative de l' ADN. Le processus de réplication semi-conservatrice pour le site de réplication de l'ADN est une structure d'ADN en forme de fourche, la fourche de réplication, où l'hélice d'ADN est ouverte ou déroulée, exposant les nucléotides d' ADN non appariés pour la reconnaissance et l'appariement de bases pour l'incorporation de nucléotides libres en double -ADN brin.
Initiation
L'initiation de la réplication de l'ADN eucaryote est la première étape de la synthèse de l'ADN où la double hélice d'ADN est déroulée et un événement d'amorçage initial par l'ADN polymérase se produit sur le brin principal. L'événement d'amorçage sur le brin retardé établit une fourche de réplication. L'amorçage de l'hélice d'ADN consiste en la synthèse d'une amorce d'ARN pour permettre la synthèse d'ADN par l'ADN polymérase . L'amorçage se produit une fois à l'origine sur le brin principal et au début de chaque fragment d'Okazaki sur le brin retardé.
La réplication de l'ADN est initiée à partir de séquences spécifiques appelées origines de réplication , et les cellules eucaryotes ont plusieurs origines de réplication. Pour initier la réplication de l'ADN, plusieurs protéines réplicatives s'assemblent et se dissocient de ces origines réplicatives. Les facteurs individuels décrits ci-dessous travaillent ensemble pour diriger la formation du complexe de pré-réplication (pré-RC), un intermédiaire clé dans le processus d'initiation de la réplication.
L'association du complexe de reconnaissance d'origine (ORC) avec une origine de réplication recrute la protéine du cycle de division cellulaire 6 (Cdc6) pour former une plate-forme pour le chargement des protéines du complexe de maintenance du minichromosome (Mcm 2-7) , facilitée par la licence de la chromatine et l'ADN protéine du facteur de réplication 1 (Cdt1). L'ORC, Cdc6 et Cdt1 ensemble sont nécessaires pour l'association stable du complexe Mcm2-7 avec des origines réplicatives pendant la phase G 1 du cycle cellulaire.
Complexe pré-réplicatif
Les origines eucaryotes de réplication contrôlent la formation d'un certain nombre de complexes protéiques qui conduisent à l'assemblage de deux fourches de réplication bidirectionnelles de l'ADN. Ces événements sont initiés par la formation du complexe de pré-réplication (pré-RC) aux origines de la réplication. Ce processus a lieu au stade G 1 du cycle cellulaire. La formation pré-RC implique l'assemblage ordonné de nombreux facteurs de réplication, y compris le complexe de reconnaissance d'origine (ORC), la protéine Cdc6, la protéine Cdt1 et les protéines de maintenance des minichromosomes (Mcm2-7). Une fois que le pré-RC est formé, l'activation du complexe est déclenchée par deux kinases , la kinase cycline-dépendante 2 (CDK) et la kinase Dbf4-dépendante (DDK) qui aident à faire passer le pré-RC au complexe d'initiation avant l'initiation de Réplication de l'ADN. Cette transition implique l'assemblage ordonné de facteurs de réplication supplémentaires pour dérouler l'ADN et accumuler les multiples ADN polymérases eucaryotes autour de l'ADN déroulé. Au cœur de la question de savoir comment les fourches de réplication bidirectionnelles sont établies au niveau des origines de réplication se trouve le mécanisme par lequel l'ORC recrute deux complexes Mcm2-7 tête à tête à chaque origine de réplication pour former le complexe de pré-réplication.
Complexe de reconnaissance d'origine
La première étape de l'assemblage du complexe de pré-réplication (pré-RC) est la liaison du complexe de reconnaissance d'origine (ORC) à l'origine de réplication. À la fin de la mitose, la protéine Cdc6 rejoint l'ORC lié suivi de la liaison du complexe Cdt1-Mcm2-7. ORC, Cdc6 et Cdt1 sont tous nécessaires pour charger le complexe de maintenance des minichromosomes à six protéines (Mcm 2-7) sur l'ADN. L'ORC est un complexe protéique à six sous-unités, Orc1p-6, qui sélectionne les sites d'origine réplicative sur l'ADN pour l'initiation de la réplication et la liaison de l'ORC à la chromatine est régulée tout au long du cycle cellulaire. Généralement, la fonction et la taille des sous-unités ORC sont conservées dans de nombreux génomes eucaryotes, la différence étant leurs sites de liaison à l'ADN divergents.
Le complexe de reconnaissance d'origine le plus largement étudié est celui de Saccharomyces cerevisiae ou levure qui est connu pour se lier à la séquence à réplication autonome (ARS). L' ORC de S. cerevisiae interagit spécifiquement avec les éléments A et B1 des origines de réplication de levure, couvrant une région de 30 paires de bases . La liaison à ces séquences nécessite de l' ATP .
La structure atomique de l' ORC de S. cerevisiae liée à l'ADN de l'ARS a été déterminée. Orc1, Orc2, Orc3, Orc4 et Orc5 encerclent l'élément A au moyen de deux types d'interactions, une base non spécifique et une base spécifique, qui plient l'ADN au niveau de l'élément A. Les cinq sous-unités entrent en contact avec l'épine dorsale du phosphate de sucre en plusieurs points de l'élément A pour former une prise serrée sans spécificité de base. Orc1 et Orc2 entrent en contact avec le petit sillon de l'élément A tandis qu'un domaine d'hélice ailée d'Orc4 entre en contact avec les groupes méthyle des invariants Ts dans le grand sillon de l'élément A via une hélice d'insertion (IH). L'absence de cette IH chez les métazoaires explique le manque de spécificité de séquence dans l'ORC humain. L'ADN de l'ARS est également courbé au niveau de l'élément B1 par le biais d'interactions avec Orc2, Orc5 et Orc6. La courbure de l'ADN d'origine par ORC semble être conservée au cours de l'évolution, ce qui suggère qu'elle pourrait être requise pour le mécanisme de chargement du complexe Mcm2-7.
Lorsque l'ORC se lie à l'ADN aux origines de réplication, il sert d'échafaudage pour l'assemblage d'autres facteurs d'initiation clés du complexe pré-réplicatif. Cet assemblage complexe pré-réplicatif au cours du stade G 1 du cycle cellulaire est nécessaire avant l'activation de la réplication de l'ADN au cours de la phase S. L'élimination d'au moins une partie du complexe (Orc1) du chromosome à la métaphase fait partie de la régulation de l'ORC des mammifères pour garantir que la formation de complexe pré-réplicatif avant l'achèvement de la métaphase est éliminée.
Protéine Cdc6
La liaison de la protéine du cycle de division cellulaire 6 (Cdc6) au complexe de reconnaissance d'origine (ORC) est une étape essentielle dans l'assemblage du complexe de pré-réplication (pré-RC) aux origines de réplication. Cdc6 se lie à l'ORC sur l'ADN d'une manière dépendante de l'ATP, ce qui induit un changement dans le modèle de liaison d'origine qui nécessite Orc1 ATPase . Cdc6 nécessite ORC pour s'associer à la chromatine et est à son tour requis pour que l'heptamère Cdt1-Mcm2-7 se lie à la chromatine. Le complexe ORC-Cdc6 forme une structure en forme d'anneau et est analogue à d'autres machines à protéines dépendantes de l'ATP. Les niveaux et l'activité de Cdc6 régulent la fréquence avec laquelle les origines de réplication sont utilisées au cours du cycle cellulaire.
Protéine Cdt1
La licence de chromatine et la protéine du facteur 1 de réplication de l'ADN (Cdt1) sont nécessaires pour la licence de la chromatine pour la réplication de l'ADN. Dans S. cerevisiae , Cdt1 facilite le chargement du complexe Mcm2-7 un à la fois sur le chromosome en stabilisant la structure en anneau ouvert gauche de l'hexamère unique Mcm2-7. Il a été démontré que Cdt1 s'associe à l' extrémité C de Cdc6 pour promouvoir de manière coopérative l'association des protéines Mcm à la chromatine. La structure cryo-EM du complexe OCCM (ORC-Cdc6-Cdt1-MCM) montre que le Cdt1-CTD interagit avec le Mcm6-WHD. Chez les métazoaires, l'activité de Cdt1 au cours du cycle cellulaire est étroitement régulée par son association avec la protéine géminine , qui à la fois inhibe l'activité de Cdt1 pendant la phase S afin d'empêcher la réplication de l'ADN et l'empêche d' ubiquitination et de protéolyse ultérieure .
Complexe de protéines d'entretien des minichromosomes
Les protéines de maintenance des minichromosomes (Mcm) ont été nommées d'après un criblage génétique de mutants d'initiation de la réplication de l'ADN chez S. cerevisiae qui affectent la stabilité du plasmide d'une manière spécifique à l'ARS . Mcm2, Mcm3, Mcm4, Mcm5, Mcm6 et Mcm7 forment un complexe hexamérique qui a une structure en anneau ouvert avec un espace entre Mcm2 et Mcm5. L'assemblage des protéines Mcm sur la chromatine nécessite la fonction coordonnée du complexe de reconnaissance d'origine (ORC), Cdc6 et Cdt1. Une fois que les protéines Mcm ont été chargées sur la chromatine, ORC et Cdc6 peuvent être retirés de la chromatine sans empêcher la réplication ultérieure de l'ADN. Cette observation suggère que le rôle principal du complexe de pré-réplication est de charger correctement les protéines Mcm.
Les protéines Mcm sur la chromatine forment un double hexamère tête à tête avec les deux anneaux légèrement inclinés, tordus et décentrés pour créer un pli dans le canal central où l'ADN lié est capturé à l'interface des deux anneaux. Chaque anneau hexamérique Mcm2-7 sert d'abord d'échafaudage pour l'assemblage du réplisome, puis de noyau de l'hélicase catalytique CMG (Cdc45-MCM-GINS), qui est un composant principal du réplisome. Chaque protéine Mcm est étroitement liée à toutes les autres, mais des séquences uniques distinguant chacun des types de sous-unités sont conservées chez les eucaryotes. Tous les eucaryotes ont exactement six analogues de protéines Mcm qui appartiennent chacun à l'une des classes existantes (Mcm2-7), indiquant que chaque protéine Mcm a une fonction unique et importante.
Les protéines de maintenance des minichromosomes sont nécessaires à l'activité de l'ADN hélicase. L'inactivation de l'une des six protéines Mcm pendant la phase S empêche la progression ultérieure de la fourche de réplication, ce qui suggère que l'hélicase ne peut pas être recyclée et doit être assemblée aux origines de réplication. En plus de l'activité hélicase du complexe protéique de maintenance des minichromosomes, le complexe possède également une activité ATPase associée. Une mutation dans l'une des six protéines Mcm réduit les sites de liaison à l'ATP conservés, ce qui indique que l'hydrolyse de l'ATP est un événement coordonné impliquant les six sous-unités du complexe Mcm. Des études ont montré qu'au sein du complexe protéique Mcm se trouvent des paires catalytiques spécifiques de protéines Mcm qui fonctionnent ensemble pour coordonner l'hydrolyse de l'ATP. Par exemple, Mcm3 mais pas Mcm6 peut activer l'activité Mcm6. Ces études, confirmées par les structures cryo-EM des complexes Mcm2-7, suggèrent que le complexe Mcm est un hexamère avec Mcm3 à côté de Mcm7 , Mcm2 à côté de Mcm6 et Mcm4 à côté de Mcm5 . Les deux membres de la paire catalytique contribuent à la conformation qui permet la liaison et l'hydrolyse de l'ATP et le mélange de sous-unités actives et inactives crée une activité ATPase coordonnée qui permet au complexe protéique Mcm de terminer la liaison et l'hydrolyse de l'ATP dans son ensemble.
La localisation nucléaire des protéines de maintenance des minichromosomes est régulée dans les cellules de levure en herbe. Les protéines Mcm sont présentes dans le noyau au stade G 1 et en phase S du cycle cellulaire, mais sont exportées vers le cytoplasme au cours du stade G 2 et de la phase M. Un complexe Mcm complet et intact à six sous-unités est nécessaire pour entrer dans le noyau cellulaire. Chez S. cerevisiae , l'exportation nucléaire est favorisée par l'activité de la kinase dépendante de la cycline (CDK). Les protéines Mcm associées à la chromatine sont protégées de la machinerie d'exportation de CDK en raison du manque d'accessibilité à CDK.
Complexe d'initiation
Au cours de l' étape G 1 du cycle cellulaire, les facteurs d'initiation de la réplication, le complexe de reconnaissance d'origine (ORC), Cdc6, Cdt1 et le complexe protéique de maintenance des minichromosomes (Mcm), se lient séquentiellement à l'ADN pour former le complexe de pré-réplication (pré-RC ). Au passage du stade G 1 à la phase S du cycle cellulaire, la protéine kinase dépendante de la cycline (CDK) et la kinase Cdc7/Dbf4 (DDK) spécifiques à la phase S transforment la pré-RC en une fourche de réplication active. Au cours de cette transformation, le pré-RC est désassemblé avec la perte de Cdc6, créant le complexe d'initiation. En plus de la liaison des protéines Mcm, la protéine du cycle de division cellulaire 45 (Cdc45) est également essentielle pour initier la réplication de l'ADN. Des études ont montré que Mcm est essentiel pour le chargement de Cdc45 sur la chromatine et ce complexe contenant à la fois Mcm et Cdc45 est formé au début de la phase S du cycle cellulaire. Cdc45 cible le complexe protéique Mcm, qui a été chargé sur la chromatine, en tant que composant de la pré-RC à l'origine de la réplication au cours du stade G 1 du cycle cellulaire.
protéine Cdc45
La protéine du cycle de division cellulaire 45 (Cdc45) est un composant essentiel pour la conversion du complexe pré-réplicatif en complexe d'initiation. La protéine Cdc45 s'assemble aux origines de réplication avant l'initiation et est nécessaire pour que la réplication commence dans Saccharomyces cerevisiae , et a un rôle essentiel pendant l'élongation. Ainsi, Cdc45 joue un rôle central dans les phases d'initiation et d'élongation de la réplication de l'ADN chromosomique.
Cdc45 s'associe à la chromatine après le début de l'initiation à la fin du stade G 1 et pendant la phase S du cycle cellulaire. Cdc45 s'associe physiquement à Mcm5 et affiche des interactions génétiques avec cinq des six membres de la famille des gènes Mcm et le gène ORC2 . Le chargement de Cdc45 sur la chromatine est essentiel pour charger d'autres protéines de réplication, notamment l' ADN polymérase , l'ADN polymérase , la protéine de réplication A (RPA) et l'antigène nucléaire de prolifération cellulaire (PCNA) sur la chromatine. Dans un système sans noyau de Xenopus , il a été démontré que Cdc45 est nécessaire pour le déroulement de l'ADN plasmidique. Le système sans noyau de Xenopus démontre également que le déroulement de l'ADN et la liaison étroite de la RPA à la chromatine ne se produisent qu'en présence de Cdc45.
La liaison de Cdc45 à la chromatine dépend de l'activité de la kinase Clb-Cdc28 ainsi que des fonctions Cdc6 et Mcm2, ce qui suggère que Cdc45 s'associe à la pré-RC après activation des kinases dépendantes de la cycline (CDK) en phase S. Comme indiqué par le timing et la dépendance à CDK, la liaison de Cdc45 à la chromatine est cruciale pour l'engagement à l'initiation de la réplication de l'ADN. Pendant la phase S, Cdc45 interagit physiquement avec les protéines Mcm sur la chromatine ; cependant, la dissociation du Cdc45 de la chromatine est plus lente que celle du Mcm, ce qui indique que les protéines sont libérées par des mécanismes différents.
GINS
Les six protéines de maintenance des minichromosomes et Cdc45 sont essentielles lors de l'initiation et de l'élongation pour le mouvement des fourches de réplication et pour le déroulement de l'ADN. Les GINS sont essentiels pour l'interaction de Mcm et de Cdc45 aux origines de la réplication lors de l'initiation puis au niveau des fourches de réplication de l'ADN au fur et à mesure que le réplisome progresse. Le complexe GINS est composé de quatre petites protéines Sld5 (Cdc105), Psf1 (Cdc101), Psf2 (Cdc102) et Psf3 (Cdc103), GINS représente 'go, ichi, ni, san' qui signifie '5, 1, 2, 3 ' en japonais. Cdc45, Mcm2-7 et GINS forment ensemble l'hélicase CMG, l'hélicase réplicative du réplisome. Bien que le complexe Mcm2-7 seul ait une faible activité hélicase, Cdc45 et GINS sont nécessaires pour une activité hélicase robuste
Mcm10
Mcm10 est essentiel pour la réplication des chromosomes et interagit avec l'hélicase de maintenance des minichromosomes 2-7 qui est chargée sous une forme inactive aux origines de la réplication de l'ADN. Mcm10 chaperonne également l'ADN polymérase catalytique et aide à stabiliser la polymérase au niveau des fourches de réplication.
DDK et CDK kinases
Au début de la phase S, le complexe pré-réplicatif doit être activé par deux kinases spécifiques de la phase S afin de former un complexe d'initiation à une origine de réplication. Une kinase est la kinase Cdc7-Dbf4 appelée kinase dépendante de Dbf4 (DDK) et l'autre est la kinase dépendante de la cycline (CDK). Des tests de liaison à la chromatine de Cdc45 chez la levure et le Xénope ont montré qu'un événement en aval de l'action de CDK est le chargement de Cdc45 sur la chromatine. Cdc6 a été supposé être une cible de l'action de CDK, en raison de l'association entre Cdc6 et CDK, et de la phosphorylation dépendante de CDK de Cdc6. La phosphorylation CDK-dépendante de Cdc6 a été considérée comme nécessaire pour l'entrée dans la phase S.
Les sous-unités catalytiques de DDK, Cdc7, et la protéine activatrice, Dbf4, sont conservées chez les eucaryotes et sont nécessaires à l'apparition de la phase S du cycle cellulaire. DDK et Cdc7 sont nécessaires pour le chargement de Cdc45 sur les origines de réplication de la chromatine. La cible de liaison de la DDK kinase est le complexe Mcm, éventuellement Mcm2. DDK cible le complexe Mcm, et sa phosphorylation conduit à l'activation possible de l'activité hélicase Mcm.
Protéines Dpb11, Sld3 et Sld2
Sld3, Sld2 et Dpb11 interagissent avec de nombreuses protéines de réplication. Sld3 et Cdc45 forment un complexe associé à la pré-RC aux premières origines de réplication même dans la phase G1 1 et aux dernières origines de réplication dans la phase S d'une manière mutuellement dépendante de Mcm. Dpb11 et Sld2 interagissent avec la polymérase et des expériences de réticulation ont indiqué que Dpb11 et la polymérase coprécipitent dans la phase S et s'associent à des origines de réplication.
Sld3 et Sld2 sont phosphorylés par CDK, ce qui permet aux deux protéines réplicatives de se lier à Dpb11. Dpbl1 avait deux paires de domaines BRCA1 C Terminus (BRCT) qui sont connus sous le nom de domaines de liaison aux phosphopeptides. La paire N-terminale des domaines BRCT se lie à Sld3 phosphorylé et la paire C-terminale se lie à Sld2 phosphorylé. Ces deux interactions sont essentielles pour l'activation dépendante de CDK du bourgeonnement de l'ADN chez la levure.
Dpb11 interagit également avec le GINS et participe aux étapes d'initiation et d'élongation de la réplication de l'ADN chromosomique. Les GINS sont l'une des protéines de réplication trouvées au niveau des fourches de réplication et forment un complexe avec Cdc45 et Mcm.
Ces interactions dépendantes de la phosphorylation entre Dpb11, Sld2 et Sld3 sont essentielles pour l'activation dépendante de CDK de la réplication de l'ADN, et en utilisant des réactifs de réticulation dans certaines expériences, un complexe fragile a été identifié appelé le complexe de pré-chargement (pré-LC) . Ce complexe contient Pol ɛ, GINS, Sld2 et Dpb11. On constate que la pré-LC se forme avant toute association avec les origines d'une manière dépendante de CDK et dépendante de DDK et l'activité de la CDK régule l'initiation de la réplication de l'ADN à travers la formation de la pré-LC.
Élongation

La formation du complexe pré-réplicatif (pré-RC) marque les sites potentiels pour l'initiation de la réplication de l'ADN. En accord avec le complexe de maintenance du minichromosome entourant l'ADN double brin, la formation du pré-RC ne conduit pas au déroulement immédiat de l'ADN d'origine ou au recrutement d'ADN polymérases. Au lieu de cela, le pré-RC qui se forme pendant la phase G 1 du cycle cellulaire n'est activé que pour dérouler l'ADN et initier la réplication une fois que les cellules sont passées de la phase G 1 à la phase S du cycle cellulaire.
Une fois que le complexe d'initiation est formé et que les cellules passent en phase S, le complexe devient alors un réplisome. Le complexe de réplisome eucaryote est responsable de la coordination de la réplication de l'ADN. La réplication sur les brins avant et arrière est effectuée par l'ADN polymérase et l'ADN polymérase δ. De nombreux facteurs réplisomes, notamment Claspin, And1, le chargeur de pince du facteur de réplication C et le complexe de protection de la fourche sont responsables de la régulation des fonctions de la polymérase et de la coordination de la synthèse de l'ADN avec le déroulement du brin matrice par le complexe Cdc45-Mcm-GINS. Au fur et à mesure que l'ADN se déroule, le nombre de torsion diminue. Pour compenser cela, le nombre de contorsions augmente, introduisant des superbobines positives dans l'ADN. Ces superbobines entraîneraient l'arrêt de la réplication de l'ADN si elles n'étaient pas supprimées. Les topoisomérases sont responsables de l'élimination de ces superbobines avant la fourche de réplication.
Le réplisome est responsable de la copie de l'intégralité de l'ADN génomique dans chaque cellule proliférative. Les réactions d'appariement des bases et de formation de chaînes, qui forment l'hélice fille, sont catalysées par des ADN polymérases. Ces enzymes se déplacent le long de l'ADN simple brin et permettent l'extension du brin d'ADN naissant en « lisant » le brin matrice et en permettant l'incorporation des nucléobases puriques appropriées , adénine et guanine , et des nucléobases pyrimidiques , thymine et cytosine . Les désoxyribonucléotides libres activés existent dans la cellule sous forme de désoxyribonucléotides triphosphates (dNTP). Ces nucléotides libres sont ajoutés à un groupe 3'-hydroxyle exposé sur le dernier nucléotide incorporé. Dans cette réaction, un pyrophosphate est libéré du dNTP libre, générant de l'énergie pour la réaction de polymérisation et exposant le 5' monophosphate, qui est ensuite lié de manière covalente à l'oxygène 3'. De plus, les nucléotides mal insérés peuvent être retirés et remplacés par les nucléotides corrects dans une réaction énergétiquement favorable. Cette propriété est vitale pour une relecture et une réparation correctes des erreurs qui se produisent lors de la réplication de l'ADN.
Fourche de réplication
La fourche de réplication est la jonction entre les brins de matrice nouvellement séparés, connus sous le nom de brins principaux et retardés, et l'ADN double brin. Étant donné que l'ADN duplex est antiparallèle, la réplication de l'ADN se produit dans des directions opposées entre les deux nouveaux brins au niveau de la fourche de réplication, mais toutes les ADN polymérases synthétisent l'ADN dans le sens 5' à 3' par rapport au brin nouvellement synthétisé. Une coordination supplémentaire est nécessaire pendant la réplication de l'ADN. Deux polymérases réplicatives synthétisent l'ADN dans des orientations opposées. La polymérase synthétise l'ADN sur le brin d'ADN "principal" en continu car il pointe dans la même direction que l'ADN se déroulant par le réplisome. En revanche, la polymérase synthétise l'ADN sur le brin « retardé », qui est le brin opposé de la matrice d'ADN, de manière fragmentée ou discontinue.
Les tronçons discontinus de produits de réplication d'ADN sur le brin retardé sont connus sous le nom de fragments d'Okazaki et ont une longueur d'environ 100 à 200 bases au niveau des fourches de réplication eucaryotes. Le brin retardé contient généralement des segments plus longs d'ADN simple brin recouverts de protéines de liaison simple brin, qui aident à stabiliser les matrices simple brin en empêchant la formation d'une structure secondaire. Chez les eucaryotes, ces protéines de liaison simple brin sont un complexe hétérotrimérique connu sous le nom de protéine de réplication A (RPA).
Chaque fragment d'Okazaki est précédé d'une amorce d'ARN, qui est déplacée par le cortège du fragment d'Okazaki suivant lors de la synthèse. La RNase H reconnaît les hybrides ADN:ARN qui sont créés par l'utilisation d'amorces ARN et est responsable de leur élimination du brin répliqué, laissant une jonction amorce:matrice. L'ADN polymérase , reconnaît ces sites et allonge les cassures laissées par l'élimination des amorces. Dans les cellules eucaryotes, une petite quantité du segment d'ADN immédiatement en amont de l'amorce d'ARN est également déplacée, créant une structure de volet. Ce lambeau est ensuite clivé par des endonucléases. Au niveau de la fourche de réplication, l'espace dans l'ADN après le retrait du rabat est scellé par l' ADN ligase I , qui répare les entailles laissées entre le 3'-OH et le 5' phosphate du brin nouvellement synthétisé. En raison de la nature relativement courte du fragment eucaryote d'Okazaki, la synthèse de la réplication de l'ADN se produisant de manière discontinue sur le brin retardé est moins efficace et prend plus de temps que la synthèse du brin leader. La synthèse d'ADN est terminée une fois que toutes les amorces d'ARN sont retirées et que les entailles sont réparées.
Fil conducteur
Au cours de la réplication de l'ADN, le réplisome déroulera l'ADN duplex parental en une fourche de réplication de matrice d'ADN à deux brins dans une direction 5' à 3'. Le brin principal est le brin modèle qui est répliqué dans la même direction que le mouvement de la fourche de réplication. Cela permet au brin nouvellement synthétisé complémentaire du brin d'origine d'être synthétisé de 5' à 3' dans la même direction que le mouvement de la fourche de réplication.
Une fois qu'une amorce d'ARN a été ajoutée par une primase à l'extrémité 3' du brin de tête, la synthèse d'ADN se poursuivra dans une direction 3' à 5' par rapport au brin de tête sans interruption. L'ADN polymérase ajoutera en continu des nucléotides au brin matrice, ce qui fait que la synthèse du brin principal ne nécessite qu'une seule amorce et possède une activité ADN polymérase ininterrompue.
brin en retard
La réplication de l'ADN sur le brin retardé est discontinue. Dans la synthèse des brins en retard, le mouvement de l' ADN polymérase dans la direction opposée de la fourche de réplication nécessite l'utilisation de plusieurs amorces d'ARN . L'ADN polymérase synthétisera de courts fragments d'ADN appelés fragments d'Okazaki qui sont ajoutés à l'extrémité 3' de l'amorce. Ces fragments peuvent avoir une longueur comprise entre 100 et 400 nucléotides chez les eucaryotes.
À la fin de la synthèse du fragment d'Okazaki, l'ADN polymérase pénètre dans le fragment d'Okazaki précédent et déplace son extrémité 5' contenant l'amorce d'ARN et un petit segment d'ADN. Cela génère un volet simple brin ARN-ADN, qui doit être clivé, et l'entaille entre les deux fragments d'Okazaki doit être scellée par de l'ADN ligase I. Ce processus est connu sous le nom de maturation du fragment d'Okazaki et peut être géré de deux manières : un mécanisme volets courts, tandis que l'autre traite des volets longs. L'ADN polymérase est capable de déplacer jusqu'à 2 à 3 nucléotides d'ADN ou d'ARN avant sa polymérisation, générant un substrat de « rabat » court pour Fen1 , qui peut éliminer les nucléotides du rabat, un nucléotide à la fois.
En répétant les cycles de ce processus, l'ADN polymérase et Fen1 peuvent coordonner l'élimination des amorces d'ARN et laisser une coupure d'ADN sur le brin retardé. Il a été proposé que ce processus itératif soit préférable à la cellule car il est étroitement régulé et ne génère pas de grands lambeaux qui doivent être excisés. En cas d'activité Fen1/ADN polymérase δ dérégulée, la cellule utilise un mécanisme alternatif pour générer et traiter de longs lambeaux en utilisant l'ADN2, qui possède à la fois des activités hélicase et nucléase. L'activité nucléase de l'ADN2 est nécessaire pour retirer ces longs volets, laissant un volet plus court à traiter par Fen1. Des études en microscopie électronique indiquent que le chargement des nucléosomes sur le brin retardé se produit très près du site de synthèse. Ainsi, la maturation des fragments d'Okazaki est un processus efficace qui se produit immédiatement après la synthèse de l'ADN naissant.
ADN polymérases réplicatives
Une fois que l'hélicase réplicative a déroulé le duplex d'ADN parental, exposant deux matrices d'ADN simple brin, des polymérases réplicatives sont nécessaires pour générer deux copies du génome parental. La fonction de l'ADN polymérase est hautement spécialisée et accomplit la réplication sur des matrices spécifiques et dans des localisations étroites. Au niveau de la fourche de réplication eucaryote, il existe trois complexes polymérases réplicatifs distincts qui contribuent à la réplication de l'ADN : la polymérase , la polymérase et la polymérase ε. Ces trois polymérases sont essentielles à la viabilité de la cellule.
Parce que les ADN polymérases nécessitent une amorce sur laquelle commencer la synthèse d'ADN, la polymérase (Pol α) agit comme une primase réplicative. Pol α est associé à une ARN primase et ce complexe accomplit la tâche d'amorçage en synthétisant une amorce qui contient une courte séquence de 10 nucléotides d'ARN suivie de 10 à 20 bases d'ADN. Il est important de noter que cette action d'amorçage se produit lors de l'initiation de la réplication aux origines pour commencer la synthèse du brin principal et également à l'extrémité 5' de chaque fragment d'Okazaki sur le brin retardé.
Cependant, Pol α n'est pas capable de poursuivre la réplication de l'ADN et doit être remplacée par une autre polymérase pour poursuivre la synthèse de l'ADN. La commutation de la polymérase nécessite des chargeurs de pince et il a été prouvé que la réplication normale de l'ADN nécessite les actions coordonnées des trois ADN polymérases : Pol α pour la synthèse d'amorçage, Pol ε pour la réplication du brin principal et Pol δ, qui est constamment chargé, pour générer Fragments d'Okazaki lors de la synthèse des brins retardés.
- Polymérase (Pol α) : Forme un complexe avec une petite sous-unité catalytique (PriS) et une grande sous-unité non catalytique (PriL). Premièrement, la synthèse d'une amorce d'ARN permet la synthèse d'ADN par l'ADN polymérase alpha. Se produit une fois à l'origine sur le brin principal et au début de chaque fragment d'Okazaki sur le brin retardé. Les sous-unités Pri agissent comme une primase, synthétisant une amorce d'ARN. L'ADN Pol α allonge l'amorce nouvellement formée avec des nucléotides d'ADN. Après environ 20 nucléotides, l'élongation est prise en charge par Pol ε sur le brin leader et Pol δ sur le brin retardé.
- Polymérase δ (Pol δ) : Hautement processif et a une activité de relecture, 3'->5' exonucléase. In vivo, c'est la principale polymérase impliquée à la fois dans lasynthèse des brinsretardés et des brins principaux.
- Polymerase ε (Pol ε) : Hautement processif et a une activité de relecture, 3'->5' exonucléase. Très lié à pol δ, in vivo il fonctionne principalement dans le contrôle d'erreur de pol δ.
Complexe hélicase Cdc45–Mcm–GINS
Les ADN hélicases et polymérases doivent rester en contact étroit au niveau de la fourche de réplication. Si le déroulement se produit trop avant la synthèse, de grandes étendues d'ADN simple brin sont exposées. Cela peut activer la signalisation des dommages à l'ADN ou induire des processus de réparation de l'ADN. Pour contrecarrer ces problèmes, le réplisome eucaryote contient des protéines spécialisées conçues pour réguler l'activité de l'hélicase en amont de la fourche de réplication. Ces protéines fournissent également des sites d'amarrage pour l'interaction physique entre les hélicases et les polymérases, garantissant ainsi que le déroulement du duplex est couplé à la synthèse d'ADN.
Pour que les ADN polymérases fonctionnent, l'hélice d'ADN double brin doit être déroulée pour exposer deux matrices d'ADN simple brin pour la réplication. Les hélicases à ADN sont responsables du déroulement de l'ADN double brin lors de la réplication des chromosomes. Les hélicases dans les cellules eucaryotes sont remarquablement complexes. Le noyau catalytique de l'hélicase est composé de six protéines de maintenance des minichromosomes (Mcm2-7), formant un anneau hexamérique . Loin de l'ADN, les protéines Mcm2-7 forment un seul hétérohexamère et sont chargées sous une forme inactive aux origines de la réplication de l'ADN en tant que double hexamère tête à tête autour de l'ADN double brin. Les protéines Mcm sont recrutées aux origines de réplication puis redistribuées dans tout l'ADN génomique pendant la phase S, indiquant leur localisation à la fourche de réplication.
Le chargement des protéines Mcm ne peut se produire que pendant le G 1 du cycle cellulaire, et le complexe chargé est ensuite activé pendant la phase S par recrutement de la protéine Cdc45 et du complexe GINS pour former l'hélicase Cdc45-Mcm-GINS (CMG) active à fourches de réplication de l'ADN. L'activité Mcm est requise tout au long de la phase S pour la réplication de l'ADN. Divers facteurs régulateurs s'assemblent autour de l'hélicase CMG pour produire le « Complexe de progression du réplisome » qui s'associe aux ADN polymérases pour former le réplisome eucaryote, dont la structure est encore assez mal définie par rapport à son homologue bactérien.
L'hélicase CMG isolée et le complexe Replisome Progression contiennent un seul complexe d'anneaux protéiques Mcm, ce qui suggère que le double hexamère chargé des protéines Mcm à l'origine pourrait être divisé en deux anneaux hexamères simples dans le cadre du processus d'initiation, chaque anneau complexe protéique Mcm formant le noyau d'une hélicase CMG au niveau des deux fourches de réplication établies à partir de chaque origine. Le complexe CMG complet est requis pour le déroulement de l'ADN, et le complexe CDC45-Mcm-GINS est l'ADN hélicase fonctionnelle dans les cellules eucaryotes.
Protéines Ctf4 et And1
Le complexe CMG interagit avec le réplisome par l'interaction avec les protéines Ctf4 et And1. Les protéines Ctf4/And1 interagissent à la fois avec le complexe CMG et l'ADN polymérase . Ctf4 est un facteur accessoire de la polymérase , qui est requis pour le recrutement de la polymérase aux origines de réplication.
Protéines Mrc1 et Claspin
Les protéines Mrc1/Claspin couplent la synthèse du brin principal avec l'activité hélicase du complexe CMG. Mrc1 interagit avec la polymérase ainsi qu'avec les protéines Mcm. L'importance de ce lien direct entre l'hélicase et la polymérase du brin principal est soulignée par les résultats obtenus dans des cellules humaines en culture, où Mrc1/Claspin est nécessaire pour une progression efficace de la fourche de réplication. Ces résultats suggèrent qu'une réplication efficace de l'ADN nécessite également le couplage d'hélicases et la synthèse de brins de tête...
Antigène nucléaire cellulaire proliférant
Les ADN polymérases nécessitent des facteurs supplémentaires pour soutenir la réplication de l'ADN. Les ADN polymérases ont une structure « main » semi-fermée, ce qui permet à la polymérase de se charger sur l'ADN et de commencer la translocation. Cette structure permet à l'ADN polymérase de maintenir la matrice d'ADN simple brin, d'incorporer des dNTP au site actif et de libérer l'ADN double brin nouvellement formé. Cependant, la structure des ADN polymérases ne permet pas une interaction stable continue avec l'ADN matrice.
Pour renforcer l'interaction entre la polymérase et l'ADN matrice, des pinces coulissantes d'ADN s'associent à la polymérase pour favoriser la processivité de la polymérase réplicative. Chez les eucaryotes, la pince coulissante est une structure en anneau homotrimère connue sous le nom d'antigène nucléaire de prolifération cellulaire (PCNA). L'anneau PCNA a une polarité avec des surfaces qui interagissent avec les ADN polymérases et les attache solidement à la matrice d'ADN. La stabilisation dépendante du PCNA des ADN polymérases a un effet significatif sur la réplication de l'ADN car les PCNA sont capables d'augmenter la processivité de la polymérase jusqu'à 1000 fois. Le PCNA est un cofacteur essentiel et a la particularité d'être l'une des plates-formes d'interaction les plus courantes dans le réplisome pour s'adapter à plusieurs processus au niveau de la fourche de réplication. Le PCNA est donc également considéré comme un cofacteur régulateur des ADN polymérases.
Facteur de réplication C
Le PCNA entoure complètement le brin de matrice d'ADN et doit être chargé sur l'ADN au niveau de la fourche de réplication. Au niveau du brin principal, le chargement du PCNA est un processus peu fréquent, car la réplication de l'ADN sur le brin principal est continue jusqu'à ce que la réplication soit terminée. Cependant, au niveau du brin retardé, l'ADN polymérase doit être continuellement chargée au début de chaque fragment d'Okazaki. Cette initiation constante de la synthèse de fragments d'Okazaki nécessite un chargement répété de PCNA pour une réplication efficace de l'ADN.
Le chargement de PCNA est accompli par le complexe du facteur de réplication C (RFC). Le complexe RFC est composé de cinq ATPases : Rfc1, Rfc2, Rfc3, Rfc4 et Rfc5. RFC reconnaît les jonctions amorce-matrice et charge PCNA sur ces sites. L'homotrimère PCNA est ouvert par RFC par hydrolyse d'ATP et est ensuite chargé sur l'ADN dans la bonne orientation pour faciliter son association avec la polymérase. Les chargeurs à pince peuvent également décharger le PCNA de l'ADN ; un mécanisme nécessaire lorsque la réplication doit être terminée.
Fourche de réplication bloquée
La réplication de l'ADN au niveau de la fourche de réplication peut être interrompue par une pénurie de désoxynucléotide triphosphates (dNTP) ou par des dommages à l'ADN, entraînant un stress de réplication . Cet arrêt de la réplication est décrit comme une fourche de réplication bloquée . Un complexe de protection de la fourche de protéines stabilise la fourche de réplication jusqu'à ce que les dommages à l'ADN ou d'autres problèmes de réplication puissent être résolus. Un blocage prolongé de la fourche de réplication peut entraîner d'autres dommages à l'ADN. Les signaux de blocage sont désactivés si les problèmes à l'origine de la fourche de réplication sont résolus.
Résiliation
L'arrêt de la réplication de l'ADN eucaryote nécessite des processus différents selon que les chromosomes sont circulaires ou linéaires. Contrairement aux molécules linéaires, les chromosomes circulaires sont capables de répliquer la molécule entière. Cependant, les deux molécules d'ADN resteront liées entre elles. Cette question est traitée par décaténation des deux molécules d'ADN par une topoisomérase de type II . Les topoisomérases de type II sont également utilisées pour séparer les brins linéaires car ils sont repliés de manière complexe dans un nucléosome à l'intérieur de la cellule.
Comme mentionné précédemment, les chromosomes linéaires sont confrontés à un autre problème qui n'est pas observé dans la réplication circulaire de l'ADN. En raison du fait qu'une amorce d'ARN est requise pour l'initiation de la synthèse d'ADN, le brin retardé est désavantagé dans la réplication du chromosome entier. Alors que le brin principal peut utiliser une seule amorce d'ARN pour étendre l'extrémité 5' du brin d'ADN en réplication, plusieurs amorces d'ARN sont responsables de la synthèse des brins en retard, créant des fragments d'Okazaki. Cela conduit à un problème en raison du fait que l'ADN polymérase ne peut s'ajouter qu'à l'extrémité 3' du brin d'ADN. L'action 3'-5' de l'ADN polymérase le long du brin parent laisse une courte région d'ADN simple brin (ADNsb) à l'extrémité 3' du brin parent lorsque les fragments d'Okazaki ont été réparés. Étant donné que la réplication se produit dans des directions opposées aux extrémités opposées des chromosomes parents, chaque brin est un brin retardé à une extrémité. Au fil du temps, cela entraînerait un raccourcissement progressif des deux chromosomes filles . C'est ce qu'on appelle le problème de réplication de fin.
Le problème de la réplication finale est traité dans les cellules eucaryotes par les régions télomériques et la télomérase . Les télomères prolongent l'extrémité 3' du chromosome parental au-delà de l'extrémité 5' du brin fille. Cette structure d'ADN simple brin peut agir comme une origine de réplication qui recrute la télomérase. La télomérase est une ADN polymérase spécialisée qui se compose de plusieurs sous-unités protéiques et d'un composant ARN. Le composant ARN de la télomérase s'hybride à l'extrémité 3' simple brin de l'ADN matrice et contient 1,5 copie de la séquence télomérique. La télomérase contient une sous-unité protéique qui est une transcriptase inverse appelée transcriptase inverse de la télomérase ou TERT. TERT synthétise l'ADN jusqu'à la fin de l'ARN télomérase matrice puis se désengage. Ce processus peut être répété autant de fois que nécessaire avec l'extension de l'extrémité 3' de la molécule d'ADN parentale. Cette addition 3' fournit une matrice pour l'extension de l'extrémité 5' du brin fille en retardant la synthèse d'ADN du brin. La régulation de l'activité de la télomérase est assurée par des protéines de liaison aux télomères.
Barrières de fourche de réplication
La réplication de l'ADN procaryote est bidirectionnelle; au sein d'une origine réplicative, des complexes de réplisomes sont créés à chaque extrémité de l'origine de réplication et les réplisomes s'éloignent les uns des autres à partir du point de départ initial. Chez les procaryotes, la réplication bidirectionnelle commence à une origine réplicative sur le chromosome circulaire et se termine à un site opposé au début initial de l'origine. Ces régions de terminaison ont des séquences d'ADN connues sous le nom de sites Ter . Ces sites Ter sont liés par la protéine Tus. Le complexe Ter- Tus est capable d'arrêter l'activité de l'hélicase, mettant fin à la réplication.
Dans les cellules eucaryotes, la fin de la réplication se produit généralement par la collision des deux fourches réplicatives entre deux origines de réplication actives. L'emplacement de la collision varie en fonction du moment du tir d'origine. De cette manière, si une fourche de réplication se bloque ou s'effondre sur un certain site, la réplication du site peut être sauvée lorsqu'un réplisome se déplaçant dans la direction opposée termine la copie de la région. Il existe des barrières de fourches de réplication programmées (RFB) liées par des protéines RFB à divers endroits, dans tout le génome, qui sont capables de terminer ou de suspendre les fourches de réplication, arrêtant la progression du réplisome.
Usines de réplication
Il a été constaté que la réplication se produit de manière localisée dans le noyau cellulaire. Contrairement à la vision traditionnelle consistant à déplacer les fourches de réplication le long de l'ADN stagnant, un concept d' usines de réplication a émergé, ce qui signifie que les fourches de réplication sont concentrées vers certaines régions « usines » immobilisées à travers lesquelles les brins d'ADN matrice passent comme des bandes transporteuses.
Régulation du cycle cellulaire
La réplication de l'ADN est un processus étroitement orchestré qui est contrôlé dans le contexte du cycle cellulaire . La progression dans le cycle cellulaire et à son tour la réplication de l'ADN est étroitement régulée par la formation et l'activation de complexes pré-réplicatifs (pré-RC) qui sont obtenus par l'activation et l'inactivation de kinases dépendantes des cyclines (Cdks, CDK). Plus précisément, ce sont les interactions des cyclines et des kinases dépendantes des cyclines qui sont responsables de la transition de la phase G 1 à la phase S.
Au cours de la phase G 1 du cycle cellulaire, il existe de faibles niveaux d'activité CDK. Ce faible niveau d'activité CDK permet la formation de nouveaux complexes pré-RC mais n'est pas suffisant pour que la réplication de l'ADN soit initiée par les pré-RC nouvellement formés. Pendant les phases restantes du cycle cellulaire, il existe des niveaux élevés d'activité CDK. Ce niveau élevé d'activité CDK est responsable de l'initiation de la réplication de l'ADN ainsi que de l'inhibition de la formation de nouveaux complexes pré-RC. Une fois que la réplication de l'ADN a été initiée, le complexe pré-RC est décomposé. En raison du fait que les niveaux de CDK restent élevés pendant les phases S, G 2 et M du cycle cellulaire, aucun nouveau complexe pré-RC ne peut être formé. Tout cela permet de s'assurer qu'aucune initiation ne peut se produire tant que la division cellulaire n'est pas terminée.
En plus des kinases dépendantes de la cycline, on pense qu'un nouveau cycle de réplication est empêché par la régulation négative de Cdt1. Ceci est réalisé via la dégradation de Cdt1 ainsi que par les actions inhibitrices d'une protéine connue sous le nom de géminine . La géminine se lie étroitement à Cdt1 et est considérée comme le principal inhibiteur de la réplication. La géminine apparaît d'abord en phase S et est dégradée à la transition métaphase-anaphase, éventuellement par ubiquination par le complexe promoteur de l'anaphase (APC).
Divers points de contrôle du cycle cellulaire sont présents tout au long du cycle cellulaire et déterminent si une cellule progressera entièrement dans la division. Il est important dans la réplication du G 1 ou restriction, point de contrôle rend la détermination ou non initiation de la réplication commencera ou si la cellule sera placée dans une phase de repos connu sous le nom G 0 . Les cellules au stade G 0 du cycle cellulaire sont empêchées d'initier un cycle de réplication parce que les protéines de maintenance des minichromosomes ne sont pas exprimées. La transition vers la phase S indique que la réplication a commencé.
Protéines de point de contrôle de réplication
Afin de préserver l'information génétique pendant la division cellulaire, la réplication de l'ADN doit être effectuée avec une grande fidélité. Afin d'accomplir cette tâche, les cellules eucaryotes ont des protéines en place à certains moments du processus de réplication qui sont capables de détecter toute erreur lors de la réplication de l'ADN et sont capables de préserver l'intégrité génomique. Ces protéines de point de contrôle sont capables d'empêcher le cycle cellulaire d'entrer en mitose afin de laisser le temps à la réparation de l'ADN. Les protéines de point de contrôle sont également impliquées dans certaines voies de réparation de l'ADN, alors qu'elles stabilisent la structure de la fourche de réplication pour éviter d'autres dommages. Ces protéines de point de contrôle sont essentielles pour éviter de transmettre des mutations ou d'autres aberrations chromosomiques à la descendance.
Les protéines de point de contrôle eucaryotes sont bien conservées et impliquent deux kinases liées à la phosphatidylinositol 3-kinase (PIKK), ATR et ATM . ATR et ATM partagent une séquence de phosphorylation cible, le motif SQ/TQ, mais leurs rôles individuels dans les cellules diffèrent.
L'ATR est impliquée dans l'arrêt du cycle cellulaire en réponse aux cassures double brin de l'ADN. ATR a un partenaire de point de contrôle obligatoire, ATR-interacting-protein (ATRIP), et ensemble, ces deux protéines sont sensibles aux tronçons d'ADN simple brin qui sont recouverts par la protéine de réplication A (RPA). La formation d'ADN simple brin se produit fréquemment, plus souvent lors d'un stress de réplication. ATR-ATRIP est capable d'arrêter le cycle cellulaire pour préserver l'intégrité du génome. L'ATR se trouve sur la chromatine pendant la phase S, similaire à la RPA et au claspin.
La génération de segments d'ADN simple brin est importante pour initier les voies de contrôle en aval des dommages de réplication. Une fois que l'ADN simple brin devient suffisamment long, l'ADN simple brin recouvert de RPA est capable de recruter ATR-ATRIP. Afin de devenir pleinement active, la kinase ATR s'appuie sur des protéines de détection qui détectent si les protéines de point de contrôle sont localisées sur un site valide de stress de réplication de l'ADN. La pince hétérotrimérique RAD9 - HUS1 - Rad1 (9-1-1) et son chargeur de pinces RFC Rad17 sont capables de reconnaître l'ADN coupé ou coupé. Le chargeur de pince RFC Rad17 charge 9-1-1 sur l'ADN endommagé. La présence de 9-1-1 sur l'ADN est suffisante pour faciliter l'interaction entre ATR-ATRIP et un groupe de protéines appelées médiateurs de point de contrôle, telles que TOPBP1 et Mrc1/ claspin . TOPBP1 interagit avec et recrute le composant Rad9 phosphorylé du 9-1-1 et se lie à ATR-ATRIP, qui phosphoryle Chk1. Mrc1/Claspin est également requis pour l'activation complète d'ATR-ATRIP qui phosphoryle Chk1, la principale kinase effectrice du point de contrôle en aval. Claspin est un composant du réplisome et contient un domaine d'amarrage avec Chk1, révélant une fonction spécifique de Claspin lors de la réplication de l'ADN : la promotion de la signalisation du point de contrôle au niveau du réplisome.
La signalisation Chk1 est vitale pour arrêter le cycle cellulaire et empêcher les cellules d'entrer en mitose avec une réplication incomplète de l'ADN ou des dommages à l'ADN. L'inhibition de Cdk dépendante de Chk1 est importante pour le fonctionnement du point de contrôle ATR-Chk1 et pour arrêter le cycle cellulaire et laisser suffisamment de temps pour l'achèvement des mécanismes de réparation de l'ADN, ce qui à son tour empêche la transmission de l'ADN endommagé. De plus, l'inhibition de Cdk dépendante de Chk1 joue un rôle essentiel dans l'inhibition de la décharge d'origine pendant la phase S. Ce mécanisme empêche la synthèse continue de l'ADN et est nécessaire à la protection du génome en présence de stress de réplication et de conditions génotoxiques potentielles. Ainsi, l'activité ATR-Chk1 prévient en outre les problèmes potentiels de réplication au niveau des origines de réplication uniques en inhibant l'initiation de la réplication dans tout le génome, jusqu'à ce que la cascade de signalisation maintenant l'arrêt du cycle cellulaire soit désactivée.
Réplication par les nucléosomes
L'ADN eucaryote doit être étroitement compacté afin de s'adapter à l'espace confiné du noyau. Les chromosomes sont conditionnés en enroulant 147 nucléotides autour d'un octamère de protéines histones , formant un nucléosome . L'octamère du nucléosome comprend deux copies de chaque histone H2A , H2B , H3 et H4 . En raison de l'association étroite des protéines histones à l'ADN, les cellules eucaryotes ont des protéines conçues pour remodeler les histones avant la fourche de réplication, afin de permettre une progression en douceur du réplisome. Il existe également des protéines impliquées dans le réassemblage des histones derrière la fourche de réplication pour rétablir la conformation du nucléosome.
Il existe plusieurs chaperons d'histones qui sont connus pour être impliqués dans l'assemblage des nucléosomes après la réplication. Le complexe FACT s'est avéré interagir avec le complexe ADN polymérase -primase, et les sous-unités du complexe FACT interagissaient génétiquement avec les facteurs de réplication. Le complexe FACT est un hétérodimère qui n'hydrolyse pas l'ATP, mais est capable de faciliter le "desserrage" des histones dans les nucléosomes, mais la façon dont le complexe FACT est capable de soulager l'association étroite des histones pour l'élimination de l'ADN reste sans réponse.
Un autre chaperon d'histones qui s'associe au réplisome est Asf1 , qui interagit avec le complexe Mcm dépendant des dimères d'histones H3-H4. Asf1 est capable de transmettre le dimère H3-H4 nouvellement synthétisé aux facteurs de dépôt derrière la fourche de réplication et cette activité rend les dimères d'histone H3-H4 disponibles sur le site de dépôt d'histone juste après la réplication. Asf1 (et son partenaire Rtt109) a également été impliqué dans l'inhibition de l'expression génique à partir de gènes répliqués pendant la phase S.
Le facteur d'assemblage de la chromatine chaperon hétérotrimérique 1 ( CAF-1 ) est une protéine de formation de la chromatine qui est impliquée dans le dépôt d'histones sur les deux brins d'ADN nouvellement répliqués pour former la chromatine. CAF-1 contient un motif de liaison à PCNA, appelé boîte PIP, qui permet à CAF-1 de s'associer au réplisome via PCNA et est capable de déposer des dimères d'histone H3-H4 sur de l'ADN nouvellement synthétisé. Le chaperon Rtt106 est également impliqué dans ce processus, et associé aux dimères CAF-1 et H3-H4 lors de la formation de la chromatine. Ces processus chargent les histones nouvellement synthétisées sur l'ADN.
Après le dépôt des histones H3-H4, des nucléosomes se forment par l'association des histones H2A-H2B. On pense que ce processus se produit via le complexe FACT, car il est déjà associé au réplisome et est capable de se lier à H2A-H2B libre, ou il existe la possibilité d'un autre chaperon H2A-H2B, Nap1. Des études en microscopie électronique montrent que cela se produit très rapidement, car des nucléosomes peuvent être observés ne formant que quelques centaines de paires de bases après la fourche de réplication. Par conséquent, l'ensemble du processus de formation de nouveaux nucléosomes a lieu juste après la réplication en raison du couplage des chaperons d'histones au réplisome.
Comparaisons entre la réplication de l'ADN procaryote et eucaryote
Par rapport à la réplication de l'ADN procaryote , l'achèvement de la réplication de l'ADN eucaryote est plus complexe et implique de multiples origines de réplication et des protéines réplicatives à accomplir. L'ADN procaryote est arrangé dans une forme circulaire et n'a qu'une seule origine de réplication lorsque la réplication commence. En revanche, l'ADN eucaryote est linéaire. Une fois répliqué, il existe jusqu'à mille origines de réplication.
L'ADN eucaryote est bidirectionnel. Ici, le sens du mot bidirectionnel est différent. L'ADN linéaire eucaryote a de nombreuses origines (appelées O) et extrémités (appelées T). "T" est présent à droite de "O". Un "O" et un "T" forment ensemble un réplicon. Après la formation du complexe de pré-initiation, lorsqu'un réplicon commence l'allongement, l'initiation commence dans le deuxième réplicon. Maintenant, si le premier réplicon se déplace dans le sens des aiguilles d'une montre, le deuxième réplicon se déplace dans le sens inverse des aiguilles d'une montre, jusqu'à ce que le "T" du premier réplicon soit atteint. À "T", les deux réplicons fusionnent pour terminer le processus de réplication. Pendant ce temps, le deuxième réplicon se déplace également vers l'avant, pour rencontrer le troisième réplicon. Ce mouvement dans le sens horaire et antihoraire de deux réplicons est appelé réplication bidirectionnelle.
La réplication de l'ADN eucaryote nécessite une coordination précise de toutes les ADN polymérases et protéines associées pour répliquer l'ensemble du génome chaque fois qu'une cellule se divise. Ce processus est réalisé par une série d'étapes d'assemblages de protéines aux origines de réplication, concentrant principalement la régulation de la réplication de l'ADN sur l'association de l'hélicase MCM avec l'ADN. Ces origines de réplication dirigent le nombre de complexes protéiques qui se formeront pour initier la réplication. Dans la régulation de la réplication de l'ADN procaryote se concentre sur la liaison de la protéine initiatrice DnaA à l'ADN, l'initiation de la réplication se produisant plusieurs fois au cours d'un cycle cellulaire. L'ADN procaryote et eucaryote utilise la liaison et l'hydrolyse de l'ATP pour diriger le chargement de l'hélicase et dans les deux cas, l'hélicase est chargée sous la forme inactive. Cependant, les hélicases eucaryotes sont des hexamères doubles qui sont chargés sur de l'ADN double brin, tandis que les hélicases procaryotes sont des hexamères simples chargés sur de l'ADN simple brin.
La ségrégation des chromosomes est une autre différence entre les cellules procaryotes et eucaryotes. Les cellules à division rapide, telles que les bactéries, commenceront souvent à séparer les chromosomes qui sont encore en cours de réplication. Dans les cellules eucaryotes, la ségrégation des chromosomes dans les cellules filles n'est initiée que lorsque la réplication est complète dans tous les chromosomes. Malgré ces différences, cependant, le processus sous-jacent de réplication est similaire pour l'ADN procaryote et eucaryote.
| Réplication de l'ADN procaryote | Réplication de l'ADN eucaryote |
|---|---|
| Se produit à l'intérieur du cytoplasme | Se produit à l'intérieur du noyau |
| Une seule origine de réplication par molécule d'ADN | Avoir de nombreuses origines de réplication dans chaque chromosome |
| L'origine de la réplication a une longueur d'environ 100 à 200 nucléotides ou plus | Chaque origine de réplication est formée d'environ 150 nucléotides |
| La réplication se produit en un point de chaque chromosome | La réplication se produit en plusieurs points simultanément dans chaque chromosome |
| N'avoir qu'une seule origine de réplication | A plusieurs origines de réplication |
| L'initiation est réalisée par les protéines DnaA et DnaB | L'initiation est réalisée par le Complexe Reconnaissance d'Origine |
| La topoisomérase est nécessaire | La topoisomérase est nécessaire |
| La réplication est très rapide | La réplication est très lente |
Liste des protéines de réplication de l'ADN eucaryote
Liste des principales protéines impliquées dans la réplication de l'ADN eucaryote :
| Protéine | Fonction dans la réplication de l'ADN eucaryote |
|---|---|
| ET 1 | Charge l'ADN polymérase sur la chromatine avec le complexe CMG sur le brin retardé. Aussi connu sous le nom de Ctf4 dans la levure bourgeonnante. |
| Cdc45 | Requis pour les étapes d'initiation et d'élongation de la réplication de l'ADN. Une partie du complexe hélicase Mcm2-7. Nécessaire après l'étape pré-RC pour le chargement de diverses protéines pour l'initiation et l'élongation. |
| Complexe Cdc45-Mcm-GINS (CMG) | ADN hélicase fonctionnelle dans les cellules eucaryotes |
| Cdc6 | Requis pour l'assemblage du complexe Mcm2-7 à ORC, en conjonction avec Cdt1 . |
| Kinase Cdc7-Dbf4 ou kinase dépendante de Dbf4 (DDK) | Protéine kinase nécessaire à l'initiation de la réplication de l'ADN, probablement par phosphorylation des protéines de maintenance des minichromosomes. |
| Cdt1 | Charge le complexe Mcm2-7 sur l'ADN à l'ORC dans l'étape de pré-RC/licence. Inhibée chez les métazoaires par la géminine. |
| fermoir | Coupler la synthèse du brin leader avec l'activité hélicase du complexe CMG. Fonctionne avec Mrc1 |
| Ctf4 | Charge l'ADN polymérase sur la chromatine avec le complexe CMG sur le brin retardé. L'homologue chez les métazoaires est connu sous le nom de AND-1. |
| Kinase dépendante de la cycline (CDK) | Protéine kinase dépendante de la cycline requise pour l'initiation de la réplication et pour d'autres étapes ultérieures. |
| ADN2 | Endonucléase du volet 5' et hélicase impliquées dans le traitement des fragments d'Okazaki. |
| ADN ligase I | Rejoint les fragments d'Okazaki lors de la réplication de l'ADN. L'activité de la ligase est également nécessaire pour la réparation et la recombinaison de l'ADN. |
| ADN polymérase α (Pol α) | Contient une activité primase qui est nécessaire pour initier la synthèse d'ADN sur les brins principaux et retardés. |
| ADN polymérase δ (Pol δ) | Nécessaire pour achever la synthèse des fragments d'Okazaki sur le brin retardé qui ont été amorcés par l'ADN polymérase . |
| ADN polymérase (Pol ε) | La polymérase du brin leader. Synthétise l'ADN à la fourche de réplication. Se lie tôt aux origines via Dbp11 et est nécessaire pour charger l'ADN polymérase α. |
| Dpb11 | Protéine d'initiation de la réplication de l'ADN. Charge l'ADN polymérase sur les complexes de pré-réplication aux origines. |
| Fen1 | Endonucléase du volet 5' impliquée dans le traitement des fragments d'Okazaki. |
| Géminine | Protéine trouvée dans les métazoaires et absente des levures. Se lie à et inactive Cdt1, régulant ainsi la formation de complexes pré-réplicatifs/d'initiation. Également suggéré pour favoriser la formation de pré-RC en se liant et empêchant ainsi la dégradation de Cdt1 |
| GINS | Complexe tétramérique composé de Sld5, Psf1, Psf2, Psf3. S'associe au complexe pré-réplicatif au moment de l'initiation et se déplace avec les fourches de réplication pendant l'étape d'élongation. Requis pour le stade d'élongation de la réplication de l'ADN et peut-être une partie du complexe hélicase Mcm. |
| Protéines de maintenance des minichromosomes (Mcm) | Six protéines différentes de la famille AAA+ ATPase qui forment un hexamère en solution. Cet hexamère est recruté et chargé par ORC, Cdc6 et Cdt1 et forme un double hexamère qui est topologiquement lié autour de l'ADN pour former un complexe pré-réplicatif résistant au sel. Lors de l'initiation de la réplication, Mcm2-7 s'éloigne de l'ORC avec la fourche de réplication. |
| Mcm10 | Requis pour les étapes d'initiation et d'élongation de la réplication de l'ADN. Impliqué dans la liaison à la chromatine de Cdc45 et de l'ADN polymérase . Également requis pour la stabilité de la sous-unité catalytique de l'ADN polymérase dans la levure bourgeonnante S. cerevisiae . |
| Mrc1 | Coupler la synthèse du brin leader avec l'activité hélicase du complexe CMG. L'homologue du métazoaire est connu sous le nom de Claspin. |
| Complexe de reconnaissance d'origine (ORC) | Complexe hétérohexamérique composé de protéines Orc1-Orc6. Se lie à l'ADN et assemble le complexe Mcm2-7 sur la chromatine avec Cdc6 et Cdt1. |
| Antigène nucléaire de prolifération cellulaire (PCNA) | Protéine trimérique avec structure en forme d'anneau, enferme l'ADN empêchant la dissociation de l'ADN polymérase. Agit comme une pince coulissante pour les polymérases δ et ε, améliorant ainsi la processivité des polymérases réplicatives. |
| Facteur de réplication C (RFC) | Charge le PCNA sur des matrices amorcées et est impliqué dans le basculement entre l'ADN polymérase a et les polymérases réplicatives et ε. |
| Barrières de fourche de réplication (RFB) | Lié par des protéines RFB à divers endroits du génome. Sont capables de mettre fin ou de suspendre les fourches de réplication, arrêtant la progression du réplisome. |
| Protéine de réplication A (RPA) | Protéine de liaison monocaténaire hétérotrimérique. Stabilise l'ADN simple brin à la fourche de réplication. |
| RNase H | Ribonucléase qui digère l'ARN hybridé à l'ADN. Impliqué dans le traitement des fragments d'Okazaki. |
| Sld2 | Fonctions dans l'initiation de la réplication. Substrat clé de CDK, la phosphorylation favorise l'interaction avec Dpb11. Requis pour le lancement de la réplication. |
| Sld3 | Fonctions dans l'initiation de la réplication. Substrat clé de CDK, la phosphorylation favorise l'interaction avec Dpb11. Requis pour le lancement de la réplication. |
| télomérase | Une ribonucléoprotéine qui ajoute la séquence d'ADN "TTAGGG" se répète à l'extrémité 3' des brins d'ADN dans les télomères. |
| Topoisomérases | Réguler le surenroulement ou le sous-enroulement de l'ADN |