Structure tertiaire des protéines - Protein tertiary structure

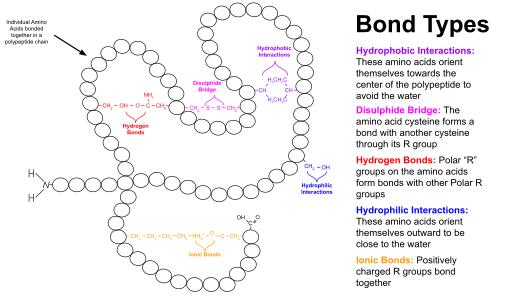
La structure tertiaire des protéines est la forme tridimensionnelle d'une protéine . La structure tertiaire aura une seule chaîne polypeptidique « squelette » avec une ou plusieurs structures secondaires protéiques , les domaines protéiques . Les chaînes latérales d' acides aminés peuvent interagir et se lier de plusieurs manières. Les interactions et les liaisons des chaînes latérales au sein d'une protéine particulière déterminent sa structure tertiaire. La structure tertiaire de la protéine est définie par ses coordonnées atomiques . Ces coordonnées peuvent se référer soit à un domaine protéique, soit à l'ensemble de la structure tertiaire. Un certain nombre de structures tertiaires peuvent se replier en une structure quaternaire .
Histoire
La science de la structure tertiaire des protéines est passée d'une hypothèse à une définition détaillée. Bien qu'Emil Fischer ait suggéré que les protéines étaient constituées de chaînes polypeptidiques et de chaînes latérales d'acides aminés, c'est Dorothy Maud Wrinch qui a incorporé la géométrie dans la prédiction des structures des protéines . Wrinch l'a démontré avec le modèle Cyclol , la première prédiction de la structure d'une protéine globulaire . Méthodes modernes sont capables de déterminer, sans prédiction, des structures tertiaires à moins de 5 Å (0,5 nm) pour les petites protéines (<120 résidus) et, dans des conditions favorables, confiant structure secondaire prédictions.
Déterminants
Stabilité des États natifs
Thermostabilité
Une protéine repliée dans son état natif ou sa conformation native a généralement une énergie libre de Gibbs plus faible (une combinaison d' enthalpie et d' entropie ) que la conformation dépliée. Une protéine va tendre vers des conformations à basse énergie, ce qui va déterminer le repliement de la protéine dans l' environnement cellulaire . Parce que de nombreuses conformations similaires auront des énergies similaires, les structures protéiques sont dynamiques , fluctuant entre un grand nombre de ces structures similaires.
Les protéines globulaires ont un noyau de résidus d'acides aminés hydrophobes et une région de surface de résidus hydrophiles chargés, exposés à l' eau . Cette disposition peut stabiliser les interactions au sein de la structure tertiaire. Par exemple, dans les protéines sécrétées , qui ne baignent pas dans le cytoplasme , les liaisons disulfure entre les résidus cystéine aident à maintenir la structure tertiaire. Il existe une communauté de structures tertiaires stables observées dans des protéines de fonctions diverses et d' évolutions diverses . Par exemple, la barrière de TIM , du nom de l'enzyme triose-phosphate isomérase , est une structure tertiaire commun est hautement stable, dimérique , superhélice structure. Par conséquent, les protéines peuvent être classées selon les structures qu'elles contiennent. Les bases de données de protéines qui utilisent une telle classification comprennent SCOP et CATH .
Pièges cinétiques
La cinétique de repliement peut piéger une protéine dans une conformation de haute énergie , c'est-à-dire qu'une conformation intermédiaire de haute énergie bloque l'accès à la conformation de plus basse énergie. La conformation à haute énergie peut contribuer à la fonction de la protéine. Par exemple, la protéine hémagglutinine de la grippe est une chaîne polypeptidique unique qui, lorsqu'elle est activée, est clivée par protéolyse pour former deux chaînes polypeptidiques. Les deux chaînes sont maintenues dans une conformation à haute énergie. Lorsque le pH local baisse, la protéine subit un réarrangement conformationnel énergétiquement favorable qui lui permet de pénétrer dans la membrane de la cellule hôte .
Métastabilité
Certaines structures protéiques tertiaires peuvent exister dans des états de longue durée qui ne sont pas l'état le plus stable attendu. Par exemple, de nombreuses serpines (inhibiteurs de protéase à sérine) présentent cette métastabilité . Ils subissent un changement de conformation lorsqu'une boucle de la protéine est coupée par une protéase .
Protéines chaperonnes
Il est communément admis que l'état natif d'une protéine est également le plus stable thermodynamiquement et qu'une protéine atteindra son état natif, compte tenu de sa cinétique chimique , avant d'être traduite . Les chaperons de protéines dans le cytoplasme d'une cellule aident un polypeptide nouvellement synthétisé à atteindre son état natif. Certaines protéines chaperons sont très spécifiques dans leur fonction, par exemple, la protéine disulfure isomérase ; d'autres ont une fonction générale et peuvent aider la plupart des protéines globulaires, par exemple, le système de protéines procaryotes GroEL / GroES et les protéines de choc thermique eucaryotes homologues (le système Hsp60/Hsp10).
Environnement cytoplasmique
La prédiction de la structure tertiaire de la protéine repose sur la connaissance de la structure primaire de la protéine et sur la comparaison de la structure tertiaire prédite possible avec les structures tertiaires connues dans les banques de données sur les protéines . Ceci ne prend en compte que l'environnement cytoplasmique présent au moment de la synthèse des protéines dans la mesure où un environnement cytoplasmique similaire peut également avoir influencé la structure des protéines enregistrées dans la banque de données protéiques.
Liaison de ligand
La structure d'une protéine, par exemple une enzyme , peut changer lors de la liaison de ses ligands naturels, par exemple un cofacteur . Dans ce cas, la structure de la protéine liée au ligand est connue sous le nom de structure holo, de la protéine non liée sous le nom de structure apo.
Structure stabilisée par la formation de liaisons faibles entre les chaînes latérales d'acides aminés - Déterminée par le repliement de la chaîne polypeptidique sur elle-même (les résidus non polaires sont situés à l'intérieur de la protéine, tandis que les résidus polaires sont principalement situés à l'extérieur) - L'enveloppement de la protéine rapproche la protéine et concerne des a-to situés dans des régions éloignées de la séquence - L'acquisition de la structure tertiaire conduit à la formation de poches et de sites adaptés à la reconnaissance et à la liaison de molécules spécifiques (biospécificité)
Détermination
La connaissance de la structure tertiaire des protéines globulaires solubles est plus avancée que celle des protéines membranaires car les premières sont plus faciles à étudier avec la technologie disponible.
Cristallographie aux rayons X
La cristallographie aux rayons X est l'outil le plus couramment utilisé pour déterminer la structure des protéines . Il fournit une haute résolution de la structure mais ne donne pas d'informations sur la flexibilité conformationnelle de la protéine .
RMN
La RMN des protéines donne une résolution comparativement plus faible de la structure des protéines. Il est limité à des protéines plus petites. Cependant, il peut fournir des informations sur les changements de conformation d'une protéine en solution.
Microscopie électronique cryogénique
La microscopie électronique cryogénique (cryo-EM) peut fournir des informations sur la structure tertiaire et quaternaire d'une protéine. Il est particulièrement bien adapté aux grosses protéines et aux complexes symétriques de sous-unités protéiques .
Interférométrie à double polarisation
L'interférométrie à double polarisation fournit des informations complémentaires sur les protéines capturées en surface. Il aide à déterminer les changements de structure et de conformation au fil du temps.
Projets
Algorithme de prédiction
Le projet Folding@home de l'Université de Stanford est un effort de recherche en informatique distribuée qui utilise environ 5 pétaFLOPS (≈10 x86 petaFLOPS) de calcul disponible. Il vise à trouver un algorithme qui prédit de manière cohérente les structures tertiaires et quaternaires des protéines en fonction de la séquence d'acides aminés de la protéine et de ses conditions cellulaires.
Une liste de logiciels pour la prédiction de la structure tertiaire des protéines est disponible sur Liste des logiciels de prédiction de la structure des protéines .
Maladies de l'agrégation protéique
Les maladies d' agrégation de protéines telles que la maladie d'Alzheimer et la maladie de Huntington et les maladies à prions telles que l'encéphalopathie spongiforme bovine peuvent être mieux comprises en construisant (et en reconstruisant) des modèles de maladie . Cela se fait en provoquant la maladie chez des animaux de laboratoire, par exemple, en administrant une toxine , telle que le MPTP pour provoquer la maladie de Parkinson, ou par manipulation génétique . La prédiction de la structure des protéines est une nouvelle façon de créer des modèles de maladies, ce qui peut éviter l'utilisation d'animaux.
Projet de récupération de la structure tertiaire des protéines (CoMOGrad)
Faire correspondre les modèles de structure tertiaire d'une protéine donnée à un grand nombre de structures tertiaires de protéines connues et récupérer les plus similaires par ordre de classement est au cœur de nombreux domaines de recherche tels que la prédiction de la fonction de nouvelles protéines, l'étude de l'évolution, le diagnostic de la maladie, la découverte de médicaments, conception d'anticorps, etc. Le projet CoMOGrad de BUET est un effort de recherche visant à mettre au point une méthode extrêmement rapide et très précise pour la récupération de la structure tertiaire des protéines et à développer un outil en ligne basé sur les résultats de la recherche.
Voir également
Les références
Liens externes
- Banque de données sur les protéines
- Afficher, analyser et superposer des structures 3D de protéines
- Alphabet des structures protéiques.
- Afficher, analyser et superposer des structures 3D de protéines
- Cours basé sur le WWW enseignant la bioinformatique élémentaire des protéines
- Évaluation critique de la prédiction de structure (CASP)
- Classification structurelle des protéines (SCOP)
- Classification de la structure des protéines CATH
- Logiciel DALI/FSSP et base de données de structures protéiques superposées
- TOPOFIT-DB Noyaux structuraux invariants entre protéines
- PDBWiki — Page d'accueil PDBWiki – un site Web pour l'annotation communautaire des structures PDB.